ISBN 978-85-85905-10-1
Área
Química Orgânica
Autores
Ferreira Oliveira, D. (UFLA) ; Aparecida Costa Campos, V. (UFLA)
Resumo
A (R)-goniotalamina (GON) foi utilizada em buscas farmacofóricas em banco de dados de ligantes de proteínas, o que permitiu selecionar a enzima aspartato amônia liase (AAL), cuja produção por nematoides era desconhecida. As sequências de aminoácidos de AAL, produzidas por outros organismos, foram utilizadas em buscas nos genomas de Meloidogyne spp., resultando em sequência que foi modelada por threading. A estrutura 3D foi submetida a simulação por dinâmica molecular e essemble docking com inibidores da AAL e com a GON, o que permitiu observar elevada afinidade da GON pela enzima, concordando com os dados experimentais obtidos previamente. Logo, o modelo gerado poderá ser empregado para propor estruturas químicas mais eficientes que a GON para o controle de nematoides
Palavras chaves
Aspartato amônia liase; Meloidogyne; Goniotalamina
Introdução
Um dos grandes problemas enfrentados pela agricultura mundial diz respeito aos nematoides parasitas de plantas, principalmente aqueles do gênero Meloidogyne, que estão distribuídos pelo mundo todo e atacam praticamente todas as plantas cultiváveis, o que acarreta prejuízos mundiais que excedem os 10 bilhões de dólares por ano (WILLIAMSON e KUMAR, 2006). No caso específico do Brasil, as espécies Meloidogyne exigua Goeldi e Meloidogyne incognita (Kofoid and White) Chitwood são responsáveis por elevadas perdas para culturas de grande importância econômica e social como café e soja (RANDIG et al., 2004; DIAS et al., 2010). Para controlar os referidos nematoides, geralmente se faz uso de nematicidas comerciais que, além de aumentarem os custos de produção, podem contaminar o ambiente e o homem com substâncias de elevada toxicidade. Consequentemente, em trabalho previamente realizado (resultados não publicados) com vistas a identificar substâncias com propriedades nematicidas, observou-se que a (R)-goniotalamina (GON) era ativa in vitro e in vivo contra Meloidogyne spp. Para dar continuidade a este trabalho, objetivou-se identificar através de métodos in silico, o alvo enzimático desta substância nos nematoides, para gerar um modelo que pudesse servir para o desenvolvimento de produtos análogos da GON, com maior eficiência nematicida.
Material e métodos
A estrutura da GON foi submetida à busca farmacofórica por ligantes de enzimas através do programa ReverseScreen3D (KINNINGS e JACKSON, 2011). Com base no valor de 3Dscore, selecionou-se a enzima AAL, cujas sequências de aminoácidos foram obtidas do RCSB Protein Data Bank (http://www.pdb.org) e utilizadas na busca por sequências de aminoácidos similares nos bancos de dados do INRA- Meloidogyne incognita (http://www6.inra.fr/meloidogyne_incognita) e do Nematode.net v 3.0 (http://nematode.net/NN3_frontpage.cgi). A sequência com maior similaridade, oriunda de Meloidogyne hapla Chitwood, foi modelada com o programa I-TASSER (ZHANG, 2008). As estruturas 3D geradas foram empregadas na obtenção de homotetrâmeros com base nas enzimas AAL 3E04 e 1YFM (http://www.pdb.org). Após otimização dos tetrâmeros com os programas computacionais SCWRL4 (KRIVOV et al., 2009) e NAMD 2.9 (PHILLIPS et al., 2005), as estruturas finais foram avaliadas com os programas FOLDX 2.5.2 (SCHYMKOWITZ et al., 2005) e GOAP (ZHOU e SKOLNICK, 2011). A melhor estrutura (ita2-1YFM), segundo ambos os programas, foi submetida à simulação por dinâmica molecular com o programa NAMD 2.9, representando-se o solvente explicitamente, por 4 ns, sob condições periódicas de fronteira, considerando-se o ensemble isobárico- isotérmico (NPT). As conformações da enzima, presentes no último 1 ns do processo de simulação, foram submetidas a ancoramentos as cegas com o programa computacional Autodock Vina 1.1.2 (TROTT e OLSON, 2010). Tal ancoramento foi realizado com as seguintes substâncias: GON, citrato, fumarato, (S)-malato, oxalatoacetato e meso-tartarato.
Resultado e discussão
A busca farmacofórica realizada com a estrutura química da GON revelou que a
enzima AAL deveria ser o seu alvo enzimático mais provável, já que se observou
elevado valor de 3Dscore (KINNINGS & JACKSON, 2011) para o ligante desta enzima.
Como não havia qualquer relato sobre a produção da AAL por nematoides,
utilizaram-se as sequências de aminoácidos de tal enzima, produzida por outros
organismos, para a procura por sequências similares nos genomas de
Meloidogyne spp. O melhor resultado foi obtido para uma sequência oriunda
do genoma de M. hapla, com similaridades entre 46 e 77% em relação às AAL
conhecidas. Na modelagem da sequência selecionada se obtiveram nove estruturas
3D. Logo, foi necessário otimizá-las e ranqueá-las para que a melhor, segundo
ambos os programas de ranqueamento, fosse escolhida. A seguir, com vistas a
permitir o relaxamento da estrutura e gerar várias conformações, foi feita a
simulação por dinâmica molecular até que o valor de root mean square
deviation (RMSD) estabilizasse. O processo de ancoramento as cegas permitiu
observar que a GON apresentava maior afinidade pela AAL que todas as outras
estruturas (Figura 1), que se tratam de inibidores comuns das enzimas AAL
produzidas por outros organismos (BEECKMANS e DRIESSCHE, 1998; GREENHUT et al.,
1985). Este resultado sugere que a GON apresente elevado potencial para uso no
desenvolvimento de potentes inibidores da AAL de Meloidogyne spp. e,
consequentemente, de novos produtos para o controle do referido nematoide.
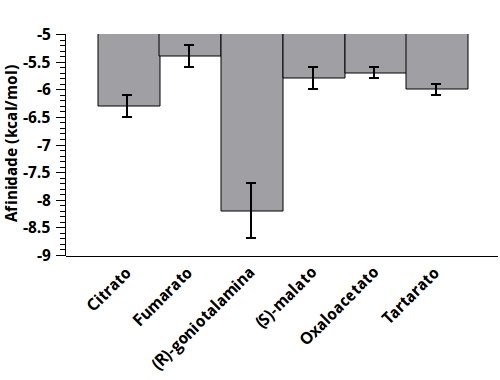
Afinidades das substâncias pela enzima aspartato amônia liase de acordo com os cálculos realizados com o programa Autodock Vina 1.1.2.
Conclusões
O estudo in silico realizado permitiu gerar um modelo compatível com os dados experimentais previamente obtidos, o que sugere que o mecanismo de ação da GON contra Meloidogyne spp. consista na inibição da AAL produzida pelos nematoides. Com isto, foi construído um modelo que poderá ser de grande importância para gerar estruturas mais eficientes que a GON para o controle de fitonematoides.
Agradecimentos
À CAPES pela concessão de bolsa de estudo, ao CNPq e à FAPEMIG pela concessão de recursos, e ao CENAPAD-SP no qual parte da pesquisa foi desenvolvida.
Referências
BEECKMANS, S. AND DRIESSCHE, V. E.; Pig heart fumarase contains two distinct substrate-binding sites differing in affinity. Journal of Biological Chemistry v. 273, p. 31661-31669.
DIAS, W.P.; GARCIA, A.; SILVA, J.F.V; CARNEIRO, G.E.S. Nematoides em soja: Identificação e controle. Circular Técnica: EMBRAPA, Londrina, 2010.
GREENHUT, J.; UMEZAWA, H.; RUDOLPH, F. B. Inhibition of fumarase by S-2,3 dicarboxyaziridine. Journal of Biological Chemistry v. 260, p. 6684-6686, 1985.
KINNINGS SL AND JACKSON RM. ReverseScreen3D: A structure-based ligand matching method to identify protein targets. Journal of Chemical Information and Modeling v. 28, p. 624-634, 2011.
KRIVOV, G. G.; SHAPOVALOV, M. V.; DUNBRACK, R. L. Jr. Improved prediction KUBINYI, H. New Approaches in Drug Design p. 16-11, 2005.
PHILLIPS, C.J.; BRAUN, R.; WANG, W.; GUMBART, J.; TAJKHORSHID, E.; VILLA, E.; CHIPOT, C.; SKELL, R.D.; KALE, L.; SXHULTEN, K. Scalable molecular dynamics with NAMD. Journal of Computational Chemistry v. 26, p. 1781-1802, 2005.
RANDIG, O.; CARNEIRO, R. M. D. G.; CASTAGNONE-SERENO, P. Identificação das principais espécies de Meloidogyne parasitas do cafeeiro no Brasil com marcadores SCAR-café em Multiplex-PCR. Nematologia Brasileira, v. 28, n.1, p. 1-10, 2004.
SCHYMKOWITZ, J.; BORG, J.; STRICHER, F.; NYS, R.; ROUSSEAU, F.; SERRANO, L. The FoldX web server: an online force field. Nucleic Acids Res, v. 33, p.382-388, 2005.
TROTT, O.; OLSON, A. J. AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization and multithreading. Journal of Computational Chemistry, v. 31, p. 455-461, 2010.
WILLIAMSON, V. M. AND KUMAR, A. Nematode resistance in plants: the battle underground. Trends in Genetics, v. 22, p. 396-403, 2006.
ZHANG, Y. I-TASSER server for protein 3D structure prediction. Bioinformatics, v.9, p. 40, 2008.
ZHOU, H.; SKOLNICK, J. GOAP: A generalized orientation-dependent, all-atom statistical potential for protein structure prediction. Biophysical Journal, v. 101, n. 8, p. 2043-2052, 2011.










