ISBN 978-85-85905-15-6
Área
Química Analítica
Autores
Gimenez, T.D. (UFG) ; Fiaccadori, F.S. (UFG) ; Rosa, H.C. (UFG) ; Bailão, A.M. (UFG) ; Duarte, G.R.M. (UFG)
Resumo
A obtenção de RNA de alta qualidade é crucial para o sucesso das etapas subsequentes de uma análise genética. Nesse sentido, este trabalho tem como objetivo o desenvolvimento de uma metodologia de extração dinâmica em fase sólida (dSPE) para purificação de RNA a partir de amostras de soro de pacientes infectados com o vírus DENV. A dSPE envolve o uso de partículas magnéticas de sílica como fase sólida e se desenvolve através da realização de três etapas: adsorção de RNA nas partículas; lavagem das partículas; e dessorção e eluição do RNA puro. Inicialmente os experimentos foram realizados em microtubos e posteriormente em microchips de PeT. RNA de alta qualidade (amplificável via RT-PCR) foi obtido, demonstrando o sucesso da dSPE para purificação de RNA a partir de uma amostra complexa.
Palavras chaves
dSPE; RNA; Microchips de PeT
Introdução
Uma etapa comum em todas as técnicas moleculares para a detecção do vírus DENV é a extração do RNA viral. Devido ao RNA viral estar presente em pequenas quantidades no plasma de pacientes infectados, a qualidade da extração do RNA é uma etapa crucial para as reações subsequentes (DETTOGNI; LOURO, 2011). Os métodos de extração de ácidos nucleicos em fase sólida realizados em microchips utilizam convencionalmente partículas de sílica empacotadas dentro dos canais microfluídicos, e as amostras e reagentes são levados aos canais através da utilização de bombas. Recentemente, outras alternativas para o uso de sílica como fase sólida foram desenvolvidas, como a utilização de partículas magnéticas de sílica. O uso de partículas magnéticas, no lugar de partículas empacotadas no canal, proporciona maior interação da fase sólida com as amostras e dispensa o uso de bombas, devido ao fato de as partículas magnéticas terem a capacidade de ficarem livres, podendo se movimentar através da aplicação de um campo magnético (REINHOLT; BAEUMNER, 2014). O objetivo deste trabalho é o desenvolvimento de uma metodologia de extração dinâmica em fase sólida (dSPE) para purificação de RNA viral a partir de amostras complexas como o soro. A dSPE utilizada neste trabalho envolve a utilização de partículas magnéticas de óxido de ferro revestidas com dióxido de silício, com a realização de três etapas: (1) adsorção das moléculas de RNA nas partículas magnéticas de sílica em um meio contendo um sal de guanidina; (2) lavagem das partículas com álcool; e (3) dessorção e eluição das moléculas de RNA em água. A extração de RNA em microchips de poliéster-toner (PeT) é a primeira etapa para o desenvolvimento de um microdispositivo de PeT completamente integrado para o diagnóstico molecular da dengue.
Material e métodos
Primeiramente, foram feitas extrações de RNA a partir de amostra de soro em microtubos de polipropileno. Nesta etapa, 12 µL de partículas magnéticas em hidrocloreto de guanidina (GuHCl) 6 mol/L e pH 6,1 foram adicionados a um microtubo, juntamente com 12 µL de amostra lisada de soro. As partículas foram agitadas por 5 minutos, o sobrenadante removido, e as partículas foram lavadas com 2 frações de 30 µL de etanol 80%. O álcool foi removido e o microtubo aberto foi aquecido a 56 °C por 10 minutos. Após a adição de 18 µL de água, as partículas foram novamente agitadas por 5 minutos e o sobrenadante contendo RNA foi eluído e coletado em outro microtubo. Na sequência, foram feitas extrações de RNA em microchips de PeT. Para a produção dos microdispositivos foi utilizada a metodologia proposta por Duarte e colaboradores (DUARTE et al., 2011). Para as extrações, os canais microfluídicos foram preenchidos com GuHCl 6 mol/L pH 6,1. Foram adicionados 2 µL de partículas magnéticas de sílica em GuHCl e 3 µL de amostra lisada de soro, com posterior agitação das partículas magnéticas por 5 minutos. Foram feitas lavagens com 4 frações de 2 µL de etanol 80%, agitação das partículas por 1 minuto e lavagens com mais 4 frações de 2 µL de etanol 80%. Em seguida, foram feitas lavagens com 8 frações de 2 µL de água e as partículas magnéticas foram agitadas por 5 minutos. As moléculas de RNA foram eluídas em 8 frações, cada uma contendo um volume de 2 µL. A pureza do RNA extraído foi avaliada por espectrofotometria de absorção molecular UV-Vís (NanoDrop®) e a qualidade do RNA extraído foi avaliada através da amplificação de um fragmento por meio da transcrição reversa seguida da reação em cadeia da polimerase (RT-PCR). As frações eluídas também foram quantificadas pelo NanoDrop®.
Resultado e discussão
A Figura 1 mostra o produto de amplificação de RNA extraído a partir de soro
através da dSPE. O sucesso da RT-PCR mostra que a metodologia utilizada foi
capaz de extrair RNA de alta qualidade a partir de amostras complexas, como
a amostra de soro. RNA é adsorvido na superfície das partículas magnéticas
de sílica em um meio contendo GuHCl 6 mol/L pH 6,1 e dessorvido em água
ultrapura. As lavagens com etanol 80% são suficientes para a remoção de
substâncias indesejadas que possam ser adsorvidas nas partículas de sílica.
Além disso, a metodologia apresentou vantagens importantes, como tempo
reduzido e uso de reagentes pouco tóxicos, quando comparada a muitos
protocolos de extração de RNA.
Após a verificação da viabilidade da dSPE para a purificação de RNA a partir
de soro em microtubos, foram feitos experimentos de dSPE em microchips de
PeT. A Figura 2 mostra o perfil de eluição de RNA extraído a partir de soro
em microchips de PeT utilizando a dSPE. As duas primeiras frações exibidas
na Figura 2 correspondem às duas últimas frações das lavagens com água,
antes de se promover a agitação das partículas magnéticas para a dessorção
do RNA. A primeira fração coletada contendo RNA eluído após agitação das
partículas magnéticas é a terceira fração. O perfil de eluição mostra que,
nas extrações em microchips, a primeira fração eluída é a que apresenta
maior quantidade de RNA purificado. Os resultados demonstraram que tanto o
toner quanto o poliéster são compatíveis com a extração de RNA.
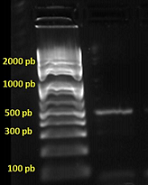
Produto de amplificação de RNA extraído em microtubo de polipropileno.
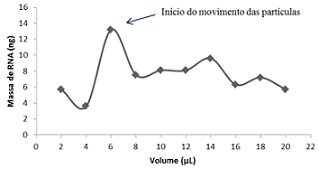
Perfil de eluição de RNA extraído em microchips de PeT.
Conclusões
A dSPE mostrou ser um método simples e rápido para purificação de RNA de alta qualidade, ou seja, RNA que pode ser amplificado por meio da RT-PCR, a partir de amostras complexas. A dSPE apresentou também como vantagens o uso de reagentes pouco tóxicos e a possibilidade de adaptação para utilização em plataformas miniaturizadas. O desenvolvimento de uma metodologia de extração de RNA do vírus DENV a partir de amostras de soro de pacientes infectados será a primeira etapa em direção ao desenvolvimento de um diagnóstico molecular da dengue em microchip de poliéster-toner integrado.
Agradecimentos
Os autores agradecem ao CNPq, à FAPEG, e aos Institutos de Química, Ciências Biológicas e Patologia Tropical e Saúde Pública da Universidade Federal de Goiás.
Referências
DETTOGNI, R. S.; LOURO, I. D. Dengue virus RNA purification from human plasma: a comparison of two techniques. Molecular Biology Reports, 38, p. 4979-4983, 2011.
DUARTE, G. R. M.; PRICE, C. W.; AUGUSTINE, B. H.; CARRILHO, E.; LANDERS, J. P. Dynamic solid phase DNA extraction and PCR amplification in polyester-toner based microchip. Analytical Chemistry, vol. 83, n. 13, p. 5182-5189, 2011.
REINHOLT, S. J.; BAEUMNER, A. J. Microfluidic isolation of nucleic acids. Angewandte Chemie, 53, p. 13988-14001, 2014.