ISBN 978-85-85905-15-6
Área
Produtos Naturais
Autores
Bandeira, F.S. (EMBRAPA AGROENERGIA) ; Souto, A.L. (EMBRAPA AGROENERGIA) ; Ribeiro, J.A.A. (EMBRAPA AGROENERGIA) ; Abdelnur, P.V. (EMBRAPA AGROENERGIA) ; Rodrigues, C.M. (EMBRAPA AGROENERGIA)
Resumo
A metabolômica pode ser definida como um estudo analítico de todos os tipos de moléculas presentes em um determinado sistema biológico, o metaboloma. No entanto, devido a grande diversidade de metabólitos secundários contidos nas plantas, a escolha da estratégia de preparo da amostra é crucial para o sucesso de um experimento. O objetivo deste trabalho foi desenvolver um protocolo eficiente e reprodutível acerca do preparo de amostra de folhas de Elaeis guineensis para a detecção do maior número de metabólitos polares presentes nesta matriz. Extratos dos folíolos basais, centrais e apicais produzidos por metodologias diferentes foram analisados por UPLC-PDA. A metodologia mais indicada para o estudo metabolômico foi a extração aquosa por infusão dos folíolos apicais da espécie em estudo.
Palavras chaves
Fingerprinting MS; UPLC; Dendê
Introdução
Nos últimos anos, a metabolômica tem sido aplicada em diversas áreas, e particularmente na ciência de plantas, provendo informações de grande importância (FREDERICH et al., 2009). A metabolômica pode ser definida como um estudo analítico de todos os tipos de moléculas presentes em um determinado sistema biológico, o metaboloma (VUCKOVIC, 2012). A análise da composição metabólica de várias amostras pode auxiliar na identificação de padrões ou marcadores biológicos que são específicos de uma determinada espécie, cultivar ou de um determinando estágio de desenvolvimento da planta. Pode-se identificar também, padrões característicos de plantas doentes, estressadas ou ainda padrões metabólicos de plantas em determinadas estações do ano (KIM et al., 2011). Entretanto, um dos maiores entraves da metabolômica está em detectar, através da análise de uma única amostra, todos os metabólitos contidos em um organismo, o que ainda é virtualmente impossível, pois o metaboloma consiste de uma grande variedade de compostos, em diferentes concentrações e com diferentes polaridades (SCHRIPSEMA, 2009). Atualmente, devido a grande diversidade de metabólitos secundários contidos nas plantas, ainda não há uma metodologia padrão de preparo de amostra, e desta maneira, muitos laboratórios utilizam métodos desenvolvidos por eles mesmos, onde cada protocolo é desenvolvido para cada caso específico (FUKUSAKI E KOBAYASHI, 2005; SCHRIPSEMA, 2009). Desta maneira, pode-se dizer que a escolha da estratégia durante o preparo da amostra é crucial para o sucesso de um dado experimento (MOCO et al., 2007). Baseando-se nesta fundamentação, o objetivo deste trabalho foi desenvolver um protocolo eficiente e reprodutível acerca do preparo de amostra de folhas de Elaeis guineensis, popularmente conhecida como ”Dendê”, para a detecção do maior número de metabólitos polares presentes nesta matriz.
Material e métodos
Uma folha de Dendê (Elaeis guineensis) saudável e sem nenhuma deformidade ou doença aparente, foi coletada na Embrapa Agroenergia (Brasília, DF). Os folíolos foram separados com auxílio de tesoura em 3 partes equivalentes: folíolos basais (mais velhos), folíolos centrais e folíolos apicais (mais jovens). Posteriormente, o material botânico foi lavado separadamente com solução de hipoclorito de sódio a 1% por 2 minutos, seguido de lavagem com água destilada e secagem em estufa a 40 ºC até obter-se peso constante. Após a etapa de secagem, os folíolos foram triturados em moinho analítico de bancada, seguido de uma segunda trituração em moinho ultracentrífugo à 11.000 g. O material seco e pulverizado foi então tamisado por 10 minutos com tamis de malha número 100, a fim de padronizar o tamanho das partículas e obter extrações mais homogêneas e reprodutíveis. Entre as triturações e tamisações das diferentes partes da folha (base, meio e ápice) foi feita uma limpeza dos equipamentos com ar comprimido a fim de não ocorrer contaminação cruzada. O material tamisado das partes basal, central e apical foi submetido à etapa de extração (feita em triplicata) em erlenmeyer com água ultrapura a 80 ºC (proporção de 1 g para 25 mL) seguido de sonicação por 10 minutos. A mistura foi então transferida para tubos falcon e centrifugada a 9.000 g por 10 minutos; e o sobrenadante foi então filtrado em papel para um balão volumétrico de 25 mL e o volume foi completado com o solvente de extração. O mesmo procedimento foi realizado para a extração com etanol a 70%. Alíquotas da solução extrativa foram filtradas em filtros de 0,45 µm para vials antes de serem analisadas por UHPLC (Ultra-High Performance Liquid Chromatography). Entretanto, a fim de retirar a clorofila contida nas amostras extraídas com etanol a 70%, estas foram também submetidas a uma etapa de SPE (Solid Phase Extraction) com cartuchos de 500 mg de C18 antes de serem analisadas. As amostras de cada parte da folha, extraídas com ambos os métodos, foram preparadas em triplicata e analisadas em UHPLC-PDA, (modelo Acquity H-Class, Waters) utilizando coluna Acquity BEH C18 (1,7 µm 2,1 x 150 mm), vazão de 0,5 mL/min, faixa de detecção de 200 a 400 nm e um método de eluição com gradiente exploratório utilizando o sistema constituído de acetonitrila e água com ácido trifluoracético a 0,01% (Tabela 1).
Resultado e discussão
A fim de analisar o perfil metabólico de Elaeis guineenses, primeiramente foi
preciso verificar qual o melhor método de extração a ser utilizado,
possibilitando a identificação do maior número de compostos. Adicionalmente, o
método deve ser reprodutível, e para que isso ocorra, diversas variáveis devem
ser controladas, como: horário da coleta, tipo e tempo de secagem, extração,
natureza do solvente extrator, tamanho da partícula e razão solvente/amostra.
Neste experimento, todas as variáveis citadas foram controladas. Diante disso,
decidiu-se verificar se havia alguma diferença entre os perfis metabolômicos
dos extratos etanólico e aquoso, e qual seria o melhor candidato para um
estudo metabolômico posterior. Ao analisar os perfis cromatográficos de ambos
os extratos (Figura 1), foi possível verificar que a parte apical apresentou
compostos mais concentrados em relação a outras partes da folha, demonstrando
assim que o fator envelhecimento refletia no perfil metabolômico da espécie.
Ao comparar os perfis de folíolos apicais do extrato etanólico com o do
extrato aquoso, percebeu-se que o perfil do extrato etanólico apresentava
compostos com maior intensidade de absorção. A partir da análise qualitativa,
não se observou diferenças nítidas entre os perfis detectados pelos dois tipos
de extração ou metodologia, além disso, verificou-se uma presença maior de
compostos polares e semi-polares em ambos os extratos, pois praticamente todos
os compostos se encontravam eluídos na metade da corrida.
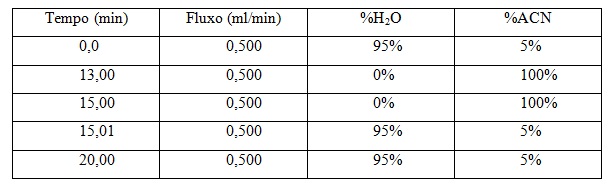
Gradiente de eluição utilizado na análise dos extratos aquoso e etanólico das folhas de Elaeis guineensis
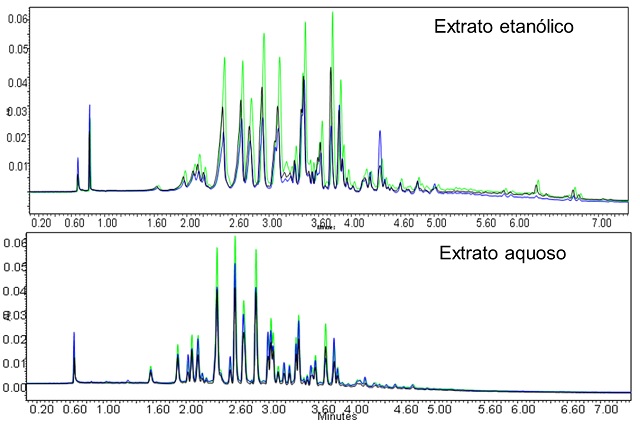
Cromatogramas dos extratos etanólico e aquoso das folhas de Elaeis guineensis. Verde: parte apical, azul:parte mediana, preto:parte basal
Conclusões
Após realizar coleta, secagem e extração dos folíolos basais, centrais e apicais de Elaeis guineensis e comparar os dados obtidos por UHPLC-PDA foi possível verificar um perfil metabólico semelhante para os extratos etanólico e aquoso, quando comparados qualitativamente. No entanto, o extrato etanólico dos folíolos apicais apresentou alguns compostos em maior concentração, sendo, portanto, o mais indicado para a realização de um estudo metabolômico detalhado da espécie.
Agradecimentos
Agradecemos a Capes e a Embrapa pelo financiamento a pesquisa e pela estrutura laboratorial fornecida
Referências
FREDERICH, M.; JANSEN, C.; DE TULLIO, P.; TITS, M.; DEMOULIN, V.; ANGENOT, L. Metabolomic analysis of Echinacea spp. by 1H nuclear magnetic resonance spectrometry and multivariate data analysis technique. Phytochemical Analysis, nº 21, 61-65, 2009.
FUKUSAKI, E.; KOBAYASHI, A. Plant metabolomics: potential for practical operation. Journal of Bioscience and Bioengineering, nº 100, 347-354, 2005
KIM, H.K.; CHOI, Y. H.; VERPOORTE, R. NMR-based plant metabolomics: where do we stand, where do we go? Trends in Biotechnology, nº 29, 267-275, 2011.
MOCO, S.; VERVOORT, J.; BINO, R.J.; DE VOS, R.C.H.; BINO, R. Metabolomics technologies and metabolite identification. Trends in Analytical Chemistry, nº 26, 855-866, 2007
SCHRIPSEMA, J. Application of NMR in plant metabolomics: techniques, problems and prospects. Phytochemical Analysis, nº 21, 14-21, 2009.
VUCKOVIC, D. Current trends and challenges in sample preparation for global metabolomics using liquid chromatography-mass spectrometry. Analytical and Bioanalytical Chemistry, nº 403, 1523-1548, 2012.