ISBN 978-85-85905-19-4
Área
Físico-Química
Autores
Galúcio, J.M. (UFOPA) ; da Costa, K.S. (UFOPA) ; Lima, A.H. (UFPA) ; Leal, E. (UFPA) ; Lameira, J. (UFPA)
Resumo
O Fator de Infecção Viral é uma proteína do HIV-1, essencial para a infectividade e replicação viral. Sua principal função é formar um complexo com as proteínas Cul5, EloB e EloC do complexo celular Ub-E3-ligase e então induzir a ubiquitinação e degradação do fator antiretroviral humano A3G. A Vif é um alvo potencial para intervenção terapêutica na AIDS e a recente elucidação de sua estrutura abriu novas possibilidades para o desenho de drogas. Neste trabalho, a estrutura do complexo com a proteína A3G foi montada. Através de docagem molecular, análogos do RN18 foram desenhados de modo a aumentar a seletividade e afinidade à Vif. Os resultados demonstram que as melhores poses obtidas para o RN18 e seus análogos encontram-se em uma cavidade próxima ao motivo 161PPLP164.
Palavras chaves
Desenho de Análogos; Docagem Molecular; RN18
Introdução
Desde o início da epidemia da síndrome da imunodeficiência adquirida (AIDS), esta doença levou à morte de mais de 35 milhões de pessoas no mundo. Embora o crescimento global da epidemia pareça estar diminuindo, com 42% de redução de mortes por AIDS desde 2004, os números ainda são muito elevados, com cerca de 36,9 milhões de infectados no mundo e dois milhões de novas infecções por ano. (UNAIDS, 2015). Ao longo das últimas duas décadas, mais de 25 medicamentos contra o vírus da imunodeficiência humana (HIV), o retrovírus que causa a AIDS, têm sido desenvolvidos visando várias fases diferentes da vida do ciclo de vida do vírus (ANDRADE et al., 2011). No entanto, o desenvolvimento de resistência às drogas e efeitos colaterais associados à HAART têm criado a necessidade de terapias mais potentes e menos tóxicas contra outros alvos virais e das interações vírus-hospedeiro. Portanto, é urgente o desenvolvimento de novos inibidores com base em novos mecanismos de ação. (ARTS; HAZUDA, 2012). O Fator de infecção viral (Vif) é uma proteína multifuncional e multidomínio expressa no Vírus da Imunodeficiência Humana (HIV) e em vários outros lentivírus. A Vif tem como alvo principal a A3G (SHEEHY et al., 2002), que, juntamente com outras proteínas, inibe a replicação de retrovírus e retrotransponsons (HE et al., 2008; XIAO et al., 2007; ZHOU et al., 2012) A degradação de APOBEC3G/APOBEC3F ocorre pela apropriação, por parte da Vif, da via celular ubiquitina-proteossomo, mais especificamente pelo sequestro do complexo E3 ubiquitina ligase, composto pelas proteínas celulares Elonguina B e C (EloBC), Culina-5 (Cul5), Nedd8 e RING-box 2 (Rbx2). Este complexo leva à poliubiquitinação das proteínas APOBECs, e consequentemente, à degradação mediada pelo proteossomo 26S.
Material e métodos
Docagem molecular e Planejamento de Moléculas Análogas do RN18 A RN18 foi docada na estrutura da Vif complexada às proteínas EloB, EloC, A3G, Cul5 e CBF-beta utilizando o algoritmo de pesquisa MolDock Optimizer disponível no programa MVD (DE AZEVEDO, 2010; THOMSEN; CHRISTENSEN, 2006). O protocolo flexível foi utilizado isto é, as cadeias laterais e os ligantes serão assumidos com estrutura flexível e a caixa de docagem foi posicionada na região mais provável de ligação. Foram utilizados os seguintes parâmetros para o algoritmo de evolução diferencial: tamanho da população = 100, taxa de crossing = 0,9, taxa de mutação = 0,5, e as iterações máximas = 2000. Foi realizado um total de 10 corridas e, depois da docagem, o programa executou uma minimização de energia nas cadeias laterais dos resíduos. As posições obtidas na docagem foram selecionadas pela pontuação de energia do Moldock e foram analisadas em diagramas 2D obtidas a partir das coordenadas 3D geradas no programa MVD usando-se o programa Biovia Discovery Studio. (Accelrys, 2012). Baseado nos resultados de docagem do RN18, modificações estruturais foram feitas de modo a aperfeiçoar a afinidade e especificidade de ligação na Vif. Moléculas análogas foram desenhadas no programa MarvinSketch (CHENAXON LTDA, 2014). Adicionalmente, foram utilizados neste estudo três análogos do RN18 desenhados e sintetizados em trabalho de Ali et al. (2012), aqui identificados como RN18-A, RN18-B e RN18-C. Antes da docagem, filtrou-se a biblioteca de moléculas desenhadas quanto às suas propriedades físico-químicas, utilizando o programa Free ADME-Tox Filtering Tool- FAF Drugs 3 e em seguida, as moléculas foram analisadas pelo programa OSIRIS Data Warrior (SANDER et al., 2009).
Resultado e discussão
Das três simulações de docagem locais, observou-se que a cavidade C, localizada perto do motivo rico em prolina (161PPLP164), mostra ser o mais favorável sítio de ligação do RN18 e exibiu uma pontuação MolDock de -147,260, a partir das três simulações realizadas. Da mesma forma, a docagem global também obteve a melhor pose na cavidade C (MolDock -141,506). Nessa cavidade, o RN18 interage com o motivo 161PPLP164 através de cinco interações principais: três interações hidrofóbicas do tipo pi-alquil envolvendo os resíduos P162 e L163 e duas ligações de hidrogênio envolvendo o resíduo L163. Interações adicionais incluem interações pi-alquil com L24, A137 e K168, ligação de hidrogênio com W21, entre outras. As oito moléculas derivadas analisadas e os análogos RN18-A, RN18-B e RN18-C, além do próprio RN18, são demonstrados em 2D na Figura 1. Todos, à exceção da molécula RN18-B, obtiveram pontuação Moldock de maior afinidade à cavidade C, destacando-se a molécula A4, de pontuação -176,8. Quanto aos demais parâmetros analisados pelo algoritmo Moldock Optimizer, a molécula A6 obteve a melhor energia de ligações de hidrogênio (-7,64), enquanto a molécula A10 obteve o melhor Rerank Score entre as moléculas analisadas (-117,55). Os análogos de RN18 obtiveram as melhores poses na cavidade C, com -146,3. -155 e -162,5 de pontuação Moldock, respectivamente. Na docagem molecular, foram comuns várias ligações, como duas ligações de hidrogênio com o resíduo S165 e interações hidrofóbicas pi-alquil com P162 e L163. Os parâmetros físico-químicos e toxicológicos do RN18 e dos seus análogos podem ser vistos na Tabela 1 e Tabela 2.
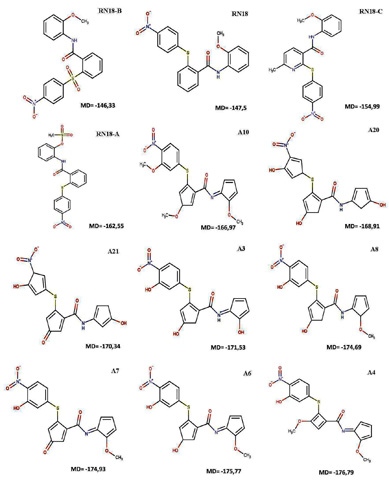
RN18 e análogos analisados neste estudo, com respectivo Moldock Score obtido nas simulações de docagem molecular na cavidade C da Vif.
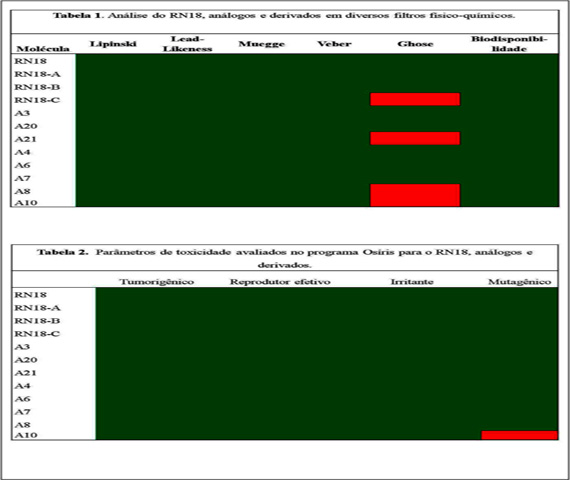
Tabela 1. Análise do RN18, análogos e derivados em diversos filtros físico-químicos Tabela 2. Parâmetros de toxicidade avaliados no programa Osiris
Conclusões
Neste estudo percebemos que o RN18 e todas as demais moléculas analisadas obtiveram as melhores poses na Cavidade C da Vif, localizada próxima ao motivo PPLP, conservando interações hidrofóbicas do tipo pialquil. A partir da comparação da docagem da molécula RN18 na cavidade C e baseado na similaridade estrutural de seus análogos, evidenciada por meio do coeficiente de Tanimoto, sugere-se que modo de ligação destes compostos possa ser semelhante. Além disso, encontramos pouca ou nenhuma violação nos parâmetros Druglikeness, físico químicos e de toxicidade para o RN18 e as moléculas desenhadas.
Agradecimentos
Referências
ANDRADE, C. H.; DE FREITAS, L. M.; DE OLIVEIRA, V. Twenty-six years of HIV science: An overview of anti-HIV drugs metabolismBrazilian Journal of Pharmaceutical Sciences, 2011.
ARTS, E. J.; HAZUDA, D. J. HIV-1 antiretroviral drug therapy. Cold Spring Harbor Perspectives in Medicine, v. 2, n. 4, 2012.
CHENAXON LTDA. MarvinSketchBudapest, 2014.
DE AZEVEDO, W. F. MolDock applied to structure-based virtual screening. Current drug targets, v. 11, n. 3, p. 327–34, mar. 2010.
GHOSE, A. K.; VISWANADHAN, V. N.; WENDOLOSKI, J. J. A Knowledge-Based Approach in Designing Combinatorial or Medicinal Chemistry Libraries for Drug Discovery. 1. A Qualitative and Quantitative Characterization of Known Drug Databases. Journal of Combinatorial Chemistry, v. 1, n. 1, p. 55–68, 1999.
HE, Z. et al. Characterization of conserved motifs in HIV-1 Vif required for APOBEC3G and APOBEC3F interaction. Journal of molecular biology, v. 381, n. 4, p. 1000–11, 12 set. 2008.
SAN DIEGO: ACCELRYS SOFTWARE INC. Discovery Studio Modeling Environment, Release 3.5.
SANDER, T. et al. OSIRIS, an entirely in-house developed drug discovery informatics system. Journal of Chemical Information and Modeling, v. 49, n. 2, p. 232–246, 2009.
SHEEHY, A. M. et al. Isolation of a human gene that inhibits HIV-1 infection and is suppressed by the viral Vif protein. Nature, v. 418, n. 6898, p. 646–650, 2002.
THOMSEN, R.; CHRISTENSEN, M. H. MolDock: a new technique for high-accuracy molecular docking. Journal of medicinal chemistry, v. 49, n. 11, p. 3315–21, jun. 2006.
UNAIDS. AIDS by the numbers. Jc2571/1/E, p. 1–11, 2015.
XIAO, Z. et al. Characterization of a novel Cullin5 binding domain in HIV-1 Vif. Journal of molecular biology, v. 373, n. 3, p. 541–50, 26 out. 2007.
ZHOU, X. et al. Characterization of the interaction of full-length HIV-1 Vif protein with its key regulator CBFβ and CRL5 E3 ubiquitin ligase components. PloS one, v. 7, n. 3, p. e33495, jan. 2012.











