ISBN 978-85-85905-19-4
Área
Físico-Química
Autores
Moura de Sousa, B.S. (UFPA) ; Pereira, I.L.G. (UFPA) ; Reis, A.F.V.F. (UFPA) ; Neto, A.F.G. (UFPA) ; Neto, A.M.J.C. (UFPA)
Resumo
O cocaetileno, produto obtido a partir da transesterificação da cocaína pelo etanol, apresenta um potencial tóxico muito maior do que a própria cocaína, além de permanecer por mais tempo no corpo humano. Estudos comprovam essa alta toxicidade do cocaetileno, principalmente o seu grande maleficio ao sistema cardiovascular. Tendo conhecimento dessas informações o presente trabalho tem como objetivo o estudo computacional do efeito genotoxico do cocaetileno, usando o software AmberTools15. Por meio da simulação de dinâmica molecular, foram extraídos dados como ΔG° e RMSD, com os quais é possível analisar a espontaneidade e estabilidade da ancoragem do sistema cocaetileno/DNA. Tais resultados indicaram um possível potencial genotóxico do cocaetileno.
Palavras chaves
Cocaetileno ; Potencial genotóxico; Dinâmica molecular
Introdução
Obtida de arbustos da espécie Erythroxylum coca, a cocaína é um alcaloide que atua no sistema nervoso central (SNC) e apresenta grande potencial anestésico. Considerada uma das drogas mais utilizadas para fins recreativos, segundo dados da UNODOC(WORLD DRUG REPORT, 2015), no Brasil o consumo de cocaína é 4 vezes superior ao da média mundial. O consumo desenfreado desse e outros tipos de drogas é um problema de saúde pública, e buscar entender os malefícios proporcionados pela mesma ao organismo é essencial. Apontado como um dos metabólitos mais tóxicos da cocaína, o cocaetileno (CE) é produzido somente quando o consumo da mesma ocorre concomitante ao de substancias alcoólicas, por meio de uma reação de transesterificação. Pesquisas cientificas já comprovaram que o cocaetileno apresenta toxicidade maior do que a própria cocaína, essa substância é tida como a principal responsável por problemas no sistema cardiovascular(WILSON et al,2011) que acometem usuários de cocaína e derivados. Baseando-se nestas pesquisas o presente trabalho tem por finalidade apresentar o estudo computacional do potencial de interação do CE com o DNA, e assim constatar a sua genotoxicidade. Para realizar tal trabalho foi utilizado o software AMBERTOOLS15 (CASE et al,2015), no qual foi obtido valores de energias e RMSD, possibilitando analisar a espontaneidade das interações no sistema.
Material e métodos
Inicialmente a molécula de cocaetileno foi desenhada utilizando o software HyperChem 6.01 (CHEMPLUS, 2000) , posteriormente, otimizamos essa estrutura através do modelo contínuo polarizável (PCM) utilizando o software Gaussian09W (FRISCH et al, 2009) para otimização foi usado o método DFT através do funcional B3LYP. Na preparação do receptor foi utilizado uma secção de DNA na conformação B-DNA constituído por 12 bases nitrogenadas na sequência d[CGCGAATTCGCG]2 solvatado em água. Obtida a partir do Protein Data Bank (PDB; http://www.rcsb.org/pdb/), a secção de DNA (PDB ID: 1BNA), na forma cristalizada, foi separada dos resíduos de água e em seguida otimizada através do software AmberTools15 (CASE et al,2015) com o campo de força standard AMBER ff14SB force field, com a finalidade de obter a estrutura de menor energia e consequentemente mais estável. O sistema CE-DNA encontra-se hidratado e com presença de íons Na+, com o objetivo de manter o sistema eletricamente neutro. Finalmente, realizaram-se as simulações de dinâmica molecular (DM) com o package AmberTools15(CASE et al,2015). Na qual foram aplicados os campos de força standard AMBER ff14SB force field e General Amber Force Field (GAFF), o tempo de simulação utilizado foi de 20 ns, estando de acordo com o que foi encontrado em alguns estudos de dinâmica molecular para sistemas biológicos similares ao desse estudo(SONG et al, 2015).
Resultado e discussão
Durante a dinâmica molecular, cujo tempo de simulação foi de 20 ns,
a molécula de cocaetileno permaneceu aderida a superfície do sulco menor do
DNA (Figura 1) apresentando fortes interações do tipo Van Der Waals. O
sistema CE-DNA apresentou alta estabilidade no período de simulação, como
pode ser observado no gráfico RMSD da Figura 2, no qual obtivemos valores
RMSD < 1,6 Ǻ, de acordo com estudos quanto menor a variação em Å do RMSD
mais estável é o sistema analisado.
Logo após os 15 ns de simulação podem ser observadas poucas variações no
gráfico, apresentando uma considerável flutuação após este instante, tal
variação está associada a uma reconformação da extremidade do DNA
para melhor encaixe da molécula de CE. Por meio do valor de energia de Gibbs
obtido ΔG= -96,57 kJ/mol,pode-se estimar a espontaneidade da reação
do sistema CE-DNA, sendo que a ΔEVDW = -147,15 kJ/mol, contribuiu
significativamente para tal resultado. Os resultados obtidos estimam um
possível efeito genotóxico do cocaetileno.
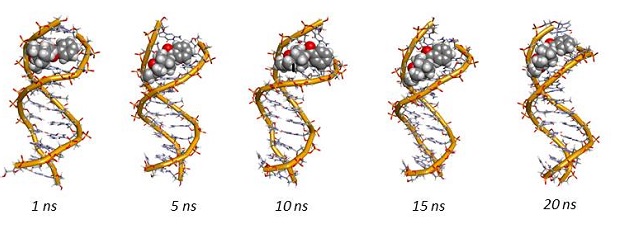
Snapshot da dinâmica Molecular do sistema Cocaetileno-DNA durante 20 ns de simulação.
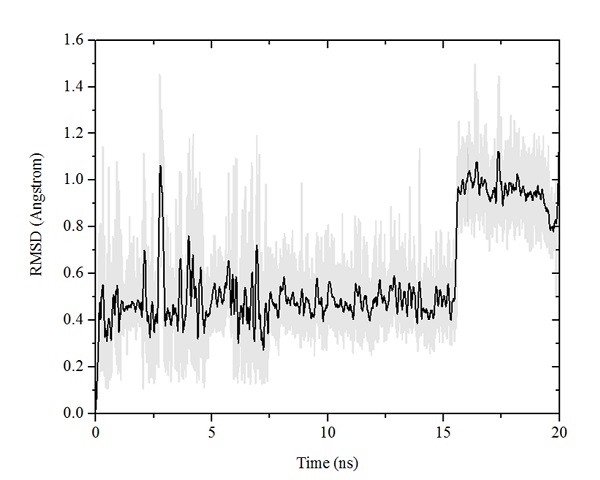
Gráfico Root-mean-square deviation (RMSD) da dinâmica molecular do sistema CE-DNA
Conclusões
Por meio dos resultados obtidos na simulação de dinâmica molecular foi possível verificar que o cocaetileno pode apresentar potencial genotóxico, indicando que a combinação de cocaína com álcool é capaz de causar danos extremos ao organismo.
Agradecimentos
Agradecemos ao suporte dado por: UFPA, PROEX, PROAD, Capes, CNPq e PROPESP.
Referências
CASE, D.A.; BERRYMAN, J.T.; BETZ, R.M.; CERUTTI, D.S.; CHEATHAM, T.E.; III, DARDEN, T.A.; DUKE, R.E.; GIESE, T.J.; GOHLKE, H; GOETZ, A.W.; HOMEYER, N; IZADI, S; JANOWSKI, P; KAUS, J; KOVALENKO, A; LEE, T.S.; LEGRAND, S; LI, P; LUCHKO, T; LUO, R; MADEJ, B; MERZ, K.M.; MONARD, G; NEEDHAM, P; NGUYEN, H; NGUYEN, H.T.; OMELYAN, I; ONUFRIEV, A; ROE, D.R.; ROITBERG, A; SALOMON-FERRER, R; SIMMERLING, C.L.; SMITH, W; SWAILS, J; WALKER, R.C.; WANG, J; WOLF, R.M.; WU, X; YORK. D.M.; KOLLMAN, P.A. (2015), AMBER 2015, University of California, San Francisco.
CHEMPLUS: Modular Extensions for HyperChem Release 6.02, Molecular Modeling for Windows, HyperClub, Inc., Gainesville, 2000.
FRISCH, M.J; TRUCKS, G.W; SCHLEGEL, H.B; SCUSERIA, G.E; ROBB, M.A; CHEESEMAN, J.R; SCALMANI, G; BARONE, V; MENNUCCI, B; PETERSSON, G.A; NAKATSUJI, H; CARICATO, M; LI, X; HRATCHIAN, H.P; IZMAYLOV, A.F; BLOINO, J; ZHENG, G; SONNENBERG, J.L; HADA, M; EHARA, M; TOYOTA, K; FUKUDA, R; HASEGAWA, J; ISHIDA, M; NAKAJIMA, T; HONDA, Y; KITAO, O; NAKAI, H; VREVEN, T; MONTGOMERY J.R.J.A; PERALTA, J.E; OGLIARO, F; BEARPARK, M; HEYD, J.J; BROTHERS, E; KUDIN K, N; STAROVEROV, V.N; KOBAYASHI, R; NORMAND, J; RAGHAVACHARI, K; RENDELL, A; BURANT, J.C; IYENGAR, S.S; TOMASI, J; COSSI, M; REGA, N; MILLAM, J.M; KLENE, M; KNOX, J.E; CROSS, J.B; BAKKEN, V; ADAMO, C; JARAMILLO, J; GOMPERTS, R; STRATMANN, R.E; YAZYEV, O; AUSTIN, A.J; CAMMI, R; POMELLI, C; OCHTERSKI, J.W; MARTIN, R.L; MOROKUMA, K; ZAKRZEWSKI V, G; VOTH, G.A; SALVADOR, P; DANNENBERG, J.J; DAPPRICH, S; DANIELS, A.D; FARKAS, Ö; FORESMAN, J.B; ORTIZ, J .V; CIOSLOWSKI, J; Fox D J Gaussian, Gaussian 09W, REVISION A.1, Inc., Wallingford CT, (2009)
RCSB PDB PROTEIN DATA BANK. Disponível em: <http://www.rcsb.org/pdb/>. Acesso em: 11 de out. 2015
SONG, C.; ZHANG, X.; LI, F.; ZHU, Y.;LIU, C.;LI, X.; LI, Y.;ZHANG, W.; WEI, D.;TANG, M. Theoretical study of binding affinity for diamidine with DNA. Struct Chem. n° 27, 681–696, 2015.
United Nations Office on Drugs and Crime. World Drug Report 2015. Disponível em: <http://www.unodoc.org/wdr2015/> . Acesso em: 11 de março. 2016.
WILSON, L.D.; JEROMIN, J; GAARVY, L; DORBANDT, A. Cocaine, Ethanol, and Cocaethylene Cardiotoxity in an Animal Model of Cocaine and Ethanol Abuse. Academic Emergency Medicine, v. 8, n° 3, mar. 2011.











