ISBN 978-85-85905-19-4
Área
Química Tecnológica
Autores
Vogado Abrahão, J.H. (UFPA) ; Viana Cruz, J. (UNIFAP) ; Campos Silva, R. (UFPA) ; Carvalho Lobato, C. (UFPA) ; Belém dos Santos, K.L. (UFPA) ; da Silva Costa, J. (UFPA) ; da Silva Hage-melim, L.I. (UNIFAP) ; do Socorro Barros Brasil, D. (UFPA) ; de Lima Barros, C.A. (UFPA) ; Rodrigues dos Santos, C.B. (UNIFAP)
Resumo
Cepas bacterianas resistentes a diferentes grupos de antibióticos surgem e dificultam o tratamento de enfermos e o combate da disseminação dos diversos níveis de infeções hospitalares. As Proteínas de Ligação à Penicilina/“Penicillin Binding Proteins” (PLPs/PBPs) são alvos biológicos importantes para o desenvolvimento de novos fármacos contra infecções bacterianas. Este trabalho busca conhecer e elucidar as propriedades farmacocinéticas e toxicológicas dos 20 melhores compostos obtidos via triagem virtual baseado em farmacóforo decorrente do alinhamento de 12 compostos relacionados com o complexo formado pela Proteína de Ligação às Penicilinas 1B (PBP-1B) e um baronato de aquila (ZA2), visando o planejamento de novos fármacos mais seletivos e eficazes no combate às infecções bacterianas.
Palavras chaves
Bactérias; PLPs; β-lactâmicos
Introdução
O desenvolvimento de fármacos eficientes no combate a infecções bacterianas diminuiu drasticamente a mortalidade causada por doenças microbianas. A disseminação do uso de antibióticos fez com que as bactérias desenvolvessem defesas relativas aos agentes antibacterianos, com o consequente aparecimento de resistência. Esta resistência prolifera-se rapidamente através de transferência genética, estando presente nas espécies de bactérias Gram-positivas mais prevalentes, como enterococos, estafilococos e estreptococos (SILVEIRA et al, 2006). As proteínas de ligação à Penicilina/“Penicillin Binding Proteins” (PLPs/PBPs) constituem um grupo de proteínas caracterizado pela sua afinidade de ligação a antibióticos β- lactâmicos. A inibição das PBPs leva a irregularidades na estrutura da parede celular. Os antibióticos β-lactâmicos ligam-se às PBPs em razão da semelhança estrutural desses antibióticos com o grupo terminal D-alanil-D- alanina das unidades de peptidoglicano da parede celular bacteriana. Os antibióticos β-lactâmicos inibem a transpeptidase mediante uma ligação covalente com o resíduo de serina do sítio ativo das PBPs. O desenho de novas estruturas está relacionada com a predição do modo de ligação e a afinidade de interação de moléculas no sítio de ligação de uma proteína (VERDONK et al, 2003). Desta forma, estudos de interação fármaco-ligante utilizando métodos computacionais possuem papel importante na procura por fármacos com atividade farmacológica. Assim, realizou-se o estudo computacional e das propriedades farmacocinéticas de absorção intestinal humana (HIA), permeabilidade das células (PCaCO2), ligação às proteínas plasmáticas (PPB) e penetração na barreira hemato-encefálica (BHE) e toxicológicas das proteínas de ligação à Penicilina complexada ao Boronato.
Material e métodos
A) Seleção e Modelagem dos Compostos – Neste trabalho foi selecionado inicialmente a forma cristalográfica do complexo formado entre a Proteína de Ligação às Penicilinas 1B (PBP-1B) com um baronato de aquila (ZA2) [Protein Data Base - 2Y2H]. A partir da seleção desta molécula pivô, foram selecionados outras 12 moléculas (Fig. 1) mais ativos, baseados no valor de Ki, e estruturalmente semelhantes a molécula pivô por meio da ferramenta online BindingDataBase (https://www.bindingdb.org) (LIU et al, 2007). A partir da seleção, os 12 compostos foram aprimorados por meio da utilização da ferramenta Discovery Studio 4.0 (http://www.accelrys.com) (BIOVIA et al, 2015). B) Geração do Grupo Farmacofórico – A derivação do farmacóforo foi realizada com o auxilio do servidor web PharmaGist (http://bioinfo3d.cs.tau.ac.il/PharmaGist/) (SCHNEIDMAN-DUHOVNY et al, 2008) utilizando as 12 moléculas selecionados a partir de BindingDB com o objetivo de determinar o grupo farmacofórico, tomando como referência a forma cristalográfica da molécula pivô obtida do PDB (código 2Y2H). C) Triagem Virtual Baseado em Farmacoforo – Esta etapa foi realizada utilizando o ZincPharmer (http://zincpharmer.csb.pitt.edu/) (KOES; CAMACHO, 2012) a partir da busca por compostos com maior similaridade com farmacóforo baseadas no menor valor do RMSD na base de dados da ZINC. D) Predição das Propriedades Farmacocinéticas e Toxicológicas – As propriedades de absorção, distribuição, metabolismo, excreção e toxidade (ADME/Tox) foram realizadas apenas para 20 melhores compostos selecionados via ZincPharmer por meio da utilização do preADMET (http://preadmet.bmdrc.org/).
Resultado e discussão
A derivação do farmacóforo determinado pelo PharmaGist, obteve um score de
18.000 (ver Fig. 1). Os compostos alinhados compartilham 3 características
espaciais: uma região doadora (cor amarela) e duas receptoras de hidrogênio
(cor verde). Analisando os resultados dos testes de ADME das moléculas
selecionadas. Foi observado que os compostos apresentaram valores de
absorção intestinal humana (HIA) num intervalo de 14,5347 ≤ HIA% < 95,1089.
Sendo o destaque para os compostos #10 e #11, que demostraram os mais
elevados graus de HIA, com valores acima de de 95%. Os compostos
analisados, mostram conforme dados apresentados uma permeabilidade média de
16,7086 nm/s. Foi verificado que a PCaCO2 (nm / s) foi moderada, variando de
1,5477nm/s a 21,1954 nm/s. Apenas os compostos #10, #11 e #15 apresentaram
valores superiores a 90%, o que demostra alta capacidade de ligação às
proteínas plasmáticas. Os compostos que apresentam valores mais altos de 1
(CBrain / CBlood> 1) são considerados ativos no SNC. Todos os compostos
analisados apresentam valores inferiores a 1 (CBrain / CBlood <1), sendo
classificados como inativos no SNC.
O ensaio de carcinogenicidade tem o objetivo de identificar o potencial
cancerígeno em animais e avaliar o risco disto em seres humanos. Neste
teste, foi identificado que os compostos #13, #14, #17, #18, #19 e #20
possuem predição não carcinogênica. Enquanto os restantes dos compostos
apresentam predição carcinogênica. Ao analisar carcinogenicidade em ratos,
os compostos #1, #3 e #15 foram classificados como positivos, mostrando
evidencias não carcinogênicas e os demais compostos, apresentado previsões
negativas que mostram fortes evidências cancerígenas em ratos.
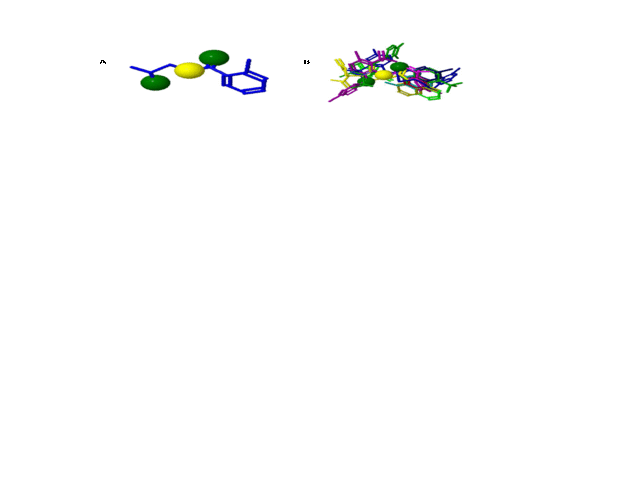
Grupo farmacofórico da molécula pivô (A) e o alinhamento dos 12 compostos selecionados mais ativos, baseados no valor de Ki (B).
Conclusões
Após análise, considerando a necessidade de elencar moléculas com potencial de utilização como medicamentos e com objetivo de aprofundamento, apenas as moléculas #13, #14 e #20, apresentam as qualidades necessárias para eventuais candidatos ao desenvolvimento racional de fármacos mais eficientes e com menos efeitos indesejáveis. Nenhuma das 20 moléculas atenderam, simultaneamente, os três critérios checados pelos testes toxicológicos, o que demonstra, que há a necessidade de aprofundar as análises relativas ao potencial mutagênico e carcinogênico destas substâncias.
Agradecimentos
Referências
BIOVIA, D. S. Discovery Studio Modeling Environment, Release 4.5, San Diego: Dassault Systèmes, 2015.
KOES, R. D; CAMACHO, C. J. ZINCPharmer: pharmacophore search of the ZINC database. Nucleic Acids Research, v. 40, 2012.
LIU, T.; LIN, Y.; WEN, X.; JORISSEN, R. N.; GILSON, M. K. BindingDB: a web-accessible database of experimentally determined protein-ligand binding affinities. Nuceic Acids Research, v. 35, p. 198-201, 2007.
SCHNEIDMAN-DUHOVNY, D.; DROR, O.; INBAR, Y.; NUSSINOV, R.; WOLFSON, H. J. PharmaGist: a webserver for ligand-based pharmacophore detection. Nucleic Acids Research, 2008.
SILVEIRA, G. P. et al. Estratégias utilizadas no combate a resistência bacteriana. Quim. Nova, Florianópolis, v. 29, p. 844-855, mar. 2006.
VERDONK, M.L.; COLE, J.C.; HARTSHORN, M.J.; MURRAY, C.W.; TAYLOR, R.D. Improved protein-ligand docking using GOLD. Proteins: Structure, Functions and Bioinformatics, 52: 609, 2003.











