Correlação entre atividade biológica e interação com ctDNA de piperidinas funcionalizadas empregando técnicas espectroscópicas e estudos teóricos
ISBN 978-85-85905-21-7
Área
Química Orgânica
Autores
de Magalhães Silva, M. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL) ; de Araújo Dantas, M.D. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL) ; Carinhanha Caldas Santos, J. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL) ; Martins Figueiredo, I. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL) ; das, S. (UNIVERSIDADE DE VISVA-BHARATI) ; Brahmachari, G. (UNIVERSIDADE DE VISVA-BHARATI) ; de Fátima, (UNIVERSIDADE FEDERAL DE MINAS GERAIS) ; Tasca Góis Ruiz, A.L. (UNIVERSIDADE ESTADUAL DE CAMPINAS) ; Ferreira da Silva-júnior, E. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL) ; Mendonça de Aquino, T. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL)
Resumo
A interação entre os derivados de piperidinas e o ctDNA (Calf thymus) foi avaliada empregando UV-vis e fluorescência molecular. Por meio dos estudos de interação, verificou-se que os compostos avaliados interagem com o modelo de DNA utilizado, havendo formação de complexo supramolecular com valores das constantes de ligação (Kb) variando de 0,10 a 8,00 x 104 L mol-1, sendo intercalação o modo de ligação preferencial. Adicionalmente, os resultados espectroscópicos foram confirmados pelos ensaios de dinâmica e docking molecular. Por fim, observou-se correlação linear entre os valores de GI50 e Kb para diferentes compostos, indicando que o mecanismo de ação desses compostos pode estar relacionado a interação com DNA como principal alvo biológico.
Palavras chaves
Interação com ctDNA; correlação bioanalítica; técnicas espectroscópicas
Introdução
Estudos sobre o mecanismo de ligação de pequenas moléculas com o DNA foram identificados como um dos principais temas durante os últimos anos (DORRAJI; JALALI, 2013; ZHAO et al, 2014; HASSAN; RAUF, 2015). Dentro deste contexto geral, as interações DNA-ligante visando à obtenção de fármacos são de particular importância, pois o DNA constitui o alvo intracelular para uma vasta gama de medicamentos, visto que, moléculas pequenas podem interagir com proteínas associada ao DNA, híbridos de DNA-RNA e diretamente com a dupla hélice do ácido nucleico. Assim, as moléculas avaliadas nesse trabalho foram os derivados de piperidinas, que são compostos heterocíclicos contendo nitrogênio, amplamente encontrados em produtos naturais e farmacêuticos (BEHENA et al, 2012). Muitos deles desempenham funções essenciais no organismo humano e apresentam propriedades biológicas significativas, tais como: anti- hipertensivos, antibacterianos, antiproliferativa, antioxidantes, entre outras (PATIL et al, 2014). Atualmente, vários compostos que contêm o núcleo de piperidina são empregados como fármacos no tratamento de diferentes patologias, como por exemplo, Donepezil, Pipamperona e Vimblastina. Assim, devido a importância do núcleo piperidínico presente em diferentes fármacos, o objetivo desse trabalho consiste em avaliar a interação dos derivados de piperidinas com o DNA, buscando o entendimento do mecanismo de ação desses compostos, uma vez que diversas patologias tem o DNA como principal alvo biológico.
Material e métodos
Todos os reagentes utilizados foram de alto grau de pureza. A solução estoque do DNA Calf Thymus (ctDNA, USA, Sigma Aldrich) foi preparada em tampão Tris-HCl (10 mmol L-1, pH = 7,4 ± 0,10) com 0,1 mol L-1 de NaCl para o ajuste da força iônica. Para avaliar a pureza do ácido nucleico, mediu-se a absorvância (em espectrofotômetro Micronal de feixe duplo modelo AJX- 6100PC) em 260 e 280 nm, sendo que quando a razão A260/A280 = 1,8-1,9, indica que a macromolécula está livre de contaminação por proteínas. Além disso, para calcular a concentração do DNA utilizou-se a A260, com coeficiente de extinção molar de 6600 L mol-1. Os derivados piperidínicos foram inicialmente solubilizados em DMSO (USA, Sigma Aldrich) por apresentar baixa solubilidade em meio aquoso, e depois diluídos em solução tampão. Nos estudos por titulação fluorimétrica (espectrofluorímetro Shimadzu, modelo RF 5301) manteve-se a concentração de cada composto fixa em 10 µmol L-1, e adicionou-se incrementos de DNA que variaram de 10 a 200 µmol L-1. Nos ensaios para avaliar o modo de ligação com o ctDNA, foi empregado uma solução estoque de iodeto de potássio a 0,2 mol L-1, e uma solução de brometo de etídio a 0,5 mmol L-1, utilizando como parâmetros instrumentais λex = 525 nm, λem = 590 nm com slit de 5 nm para excitação e 3 nm para emissão. Para os ensaios de dinâmica molecular utilizou-se a estrutura de cristal de raio-X do dodecâmero ctDNA (CGCGAATTCGCG) (PDB ID: 1BNA), disponível no doi 10.2210/pdb1bna/pdb. Todos os ensios dinâmicos moleculares e os estudos de docking foram realizados de acordo com SILVA et al, 2016.
Resultado e discussão
As estruturas dos compostos avaliados são apresentados no Esquema 1a, os
quais foram sintetizados e caracterizados previamente (BRAHMACHARI, DAS,
2012). Nos estudos por fluorescência molecular, verificou-se que ao
adicionar quantidades crescentes de ctDNA ao sistema, ocorreu decréscimo
gradual no sinal analítico, em virtude da interação entre o DNA e os
ligantes avaliados (Esquema 1b). Segundo MUKHERJEE e SING (2017) esse
fenômeno é denominado de quenching, e pode ocorrer por diferentes
mecanismos. Para avaliar a força da interação, fez-se a linearização do
espectro (Esquema 1b) e calculou-se a constante de ligação entre os ligantes
avaliados e a macromolécula.
Os valores da constante de interação (Kb) para os compostos avaliados
variaram de 0,10 a 8,0 × 104 L mol-1 como apresentado no Esquema 1c,
demonstrando a magnitude da interação ctDNA-ligante. De forma geral, para
maioria dos compostos o valor de Kb indica afinidade média, com exceção do
composto 6 que apresentou constante na ordem de 102, sendo classificada de
baixa afinidade (SILVA et al, 2017). Assim, em termos gerais observou-se que
os valores das constantes de ligação com ctDNA seguiram a seguinte ordem: 5
> 7 > 4 > 8 > 3 > 1 > 2 > 6, sendo o composto mais ativo e seletivo (5) o
que exibiu maior valor de Kb e por consequência, os menos ativos (2 e 6) os
que apresentaram os menores valores de constantes de ligação.
Os resultados obtidos são concordantes com trabalhos da literatura, que
avaliaram a interação de diferentes compostos com o núcleo piperidínico com
DNA, como morfina (Kb = 0,39 × 104 L mol-1), vincristina (Kb = 1,70 × 104 L
mol-1), vimblastina (Kb = 0,17 × 104 L mol-1) e berberina (Kb = 1,55 × 104 L
mol-1) (LI, DONG, 2009; MOHAMMADGHOLI et al, 2013; LI et al, 2012). Além
disto, a relação estequiométrica entre DNA:ligante foi igual a 1:1 para os
compostos avaliados, e os valores de ΔG foram todos negativos,
caracterizando o processo de interação como espontâneo.
Para inferir sobre o mecanismo de ação dessa classe de compostos, se avaliou
a correlação entre os valores de constantes de interação (Kb) das
piperidinas com ctDNA e a atividade citotóxica (GI50) referente as linhagens
de células cancerígenas humanas, onde observou-se que o composto que
apresentou maior interação com ctDNA (derivado 5), de forma geral, foi o
mais ativo frente as linhagens de células avaliadas; enquanto que os
compostos menos ativos (derivados 2 e 6) foram aqueles com as menores
constantes de interação com ctDNA (Esquema 1d). Os valores dos coeficientes
de correlação obtidos pelo gráfico GI50 vs Kb apresentaram coeficiente de
correlação na faixa de 0,8174 ≤ r ≤ 0,9868 (considerando o valor de r em
módulo), sendo que as linhagens HT29, NCI-H460 e 786-0 apresentaram
tendência linear inversa, enquanto para NCI/ADR-RES se observou relação
exponencial inversa. Para estas linhagens o valor de GI50 foi inversamente
proporcional a Kb, indicando que a interação com DNA pode ser um dos
possíveis mecanismos de ação destes compostos.
Nos ensaios por UV-vis verificou-se que ao adicionar ctDNA ao sistema
ocorreu um aumento na absorvância, denominado de efeito hipercrômico,
indicando que está ocorrendo interação entre o ligante e a biomolécula, com
possível formação de complexo. Para confirmar a existência dessa interação,
fez-se um espectro da diferença entre o complexo ctDNA-piperidina e o ctDNA
livre, verificando-se que os espectros de absorção dos derivados
piperidinicos não são sobreponíveis. Estas mudanças espectrais são melhores
observadas a partir dos valores de absorvância da mistura ligante-ctDNA
(Acomplexo = 0,8626), e a soma dos valores das absorvâncias do composto
livre e ligado ao ctDNA (A5 + ActDNA = 0,5407). Por meio da equação ΔA =
Acomplexo - (Acomposto + ActDNA), verifica-se que o valor de ΔA ≠ 0 (ΔA=
0,3219), o qual indica que ocorreram alterações no estado fundamental,
caracterizando um mecanismo preferencial de quenching estático, devido à
formação de complexo macromolécula-ligante, reforçando a proposta obtida
pelos resultados por fluorescência molecular. Entretanto, caso o quenching
dinâmico fosse o mecanismo predominante no processo de interação, não se
esperaria alterações, uma vez que este afeta somente o estado excitado.
O modo de ligação dos derivados piperidinicos e o ctDNA foi avaliado a
partir de dois ensaios: estudo do quenching pelo KI e competição com brometo
de etídio, apresentados na Figura 1.
Os estudos de quenching de fluorescência fornecem informações sobre a
acessibilidade dos ligantes a molécula supressora de fluorescência, no caso
o íon iodeto. Dessa forma, avalia-se a magnitude da constante de Stern-
Volmer na presença e ausência de ctDNA. Nota-se que os valores de KSV
(constante de quenching de Stern-Volmer) na presença de ctDNA (barras em
cinza) foram sistematicamente menores que na ausência da macromolécula
(barras em vermelho), indicando que o ânion iodeto não conseguiu ter acesso
aos ligantes, sugerindo que estes estão protegidos pelos pares de bases do
DNA, e assim, impedindo o acesso do supressor aniônico (Figura 1a). Logo,
pode-se inferir que o principal modo de ligação desses compostos ao ctDNA
ocorrem via intercalação. Para confirmar esses resultados realizou-se um
estudo de competição com o brometo de etídio, uma sonda clássica que liga-se
ao DNA via intercalação (USMAN, AHMAD, 2017). Em solução aquosa, esse
competidor quando livre apresenta baixa fluorescência, no entanto quando
liga-se ao DNA seu sinal aumenta significativamente devido a sua localização
entre os pares de bases nitrogenadas. Assim, qualquer pequena molécula que
substitua o EB na macromolécula irá interagir com o mesmo modo de ligação
que a sonda. Nesse sentido, a Figura 1b mostra que, ao adicionar quantidades
crescentes do composto 5 ao sistema, ocorre uma diminuição gradual no sinal
de fluorescência, evidenciando que o brometo de etídio está sendo deslocado
do ctDNA. O percentual de decréscimo do sinal variou de 42,9 a 61,9% (Figura
1c), utilizando um excesso de até 30 vezes dos ligantes em relação à
quantidade inicial de brometo de etídio, sugerindo assim que os compostos
avaliados interagem preferencialmente por intercalação, confirmando os
resultados anteriores.
Adicionalmente, pelos ensaios de dinâmica e docking molecular, verificou-se
que o modo de ligação preferencial é via intercalação e que as interações
hidrofóbicas são as dominantes no processo de interação.
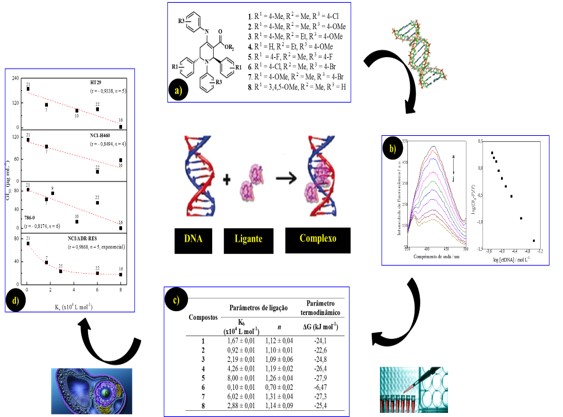
Estudos de interação DNA-ligante; a)Estruturas das moléculas avaliadas; b)Titulação fluorimétrica; c)Parâmetros de ligação; d)Correlação bioanalítica.
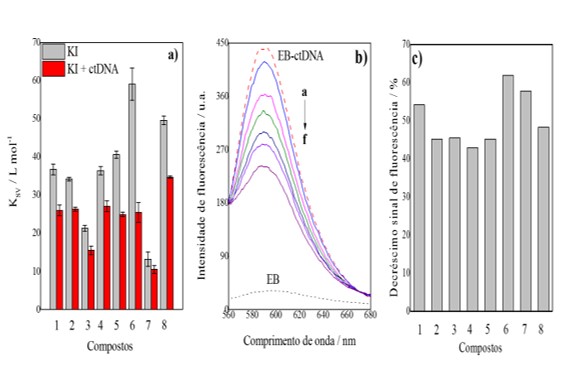
Avaliação do modo de interação DNA ligante;a) Valores das constantes de Stern- Volmer;b)Competição com brometo de etídio;c)Decréscimo da fluorescência.
Conclusões
Verificou-se pelos estudos de interação com o ctDNA, que as moléculas avaliadas interagem com o ctDNA, havendo formação de complexo supramolecular entre os ligantes e a macromolécula, onde os valores de Kb variaram de 0,10 a 8,00 x 104 L mol-1. Além disso, por meio da correlação das constantes de ligação com os valores de GI50, pôde-se observar que os coeficientes de correlação variaram na faixa de 0,8174 ≤ r ≤ 0,9868 (considerando o valor de r em módulo, para as linhagens HT29, NCI-H460, 786-0 e NCI/ADR-RES, sugerindo que o mecanismo de ação preponderante desses compostos pode estar associado ao DNA como alvo biológico. Os ensaios de KI, competição com brometo de etídio e estudos teóricos sugeriram que os compostos avaliados interagem com o DNA preferencialmente via intercalação. Por fim, os estudos de docking e dinâmica molecular confirmaram os resultados espectroscópicos obtidos.
Agradecimentos
Os autores agradecem ao IQB-UFAL, CAPES, CNPq e FAPEAL.
Referências
BEHENNA, D. C.; LIU, Y.; YURINO, T.; KIM, J.; WHITE, D. E.; VIRGIL, S. C.; STOLTZ, B. M. Enantioselective Construction of Quaternary N-Heterocycles by Palladium-Catalyzed Decarboxylative Allylic Alkylation of Lactams. Nature Chemistry, 4, p. 130-133, 2012.
BRAHMACHARI, G.; DAS, S. Bismuth nitrate-catalyzed multicomponent reaction for efficient and one-pot synthesis of densely functionalized piperidine scaffolds at room temperature. Tetrahedron Letters, 53, p. 1479-1484, 2012.
DORRAJI, P. S.; JALALI, F. Investigation of the Interaction of Sertraline with Calf Thymus DNA by Spectroscopic Methods. Journal of the Brazilian Chemical Society, 24, p. 939-945, 2013.
HASSAN, M. F.; RAUF, A. Synthesis andmulti-spectroscopic DNA binding study of 1,3,4-oxadiazole and 1,3,4-thiadiazole derivatives of fatty acid. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 153, p. 510–516, 2015.
LI, J. F.; DONG, C. Study on the interaction of morphine chloride with deoxyribonucleic acid by fluorescence method, Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 71, p. 1938-1943, 2009.
Li, X.; Hu, Y.; Wang, H.; Yu, B.; Yue, H. Molecular spectroscopy evidence of berberine binding to DNA: comparative binding and thermodynamic profile of intercalation. Biomacromolecule, 13, p. 873-880, 2012.
MOHAMMADGHOLI, A.; RABBANI-CHADEGANI, A.; FALLAH, S. Mechanism of the Interaction of Plant Alkaloid Vincristine with DNA and Chromatin: Spectroscopic Study, DNA and Cell Biology, 32, p. 228-235, 2013.
MUKHERJEE, A.; SINGH, B. Binding interaction of pharmaceutical drug captopril with calf thymus DNA: a multispectroscopic and molecular docking study. Journal of Luminescence, 190, p. 319-327, 2017.
PATIL, P. O.; BARI, S. B. Nitrogen heterocycles as potential monoamine oxidase inhibitors: Synthetic aspects. Arabian Journal of Chemistry, 7, p. 857-884, 2014.
SILVA, M. M.; SAVARIZ, F. C.; SILVA-JÚNIOR, E. F.; DE AQUINO, T. M.; SARRAGIOTTO, M. H.; SANTOS, J. C. C.; FIGUEIREDO, I. M. Interaction of β-Carbolines with DNA: Spectroscopic Studies, Correlation with Biological Activity and Molecular Docking. Journal Brazilian Chemical Society, 27, p. 1558-1568, 2016.
USMAN, A.; AHMAD, M. Binding of Bisphenol-F, a bisphenol analogue, to calf thymus DNA by multi-spectroscopic and molecular docking studies. Chemosphere, 181, p. 536-543, 2017.
ZHAO, T.; BI, S.; WANG, Y.; WANG, T.; PANG, B.; GU, T. In vitro studies on the behavior of salmeterol xinafoate and its interaction with calf thymus DNA by multi-spectroscopic techniques. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 132, p. 198–204, 2014.
































