Construção de um banco de DNA metagenômico de diferentes ecossistemas com potencial biotecnológico relevante da Baixada Fluminense do Estado do Rio de Janeiro
ISBN 978-85-85905-23-1
Área
Bioquímica e Biotecnologia
Autores
Cugler da Silva, R. (IFRJ) ; Rocha Castro, M. (IFRJ) ; da Silva Pissurno, B. (IFRJ) ; Silva de Oliveira, M. (IFRJ) ; de Souza Pinheiro, V. (IFRJ)
Resumo
A Baixada Fluminense localizada em Duque de Caxias/RJ possui ecossistemas ainda conservados e áreas de proteção ambiental que apresentam uma biodiversidade endêmica expressiva e um potencial biotecnológico relevante. O objetivo do trabalho proposto é construir um banco de DNA utilizando uma abordagem metagenômica para investigar a diversidade de micro-organismos presentes em diferentes ecossistemas (terrestres e aquáticos) localizados nessa região. Um protocolo de extração de DNA metagenômico economicamente viável foi otimizado. Os resultados obtidos apontam a necessidade de ajustes relacionados a concentração e a qualidade do material genético extraído. Com a padronização da metodologia, há a possibilidade de garantir a conservação do patrimônio genético dos ecossistemas de interesse.
Palavras chaves
Banco de DNA; Metagenômica; Potencial biotecnológico
Introdução
Os bancos de DNA presentes no Brasil são de coleções biológicas de espécies nativas ameaçadas de extinção. Portanto, até o momento não existem bancos de DNA metagenômico, com ênfase na diversidade de micro-organismos autóctones e/ou endêmicos que habitam ecossistemas com potencial biotecnológico relevante. O termo metagenômica se refere a uma abordagem independente de cultivo baseada na investigação das moléculas de DNA de uma mistura de populações microbianas, extraído diretamente de amostras ambientais (THOMAS et al., 2012). Existem métodos para extração de DNA que incluem o uso de produtos químicos e equipamentos sofisticados, porém consomem muito tempo e reagentes. Os kits para extração de DNA metagenômico disponíveis no mercado, possuem procedimentos de fácil execução em um tempo relativamente curto, no entanto, os custos são ainda muito elevados (DEVI et al., 2015). Por conseguinte, torna-se pertinente um estudo para otimização de um protocolo de extração de DNA metagenômico, com a finalidade da construção de um banco de DNA metagenômico. O manguezal localizado na área de proteção ambiental-APA de Guapimirim no Recôncavo da Baía de Guanabara/RJ já foi atingido por inúmeros acidentes relacionados ao vazamento de petróleo. O acidente ocorrido na Refinaria Duque de Caxias da Petrobrás, no entanto, foi o maior registrado. Outras áreas de importância localizadas na Baixada Fluminense e que apresentam relevante potencial biotecnológico também já tiveram seus ecossistemas suprimidos significativamente. Portanto, a partir da otimização de um protocolo de extração de DNA metagenômico, será possível investigar a diversidade de micro-organismos presentes no sedimento de mangue localizado na APA de Guapimirim-RJ, no intuito de se estabelecer uma metodologia eficiente e economicamente viável para a construção de um banco de DNA metagenômico com potencial biotecnológico.
Material e métodos
A extração do DNA metagenômico baseou-se no protocolo segundo Tagliaferro (2005) e Yeates et al. (1998) modificado da seguinte forma: Coletou-se 6,5 gramas de solo na área externa do campus IFRJ Duque de Caxias. Juntou-se com a amostra de solo 6,5 g de pó de vidro com 14 mL de tampão de extração. Em 1 min agitou-se no vórtex e colocou-se no gelo por mais 1 min. Repetiu-se 5x. Adicionou-se 1,5 mL de SDS 20% homogeneizando. Incubou-se por 1 h a 65°C agitando-se a cada 15 min e centrifugou-se por 15min, transferiu-se 1 mL do sobrenadante para os tubos. Os tubos foram incubados por 15 min a temperatura ambiente e centrifugados por mais 15 min. Adicionou-se 1 mL de isopropanol 100 % descartando-se o sobrenadante e ressuspendeu-se o precipitado em 800 µl de tampão TE com 1 mL da solução de 13% PEG 8000 e NaCl 1,6 M. Incubou-se por 2 dias a temperatura ambiente. Os tubos foram novamente centrifugados por 20 min e o sobrenadante foi descartado. Ressuspendeu-se o precipitado em 400 µl de tampão TE e foi adicionado 400 µl acetato de potássio 0,5 M. Incubou-se no gelo por 5 min e centrifugou-se por 20 min. Transferiu-se o sobrenadante para um novo tubo onde foram adicionados 1V de fenol homogeneizando-se por inversão. Centrifugou-se por 5 min e transferiu- se a fase aquosa para novos tubos. Repetiu por mais 3x o procedimento anterior. Adicionou-se 1V da solução de clorofórmio: álcool isoamílico (24:1) homogeneizando-se por inversão. Centrifugou-se por 5 min e transferiu-se a fase aquosa para um novo tubo. Repetiu-se a etapa anterior 1x. Coletou-se a fase aquosa para um novo tubo. O DNA foi então precipitado com 1V de isopropanol 100% e incubado por 1h a temperatura ambiente. Após, o mesmo foi centrifugado por 20 min. Descartou-se o sobrenadante e o precipitado foi lavado com 300 µl de etanol 70% gelado e centrifugado por 3 min. O precipitado foi mantido no tubo aberto por cerca de 30 min para facilitar a evaporação total do etanol. Após a evaporação o DNA foi ressuspendido em 30 µl de tampão TE e armazenado a -20°C no freezer. Na análise do tamanho dos fragmentos de DNA extraídos, foi realizada uma corrida eletroforética em gel de agarose 1% em tampão TAE. O gel foi corado com brometo de etídio a 0,01% (GREEN; SAMBROOK, 2012).
Resultado e discussão
O protocolo otimizado e adaptado mostrou-se eficiente para a extração do DNA
metagenômico do solo analisado. Os reagentes necessários para a execução do
procedimento são relativamente mais acessíveis economicamente quando
comparados com os Kits de biologia molecular comerciais disponíveis no
mercado. Entretanto, o tempo total do procedimento realizado é maior. Na
literatura, já foram descritos métodos de extração de DNA que se baseiam em
duas técnicas principais: extração de células de uma amostra de solo antes
da lise celular e lise direta das células. No caso da lise direta das
células, o método é mais rápido, porém a concentração de ácidos húmicos é
aumentada expressivamente, além de outras substâncias orgânicas presentes na
amostra (BACHOON; OTERO; HODSON, 2001). Como as substâncias húmicas se
tornam uma grande preocupação com a amplificação do DNA extraído de amostras
de solo ou sedimento, é importante que estas sejam removidas ou atenuadas,
pois as mesmas também podem inibir outras técnicas como, por exemplo, a PCR
(TSAI; OLSON, 1992). Na figura 1, observa-se que o tamanho do fragmento
obtido após a extração do DNA metagenômico está de acordo com o esperado, já
que o material genético apresenta um alto peso molecular. A qualidade e a
integridade do DNA extraído também foram avaliadas no gel de agarose 1%
(Figura 1). Embora não tenha sido possível realizar a quantificação do ácido
nucléico por espectrofotometria para análise da sua concentração e grau de
pureza, uma avaliação qualitativa foi possibilitada. A partir da
visualização das bandas, também não foram observados rastros que poderiam
evidenciar a degradação do material genético, o que ocasionaria uma
interferência negativa em análises moleculares posteriores. As mesmas se
apresentam com intensidade satisfatória, sugerindo que a concentração do DNA
foi adequada.
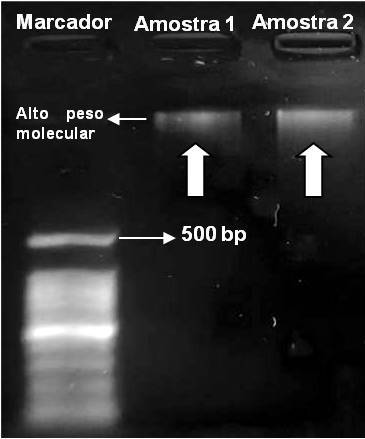
Gel de agarose 1% para visualização do DNA metagenômico extraído corado com brometo de etídio
Conclusões
Os resultados apontam a necessidade de adaptações nos procedimentos que se referem a lise celular, a purificação do ácido nucléico, a contaminação com proteínas e outros compostos, como os ácidos húmicos. Estes contaminantes são prejudiciais para análises moleculares futuras. Nos resultados não houveram degradação do material genético, porém a avaliação quantitativa do DNA metagenômico extraído é importante. Os parâmetros relacionados ao grau de pureza e a concentração do material genético necessitam de uma apuração mais especifica, através de métodos com maior sensibilidade e precisão. Com a padronização do protocolo otimizado, será possível a construção de um banco de DNA metagenômico de ecossistemas com potencial biotecnológico relevante da baixada fluminense, com ênfase na cidade de Duque de Caxias.
Agradecimentos
A Deus, pela força e sabedoria. Ao Instituto Federal de Educação, Ciência e Tecnologia do Rio de Janeiro Campus Duque de Caxias e a CNPq pela bolsa PIBIT concedida.
Referências
BACHOON, D. S.; OTERO, E.; HODSON, R. E. Effects of humic substances on fluorometric DNA quantification and DNA hybridization. Journal of Microbiological Methods, n.47, p. 73-82, 2001.
DEVI, S. G.; FATHIMA, A. A.; RADHA, S.; ARUNRAJ, R.; CURTIS, R.; RAMYA, M. A Rapid and Economical Method for Efficient DNA Extraction from Diverse Soils Suitable for Metagenomic Applications. Plos one,2015.
GREEN, M. R.; SAMBROOK, J. Molecular Cloning: a laboratory manual. 4. ed. New York: Cold Spring Harbor Laboratory Press, 2012.
TAGLIAFERRO, A. C. Análise molecular da diversidade bacteriana de solos do cerrado utilizando bibliotecas de rDNA 16S: Uma perspectiva biotecnológica. 2005. 125 f. Dissertação (Mestrado) - Curso de Ciências Genômicas e Biotecnologia, Universidade Católica de Brasília, Brasília, 2005
THOMAS, T.; GILBERT, J.; MEYER, F. Metagenomics - a guide from sampling to data analysis. Microbial Informatics and Experimentation, v. 2, n. 3, 2012.
TSAI, Y. L.; OLSON, B. H. Rapid Method for Separation of Bacterial DNA from Humic
Substances in Sediments for Polymerase Chain Reaction. Applied and environmental microbiology, v.58, n. 7, p. 2292-2295, 1992.
YEATES, C.; GILLINGS, M.R.; DAVISON, A.D.; ALTAVILLA, N.; VEAL, D.A. Methods for microbial DNA extraction from soil for PCR amplification. Biological Procedures Online, v. 1, n. 1, p. 40-47, 1998.











