ÁREA: Bioquímica e Biotecnologia
TÍTULO: MONITORAÇÃO DA OLIGOMERIZAÇÃO DE ZÍPERS DE LEUCINA FUSIONADOS A PROTEÍNAS FLUORESCENTES.
AUTORES: BUGS, M.R.(1); BORTOLETO-BUGS, R.K.(1); PAVANI FILHO, A.(1,2); MIRANDA, F.P.(1,3); HENRIQUE-SILVA, F.(1,4); GARCIA, J.W.(1,5)
1 PROJETO COGNITUS – CENPES/PETROBRÁS (MILTONBUGES@ITELEFONICA.COM.BR); 2 CENPRA; 3 CENPES/PETROBRÁS; 4 DEPARTAMENTO DE GENéTICA E EVOLUçãO – UFSCAR; 5 NOOSFERA PROJETOS ESPECIAIS.
RESUMO: A GFP é uma proteína com propriedades bioluminescentes isolada da A. victoria. Foram clonados para este estudo zíperes de leucina associados a proteínas fluorescentes, investigadas pelas técnicas de fluorescência e SAXS. Os resultados obtidos por SAXS sugerem que as proteínas fluorescentes estão na forma monomérica e as proteínas fluorescentes–zíper estão dimerizadas pela interação zíper–zíper. A caracterização biofísica das proteínas fluorescentes fusionadas ao zíper foi feita com a finalidade de fixar estas construções a um microambiente com placas dopadas com níquel monitorando em tempo real via proteínas fluorescentes. A tecnologia associada à fixação de proteínas fluorescentes fusionadas a zíper é a primeira etapa no planejamento e desenvolvimento de biossensores.
PALAVRAS CHAVES: proteína fluorescente, zíper de leucina, espalhamento de raios–x a baixos Ângulos (saxs).
INTRODUÇÃO: A A.victoria é uma medusa que quando estimulada produz numerosos pontos de luz devido à presença da green fluorescent protein (GFP) (SHIMOMURA et al., 1962; TSIEN, 1998). A GFP recombinante é amplamente utilizada como marcador biofluorescente (CHALFIE et al., 1994; GERDES and KAETHER, 1996). Mutações na seqüência primária da GFP permitiu o desenvovimento de proteínas fluorescentes com diferentes cores. A yellow fluorescent protein (YFP) foi desenvolvida a partir da mutação de quatro aminoácidosda na seqüência primaria da GFP (CLONTECH, 2001). Foram construídos plasmídios onde as proteínas fluorescentes estão fusionadas a zíperes de leucina. Estruturas diméricas coil-coil denominadas de zíperes são modelos simples definido pelo arranjo de sete aminoácidos que se repetem (GRUBER and LUPAS, 2003). A associação da GFP a zíperes de leucina produziu uma construção de interesse que pode ser aplicado tanto na investigação da interação proteína-proteína via zíperes complementares quanto na estabilidade e estequiometria da associação zíper-zíper e a predição da estrutura quaternária a partir do controle da interação entre essas construções (PINEIRO et al., 2005; MAGLIERY et al., 2005).
MATERIAL E MÉTODOS: Amostra: Foram utilizados os plasmidios moldes pEGFP-N1 e pEYFP-Mit para a GFP e YFP (CLONTECH, 2001). Na construção a cauda de histidina está na região N-terminal da proteína e o zíper no C-terminal. A purificação foi feita utilizando a resina de níquel seguida de troca iônica. As amostras foram preparadas em 50 mM Tris-HCl, pH 8,5.
Fluorescência. Os espectros de emissão de fluorescência foram feitas no comprimento de onda de excitação de 489 e 514 nm.
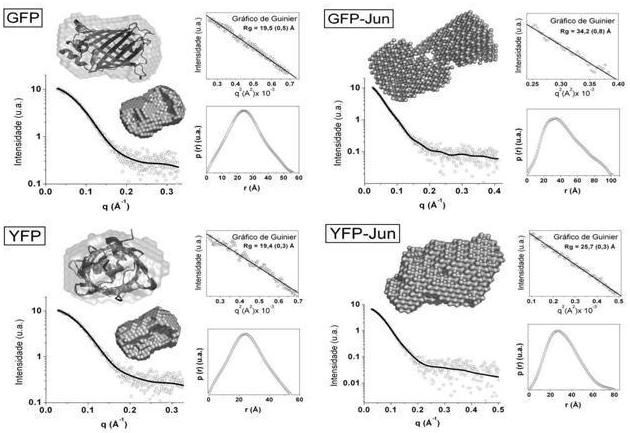
SAXS e Análise dos Dados. As medidas de SAXS foram coletadas no Laboratório Nacional de Luz Sincroton (LNLS, Campinas, SP), usando o detector 1D (KELLERMANN et al., 1997). As amostras (10 e 3 mg/ml) foram medidas (22 ºC) com o comprimento de onda de 1,488 Å e a distância amostra-detector de 648 mm cobrindo um intervalo de momento de transferência q entre 0,03-0,5 Å-1 ( ). As curvas de SAXS obtidas (exposição de 10 minutos) foram corrigidas pela homogeneidade do detector e normalizadas pelo decréscimo da intensidade do feixe incidente. Os dados de SAXS foram analisados com o programa GNOM (SVERGUN, 1992) e a forma molecular ab initio das proteínas em baixa resolução foram reconstruídas usando o programa DAMMIN (SVERGUN, 1999).
RESULTADOS E DISCUSSÃO: Segundo estudos, a adição da cauda de histidina ou da proteína de fusão GST (Glutathiona S Transferase) na região C-terminal da GFP não altera o espectro de fluorescência (PRASHER et al., 1992; ANGELIS et al., 1998). O espectro de fluorescência da GFP-JUN desloca para o mesmo comprimento de onda da YFP (527 nm) (CLONTECH, 2001).
A figura 1 representa os resultados dos experimentos de SAXS obtidos para as proteínas fluorescentes e as mesmas proteínas fusionadas ao zíper. Os resultados indicam que a GFP e a YFP estão na forma monomérica. Os dados dos experimentos GFP-JUN e YFP-JUN sugerem uma forma dimérica. Estruturas tridimensionais sugerem que a GFP pode ser encontrada tanto na forma monomérica (BREJC et al., 1997) quanto dimérica (YANG et al., 1996), dependendo das condições de cristalização. Uma das dúvidas decorrentes da construção, foi à possibilidade da dimerização ocorrer via superfície hidrofóbica da proteína fluorescente. Porém, conforme sugerem os resultados de SAXS, a forma dimérica foi decorrente da interação hidrofóbica entre os zíperes.
Figura 1: Curvas de SAXS das amostras: o raio de giro (Rg), a função distribuição p(r) e as formas dos modelos gerados.
CONCLUSÕES: Os resultados obtidos sugerem que na proteína fluorescente-zíper a dimerização é direcionada via contatos hidrofóbicos do zíper de leucina. Na GFP nativa a forma dimérica é estabilizada pelos contatos hidrofóbicos dos aminoácidos localizados na região C-terminal. Provavelmente, estes aminoácidos na construção proteína fluorescente-zíper estão inacessíveis espacialmente pela adição do zíper. A construção assume uma nova abordagem quando permite a imobilização via cauda de histidina num microambiente dopada com níquel, onde serão avaliadas as possibilidades do desenvolvimento de biossensores.
AGRADECIMENTOS:
REFERÊNCIAS BIBLIOGRÁFICA:ANGELIS, A.D.; MIESENBÖCK, G.; ZEMELNAN, V.B.; ROTHMAN, J. 1998. PRIM: Proximity
imaging of green fluorescent protein-tagged polypeptides. Protocolos National Academy Science USA, 95: 12312-12316.
BREJC, K.; SIXMA, T.K.; KITTS, P.A.; KAHN, S.R.; TSIEN, R.Y.; ORMÖ, M.; REMINGTON, S.J. 1997. Structural basis for dual excitation and photoisomerization of the Aequorea victoria green fluorescent protein. Protocolos National Academy Science USA, 94: 2306-2311.
CHALFIE, M.; TU, Y.; EUSKIRCHEN, G.; WARD, W.W.; PRASCHER, D.C. 1994. Green fluorescent protein as a marker for gene expression. Science, 263: 802-805.
CLONTECH - LIVING COLORS – User Manual Clontech Nov 2001. – www.clontech.com.
GERDES, H.H.; KAETHER, C. 1996. Green fluorescent protein: applications in cell biology. Febbs Letter, 24: 44 - 47.
GRUBER, M.; LUPAS, N. 2003. Historical review:another 50th anniversary – new periodicites in coiled coils. Trends Biochemical Science, 28: 679-685.
KELLERMANN, G.; VICENTINI, F.; TAMURA, E.; ROCHA, M.; TOLENTINO, H.; BARBOSA, A.; CRAIEVICH, A.F.; TORRIANI, I. 1997. The small-angle X-ray scattering beamline of the Brazilian synchrotron light laboratory. J Appl Cryst, 30: 880-883.
MAGLIERY, J.T.; WILSON, C.G.M.; PAN, W.; MISHLER, D.; GHOSH, I.; HAMILTON, A.D.; REGAN, L. 2005 Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. Journal American Chemical Society, 127:146-157.
PINEIRO, A.; VILLA, A.; VAGHT, T.; KOKSCH, B.; MARK A.E. 2005. A Molecular dynamics study of the formation, stability, and oligomerization state of two designed coiled coils: possibilities and limitations. Biophysical Journal, 89: 3701-3713.
PRASHER, D.C.; ECKENRODE, V.K.; WARD, W.W.; PRENDERGAST, F.G.; CORMIER, M.J. 1992. Primary structure of the Aequorea victoria green-fluorescent protein. Gene, 111: 229-233.
SHIMOMURA, O.; JOHNSON, F.H.; SAIGA, Y. 1962. Extraction, purification and properties of aequorin, a bioluminescent protein from the luminous hydromedusan Aequorea. Journal Cellular Compendium Physiology, 59: 223-239.
SVERGUN, D.I. 1992. Determination of the regularization parameter in indirect-transform methods using perceptual criteria. J Appl Cryst, 25: 495-503.
SVERGUN, D.I. 1999. Restoring low resolution structure of biological macromolecules from solution scattering using simulated annealing. Biophysical J., 76: 2879–2886.
TSIEN, Y.R. 1998. The green fluorescent protein. Annual Reviews Biochemistry, 67: 509-544.
YANG, F.; MOSS, L.G.; PHILLIPD G.N. JR. 1996. The molecular structure of green fluorescent protein. Nature Biotechnology, 14: 1246-1251.
