ÁREA: Bioquímica e Biotecnologia
TÍTULO: Docking molecular de compostos organocalcogênios anti-inflamatórios na enzima COX-2
AUTORES: Baptisitni, N. (UFSCAR) ; Caracelli, I. (UFSCAR) ; Zukerman-schpector, J. (UFSCAR) ; Olivato, P.R. (USP) ; Cerqueira Jr., C.R. (USP)
RESUMO: A enzima ciclooxigenase-2 (COX-2) tem sido relacionada a estímulos que resultam em
processos inflamatórios. A inibição desta enzima pode causar alívio aos sintomas
da inflamação e da dor1-4. A partir de 1999, a indústria farmacêutica mundial
começou a oferecer inibidores de COX-2 como se fossem um fármaco milagroso, capaz
de aliviar a dor sem causar sangramento no estômago, por não inibir também a
isoforma COX-1, presente nos tecidos saudáveis. Assim, fármacos foram aprovados
rapidamente, mas alguns causaram sérios efeitos colaterais, inclusive ataques
cardíacos ou derrames5, por isso a busca por melhores inibidores ainda continua.
PALAVRAS CHAVES: Docking; ciclooxigenase; organocalcogênios
INTRODUÇÃO: No BioMat - Grupo de Física Teórica de Materiais e Biomoléculas – Departamento
de Física da UFSCar, novos inibidores da enzima COX-2 tem sido objeto de
estudo6. Para os estudos são utilizados experimentos in silico, que tem como um
dos objetivos buscar inibidores seletivos de COX-2 e não de COX-1. Os estudos
buscam a formação de complexos enzima-COX-2-inibidores, onde o complexo putativo
formado é monitorado utilizando a visualização gráfica e é feita uma análise
minuciosa das interações intermoleculares entre a enzima e o inibidor.
No BioMat também tem sido estudadas pequenas moléculas da família dos
organocalcogênios, com uma visão estrutural e da química supramolecular e também
a presença desses tipo de compostos em moléculas biológicas7-9. Uma série de
compostos organocalcogênios foi sintetizada e alguns deles já tiveram sua
estrutura tridimensional determinada por difração de raios X10 e foram objeto de
estudo neste trabalho.
MATERIAL E MÉTODOS: Os 15 compostos organocalcogênios e seus enantiômeros foram modelados, utilizando
o programa HyperChem.11 Os compostos estudados são apresentados na Figura 1, com
um esqueleto central e substituintes X = S, SO e SO2; Y = Br, H, CH3, OCH3 e NO2.
A estrutura tridimensional da enzima COX-23a,12 foi obtida no PDB (Protein Data
Bank).12 Os cálculos de docking molecular foram feitos com o programa GOLD
(Genetic Optimization Ligand Docking).13 O escore utilizado foi o GoldScore e
flexibilização total dos ligantes. Os resultados foram analisados e avaliados o
padrão de orientação e/ou conformação (pose), o escore, as interações dos ligantes
com os aminoácidos da enzima. Para essa análise foi utilizando o programa DS
Visualizer 3.014.
RESULTADOS E DISCUSSÃO: Os cálculos de docking foram realizados com os 15 compostos R e seus
enantiômeros S. A seleção das poses (orientação/conformação) dos ligantes foi
feita utilizando a visualização molecular. Os resultados indicaram uma
preferência pelos confôrmeros S, como por ser observado na Figura 1. Os
resultados para os ligantes S estão representados na cor verde enquanto os R
estão na cor azul. Em violeta é apresentada a diferença dos escores entre
enantiômeros, com um máximo de 4,5 kcal/mol. Entre os compostos estudados, os
que apresentaram melhores resultados são os que possuem Y = OCH3 e com a
seguinte ordem para X, S < SO < SO2. Na Figura 2 podem ser observados o sítio
ativo de COX-2, onde é possível ver uma parte do grupo heme, cofator desta
enzima, os aminoácidos que interagem com o ligante cristalográfico, apresentado
com os átomos de C em magenta, e o melhor ligante da série estudada com os
átomos de C em azul. A superfície de van der Waals relativa ao ligante
cristalográfico, é adequada também para o ligante calcogênico.
Figura 1
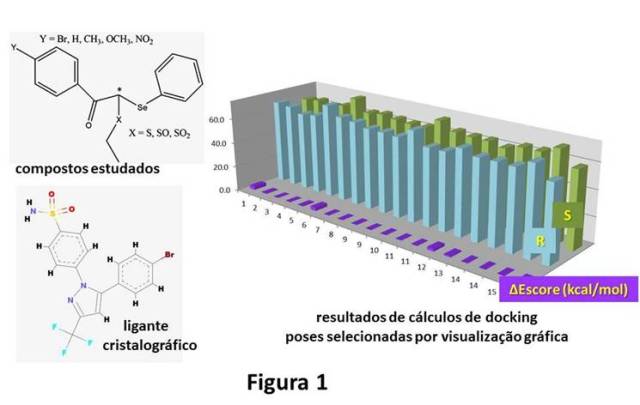
Representação dos compostos estudados e escores
associados a formação de complexo enzima-inibidores.
Figura 2
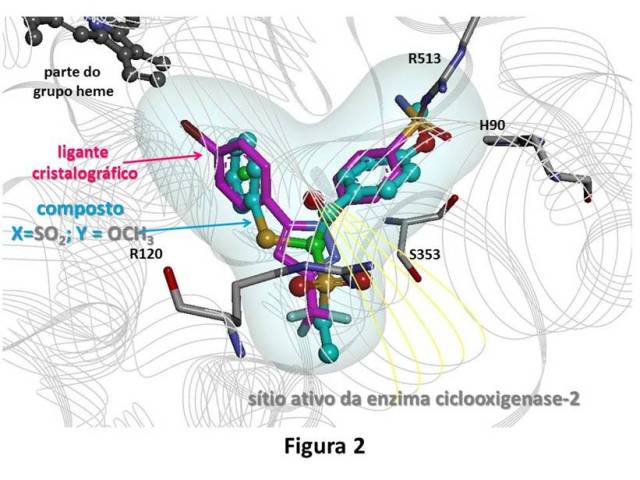
Representação do melhor ligante da série estudada e
o ligante cristalográfico da enzima COX-2 no sítio
ativo dessa enzima.
CONCLUSÕES: A série de compostos estudada apresentou pequena preferência em favor da
conformação S. O ligante cristalográfico e as moléculas estudadas apresentam
motivos similares: as duas têm anéis fenila, e na posição onde o ligante
cristalográfico apresenta o anel triazol, os ligantes apresentam o carbono
assimétrico. Assim, esta série de compostos apresenta potencial para inibir a COX-
2. Os estudos continuam com os objetivos de determinar com experimentos in sílico
o modo de ligação à enzima correlata COX-1. Também serão realizados testes de
atividade inibitória destes composto na enzima COX-2.
AGRADECIMENTOS: Agradecimentos: FAPESP (N.B.:2011/08332-7); CNPq (IC: 308116/2010-0; J.Z-S:
306532/2009-3) e CAPES (J. Z.-S. e I. C.Rede Nanobiotec-Brasil 808/2009)
REFERÊNCIAS BIBLIOGRÁFICA: 1Kim, J-B, Han, A-R., Park, E-Y, Kim, J-Y, Cho, W., Lee, J., Seo, E-K and Lee, K-T. Inhibition of LPS-induced iNOS, COX-2 and cytokines expression by poncirin through the NF-kappaB inactivation in RAW 264.7 macrophage cells. Biological and Pharmaceutical Bulletin, 30, 2345—2351, 2007.
2Bazan, N. G. COX-2 as a multifunctional neuronal modulator.Nat. Medicine,v.7,p.414-415, 2001.
3 (a) Kurumbail R.G.; Stevens A.M.; Gierse J.K. Structural basis for selective inhibition of cyclooxygenase-2 by anti-inflammatory agents. Nature, 384, 644-648, 1996. (b) Rimon, G.; Sidhu, R.S.; Lauver, D.A.; Lee, J.Y.; Sharma, N.P.; Yuan, C.; Frieler, R.A..; Trievel, R.C.; Lucchesi, B.R.; Smith, W.L. Coxibs interfere with the action of aspirin by binding tightly to one monomer of cyclooxygenase-1. Proc. Natl. Acad. Sci. USA. 107, 28-33, 2010.
4Fitzgerald G.A.; Patrono C. N. The coxibs, selective inhibitors of cyclooxygenase-2. Engl. J. Med. 345, 433-442, 2001.
5Catizone, C. Unraveling the Cox-2 Debacle. Disponível em <www.nextgenmd.org/archives/196, 2011>. Acesso em Mai. 2012.
6Stefani, H. A. ; Botteselle, G. V. ; Zukerman-schpector, J. ; Caracelli, I. ; da Silva Corrêa, D.; Farsky, S.H.P.; Machado, I.D. ; Santin, J.R. ; Hebeda, C. B. Synthesis, anti-inflammatory activity and molecular docking studies of 2,5-diarylfuran amino acid derivatives. Eur. J. Med. Chem., 47, 52-58, 2012.
7Caracelli, I.; Zukerman-schpector, J. ; Tiekink, E.R.T. Supramolecular aggregation patterns based on the bio-inspired Se(lone pair) (aryl) synthon. Coord. Chem. Rev., 256, 412-438, 2012.
8Caracelli, Ignez ; Vega-Teijido, Mauricio ; Zukerman-Schpector, Julio ; Cezari, Maria H.S. ; Lopes, José G.S. ; Juliano, Luiz ; Santos, Paulo S. ; Comasseto, João V. ; Cunha, Rodrigo L.O.R. ; Tiekink, Edward R.T. . A tellurium-based cathepsin B inhibitor: Molecular structure, modelling, molecular docking and biological evaluation. J. Mol. Structure, 1013, 11-18, 2012.
9Caracelli, I. ; Zukerman-Schpector, J. ; Maganhi, S. H. ; Stefani, H.A. ; Guadagnin, R.; Tiekink, E.R.T . 2-chlorovinyl tellurium dihalides, (p-tol)Te[C(H)=C(Cl)Ph]X2 for X = Cl, Br and I: variable coordination environments, supramolecular structures and docking studies in cathepsin B. J. Braz. Chem. Soc. 21, 2155-2163, 2010.
10Zukerman-Schpector, J. ; De Simone, C. A. ; Olivato, P. R. ; Cerqueira, C. R. ; Tiekink, E. R. T. 1-(4-Bromophenyl)-2-ethylsulfinyl-2-(phenylselanyl)ethanone monohydrate. Acta Cryst. E67, o1099-o1100, 2011.
.11HyperChem® 5.01 Computional Chemistry, Waterloo, Ont. N2L 3X2, Canada, Hypercube Inc., 1996.
12PDB - Protein Data Bank: http://www.rcsb.org/pdb/home/home.do; pdb codes 1cx2, 3kk6.
13Jones, G.; Willett, P; Glen, R. C., Leach, A. R., Taylor, R., Development and validation of a genetic algorithm for flexible docking. J. Mol. Biol., 267, 727-748, 1997.
14DsVisualizerTM-Accelrys Software Inc.Discovery Studio Visualizer 3.0.
