ÁREA: Iniciação Científica
TÍTULO: EXTRAÇÃO DE DNA DE BACTÉRIAS DO GÊNERO AZOSPIRILLUM EM MICROCHIPS DE POLIÉSTER-TONER (PT)
AUTORES: Ferreira, L.R. (UNIVERSIDADE ESTADUAL DE GOIÁS) ; Oliveira, F.A. (UNIVERSIDADE ESTADUAL DE GOIÁS) ; Didonet, C.C.G.M. (UNIVERSIDADE ESTADUAL DE GOIÁS) ; Duarte, G.R.M. (UNIVERSIDADE ESTADUAL DE GOIÁS)
RESUMO: Sistemas microfluídicos tornaram-se uma ferramenta analítica poderosa para
análise de DNA tanto para diagnóstico clínico quanto para análise forense. As
plataformas microfluídicas apresentam a oportunidade de redução do tempo e custo
necessário para realizar todas as etapas em dispositivos microfluídicos, redução
da quantidade de amostra necessária para análise e automatização da extração
reduzindo o contato manual com a amostra. O método de extração de DNA utilizado
neste trabalho, chamado de SPE dinâmica (dSPE) utiliza uma “fase sólida móvel”
manipulada através de campo magnético. A dSPE em microchip de PT apresentou
notável rapidez (extração em cerca de 10 minutos) e um ótimo perfil de eluição,
além de proporcionar uma eficiência de extração de 77,3% e frações altamente
concentradas.
PALAVRAS CHAVES: extração; DNA; microchip
INTRODUÇÃO: Atualmente, sistemas microfluídicos se tornaram uma ferramenta analítica
poderosa para análise de DNA tanto para diagnóstico clínico quanto para análise
forense. A extração (BIENVENUE et al., 2006), amplificação (HÜHMER et al., 2000)
e separação (WOOLLEY et al., 1994) de DNA já são realizadas em plataformas
miniaturizadas.
Sistemas microfluídicos integrados apresentam a oportunidade de automatização da
manipulação das soluções, reduzindo o contato manual com a amostra. Além da
redução do tempo e custo necessário para realizar todas as etapas em
dispositivos microfluídicos, a redução da quantidade de amostra necessária para
análise é uma das grandes vantagens, inerente ao uso de plataformas
miniaturizadas.
O novo método de extração de DNA utilizado neste trabalho, chamado de SPE
dinâmica, ou no inglês “dynamic SPE” (dSPE), utiliza o mesmo princípio da
adsorção de DNA em sílica, porém utiliza uma fase sólida dinâmica (móvel)
manipulada através de um campo magnético ao invés de uma fase sólida estática
como nos métodos de extração em microchip atualmente utilizados. Para isso,
utilizam-se partículas magnéticas de sílica, disponíveis comercialmente como
MagneSil. A aplicação do campo magnético faz com que as partículas magnéticas
possam se movimentar dentro do canal microfluídico.
Na busca por materiais alternativos e métodos mais simples de microfabricação,
em 2003 Lago e colaboradores descreveram outro processo, ainda mais simples e
mais barato, para produção de dispositivos microfluídicos (DO LAGO et al.,
2003). Este método descrito por Lago utiliza uma camada de toner depositada em
filme de poliéster (transparência) para definir os canais microfluídicos, além
de servir como adesivo para vedação do dispositivo através de uma etapa de
laminação a quente.
MATERIAL E MÉTODOS: Dois filmes de poliéster recobertos com toner e com os microcanais recortados
foram usados como partes intermediárias nos microchips. Para base e o topo do
microchip foram utilizados apenas o filme de poliéster, sendo o filme do topo
perfurado com furos de 2 mm do microchip antes da etapa de laminação para
servirem de acesso aos reservatórios. Os quatro filmes de poliéster foram
alinhados e laminados juntos em uma laminadora.
As amostras lisadas de cultura de bactérias do gênero Azospirillum foram
preparadas misturando 100 μL da amostra da cultura de bactérias com 5 μL de
proteinase K (20 mg/mL) e 9 μL de GuHCl 8M ( pH = 7,6) + 1% triton X-100,
totalizando 114 μL de solução. A mistura foi agitada em vortex e incubada em
banho-maria a 56 °C por 10 min. Uma alíquota de 2 μL da amostra foi utilizada na
extração.
Para realizar a extração de DNA, preencheu-se o canal do microchip com 4 µL de
GuHCl 8M ( pH = 7,6). Adicionou-se 2 µL de Magnesil e 2 µL da amostra lisada da
cultura de bactérias do gênero Azospirillum. Manteve-se a solução presente no
canal microfuídico do microchip em agitação por 5 minutos. Lavou-se a solução
presente no microchip com 4 µL de isopropanol 80% sob agitação e, em seguida,
lavou-se com 2 µL de isopropanol 80% sob agitação. Lavou-se com 2 µL de solução
tampão TE (0,1x) por seis vezes sem agitação. Agitou-se a solução no canal
microfluídico por 5 minutos. Iniciou-se a extração do DNA, colocou-se 2 µL de TE
(0,1 x) no reservatório esquerdo no canal microfuídico e no reservatório direito
coletou-se 2 µL de solução que foi posta em um eppendorfe. Colocou-se a solução
presente no canal microfuídico em agitação por 2 minutos e repetiu-se este
processo de extração por doze vezes, obtendo-se treze frações. Em cada fração
adicionou-se 18 µL de TE (0,1 x).
RESULTADOS E DISCUSSÃO: A extração dinâmica de DNA em fase sólida em microchip de PT ocorreu em cerca de
10 minutos e apresentou um excelente perfil de eluição (Figura 1), visto que
cerca de 25% do total de DNA eluído foi extraído na primeira fracão após o
início do movimento da sétima fração (14 μL) e cerca de 40% do total de DNA
extraído foram eluídos nas três primeiras frações após o início do movimento da
fase sólida.
As duas primeiras frações coletadas foram majoritariamente isopropanol 80%,
proveniente das lavagens e as quatro frações seguintes foram coletadas a partir
das lavagens com 0,1× TE sem o movimento das partículas. A sétima fração (14 μL)
corresponde à primeira fração de DNA eluído após o início o movimento das
partículas para a dessorção das moléculas de DNA da fase sólida. A extração
dinâmica de DNA em fase sólida em microchip de PT proporcionou uma eficiência de
extração de 77,3% e frações altamente concentradas.
Figura 1
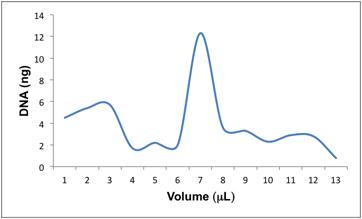
Perfil de eluição de uma extração de DNA a partir de
uma amostra de cultura de bactérias do gênero
Azospirillum.
CONCLUSÕES: A extração dinâmica de DNA em fase sólida em microchip de PT mostrou ser um
excelente método para extração de DNA combinando diversas vantagens, como rapidez
(extração em cerca de 10 minutos) e um excelente perfil de eluição. Além de,
verificar-se um método altamente eficiente para a extração de DNA a partir de uma
amostra de bactérias, proporcionando uma eficiência de extração de 77,3% e frações
altamente concentradas.
AGRADECIMENTOS:
REFERÊNCIAS BIBLIOGRÁFICA: BIENVENUE, J. M.; DUNCALF, N.; MARCHIARULLO, D.; FERRANCE, J. P.; LANDERS, J. P. Microchip-based cell lysis and DNA extraction from sperm cells for application to forensic analysis. Journal of Forensic Sciences, v. 51, n. 2, p. 266-273, 2006.
DO LAGO, C. L.; SILVA, H. D. T.; NEVES, C. A.; BRITO-NETO, J. G. A.; SILVA, J. A. F. A dry process for production devices base on the lamination of laser-printed polyester films. Analytical Chemistry, v. 75, n. 15, p. 3853-3858, 2003.
HÜHMER, A.; LANDERS, J. P. Noncontact infrared-mediated thermocycling for effective polymerase chain reaction amplification of DNA in nanoliter volumes. Analytical Chemistry, v. 72, n. 21, p. 5507-5512, 2000.
WOOLLEY, A. T.; MATHIES, R. A. Ultra-high-speed DNA fragment separations using microfabricated capillary array electrophoresis chips. Proceedings of the National Academy of Sciences, v. 91, p. 11348-11352, 1994.
