Realizado no Rio de Janeiro/RJ, de 14 a 18 de Outubro de 2013.
ISBN: 978-85-85905-06-4
ÁREA: Iniciação Científica
TÍTULO: Planejamento racional de fármacos pertencentes à classe das cefalosporinas
AUTORES: Henriques, S.V.C. (UNIFAP) ; Picanço, L.C.S. (UNIFAP) ; Medeiros, A.F. (UNIFAP) ; Alves, C.M. (UNIFAP) ; da Silva, J.O. (UNIFAP) ; Santos, C.B.R. (UNIFAP) ; Hage-melim, L.I.S. (UNIFAP)
RESUMO: O aumento da resistência bacteriana a vários agentes antimicrobianos acarreta em dificuldades no manejo de infecções e contribui para o aumento de custos do sistema de saúde e dos próprios hospitais. O objetivo deste estudo foi determinar os possíveis locais de ligação de 25 inibidores da enzima β-lactamase através de docking molecular. Foi observado que 23 inibidores se ligaram na mesma região de um composto que teve sua estrutura resolvida experimentalmente. A enzima β-lactamase foi submetida ao servidor Q-SiteFinder com a finalidade de se obter possíveis locais de ligação de inibidores. Os locais preditos coincidiram com o sítio de ligação do inibidor determinado experimentalmente. Os resultados mostraram que 23 inibidores são potenciais candidatos a fármacos antimicrobianos.
PALAVRAS CHAVES: Cefalosporinas; Antimicrobianos; Novos fármacos
INTRODUÇÃO: Nos países em desenvolvimento, poucos recursos são empregados na monitorização de ações sobre o uso racional de antibióticos o que resulta em um uso desenfreado de antibióticos sem uma cuidadosa avaliação das suas indicações apropriadas, o que pode levar ao crescimento de cepas resistentes, ou seja, acarretar em uma mutação seletiva (NICOLLINI et al., 2008).
Cefalosporinas e penicilinas são os antibióticos mais amplamente utilizados para o tratamento de doenças infecciosas, sendo as duas principais classes de antibióticos β-lactâmicos. Cada um possui um anel β-lactâmico, porém o anel tiazolidínico da penicilina é substituído por um anel dihidrotianínico nas cefalosporinas (ROMANO et al., 2010). As enzimas transpeptidases possuem um sítio ativo de serina e realizam seu ciclo catalítico por uma via de acilação e desacilação. Os antibióticos β-lactâmicos são capazes de inibir eficientemente essas enzimas, devido à semelhança estereoquímica entre a unidade β-lactâmica com os resíduos de D-alanina do substrato. Na presença do antibiótico, as transpeptidases formam um complexo covalente enzima-peniciloil que bloqueia a reação normal de transpeptidação. Esta ação resulta na formação de um peptideoglicano fracamente reticulado, o que faz com que as bactérias cresçam altamente suscetíveis à lise e morte (WILKE et al., 2005).
As enzimas β-lactamases são enzimas que conferem uma significante resistência aos antibióticos β-lactâmicos, ao hidrolisar a ligação amida do anel β-lactâmicos de 4 membros. Esta reação envolve a criação de uma serina nucleófila através da desprotonação do sítio ativo serina com uma base, através de um ataque nucleofílico ao anel β-lactâmico, formando um complexo acil-enzima intermediário que é hidrolisado por uma base ativada por água (WILKE et al., 2005).
MATERIAL E MÉTODOS: Em modelagem molecular, “docking” é o método que define a orientação preferencial de uma molécula com respeito à outra na formação de complexos estáveis, por exemplo, enzima-inibidor ou proteína-ligante. O conhecimento da orientação preferencial, por sua vez, pode ser utilizado para prever a afinidade de ligação entre as moléculas, usando, para isso, funções “score” apropriadamente desenvolvidas para esse fim (LENGAUER; RAREY, 1996). A partir das estruturas otimizadas dos inibidores relatados na literatura, simulações de docking foram realizadas entre esses ligantes e a β-lactamase com o programa PatchDock (SCHNEIDMAN-DUHOVNY et al., 2003) disponível em http://bioinfo3d.cs.tau.ac.il/PatchDock/.
A identificação da localização de um sítio de ligação numa proteína é de fundamental importância durante o planejamento de novos fármacos e, em particular, para a realização de “docking” molecular, para esta finalidade foi utilizado o web-servidor Q-SiteFinder. O Q-SiteFinder (LAURIE; JACKSON, 2005) é um método que prediz sítios de ligação favoráveis utilizando um critério puramente de energia. Esse método calcula as interações de energias de van de Waals de um grupo de prova metila com a proteína. As provas com energia de interação favoráveis são retidas e os mapas destas provas as são classificados de acordo com suas energias totais de interação. O Web-servidor encontra-se disponível em http://www.modelling.leeds.ac.uk/qsitefinder/.
RESULTADOS E DISCUSSÃO: A enzima β-lactamase está representada de acordo com sua estrutura secundária e os inibidores estão representados em formato stick entre os aminoácidos da enzima. As análises dos modos de ligação de 25 inibidores mostraram que 23 inibidores apresentaram-se semelhantes ao inibidor experimental. Foi utilizado o método de docking que envolve uma função de energia contendo parâmetros eletrostáticos, de van der Waals, de pontes de hidrogênio e, algumas vezes, hidrofóbicos, a qual gera modelos matemáticos que predizem as melhores orientações do ligante, segundo uma lista de escores de energia. Essa avaliação da interação entre o ligante e o receptor em termos quantitativos é importante para o entendimento da especificidade do receptor por um determinado ligante.
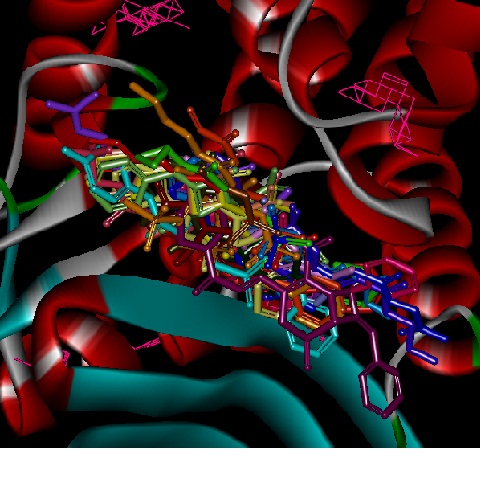
Além de conhecer o modo de ligação dos inibidores, foi identificada a localização dos sítios de ligação da enzima β-lactamase através do web-servidor Q-SiteFinder, o que é importante para o planejamento de novos candidatos a fármacos. Todos os 23 inibidores tiveram seu modo de ligação predito em sítios de ligação determinados pelo Q-SiteFinder.
Figura 1. Modos de ligação propostos por simulações de "docking" para
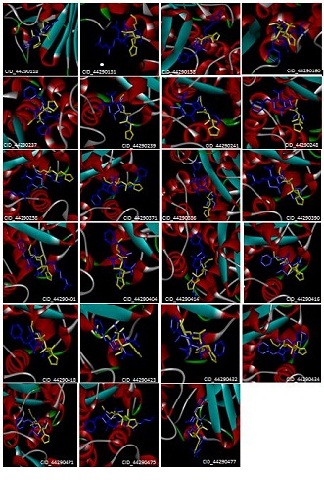
Figura 2. Sítios de ligação preditos pelo Q-SiteFinder e os 23 inibido
CONCLUSÕES: As enzimas β-lactamases conferem resistência aos antibióticos β-lactâmicos. Os resultados da análise demonstraram que 23, dos 25 inibidores de β-lactamases, apresentaram semelhanças quando comparados ao inibidor experimental, em relação à ligação com os sítios ativos da enzima. Com estes resultados, conclui-se que os 23 inibidores podem ser potenciais candidatos a fármacos, e vir a auxiliar no combate à resistência crescente das cepas à enzima, e prolongar a ação dos antibióticos.
AGRADECIMENTOS:
REFERÊNCIAS BIBLIOGRÁFICA: LAURIE, A. T.; JACKSON, R.M. Q-SiteFinder: an energy-based method for the prediction of protein-ligand binding sites. Bioinformatics, V. 21, nº 9, p. 1908-1916, 2005.
LENGAUER, T.; RAREY, M. Computational methods for biomolecular docking. Current Opinion in Structural Biology, V. 6, nº 3, p. 402-406, 1996.
NICOLLINI, P.; NASCIMENTO, J. W. L.; GRECO, K.V., DE MENEZES, F. G.Fatores relacionados à prescrição médica de antibióticosem farmácia pública da região Oeste da cidade de São Paulo.Ciência& Saúde Coletiva, v.13(Sup), 689:696, 2008.
ROMANO, A. et al. IgE-mediated hypersensitivity to cephalosporins: Cross-reactivity and tolerability of penicillins, monobactams, and carbapenems. J. Allergy Clin. Immunol. v. 126, n. 5, p. 994-999, 2010.
SCHNEIDMAN-DUHOVNY D, INBAR Y, POLAK V, SHATSKY M, HALPERIN I, BENYAMINI H, BARZILAI A, DROR O, HASPEL N, NUSSINOV R, WOLFSON HJ. Taking geometry to its edge: fast unbound rigid (and hinge-bent) docking. Proteins. 2003 Jul 1; 52(1): 107-12.
WILKE, M. S. et al. β-Lactam antibiotic resistance: a current structural perspective. Curr. Opin. Microbiol. v. 8, p. 525-533, 2005.
