ANÁLISE COMPUTACIONAL DE POTENCIAIS INIBIDORES DAS ENZIMAS QUE UTILIZAM O SUBSTRATO FOSFOENOLPIRUVATO
ISBN 978-85-85905-23-1
Área
Bioquímica e Biotecnologia
Autores
Oliveira, M.V.D. (UFPA) ; Gonçalves, A.F. (UFPA) ; Araújo, J.O. (UFPA) ; Lameira, J. (UFPA) ; Lima, A.H. (UFPA)
Resumo
O substrato fosfoenolpiruvato (PEP) é utilizado em diversos processos de interesse biológico. O objetivo deste trabalho é estudar os análogos do substrato PEP através de métodos de modelagem molecular para selecionar moléculas que façam uma boa interação no sítio ativo das enzimas KDO8PS, DAHPS, EPSPS e MurA. Utilizando técnicas computacionais, foi determinada a energia de interação entre cada derivado e as enzimas alvo. Os resultados apontam que um inibidor já descrito na literatura com potencial ação contra a enzima KDO8P sintase tem melhor energia de interação com a enzima EPSP sintase, a qual é alvo de herbicidas já consolidados no mercado.
Palavras chaves
docking molecular; fosfoenolpiruvato; inibidores enzimáticos
Introdução
As principais informações sobre a função e estrutura de proteínas e o desenho de fármacos com base na estrutura podem ser fornecidas pelo entendimento de como a catálise está ocorrendo no sítio ativo da enzima (ANDERSON, 2005). Neste sentido, busca-se conhecer não somente os substratos que são utilizados na reação, mas também os intermediários e principalmente os análogos que podem vir a ser utilizados como inibidores deste processo. Como exemplo, o Fosfoenolpiruvato (PEP) é uma molécula altamente funcionalizada, quimicamente versátil que é usada como substrato em várias etapas bioquímicas fundamentais no metabolismo energético celular e em biossínteses (WALSH et al., 1997). A enzima 5-enolpiruvoyl-chiquimato-3-fosfato (EPSP) sintase, é uma enzima da via do chiquimato envolvida na biossíntese de ácidos aromáticos (STEINRUCKE; NAMRHEIN, 1980) enquanto a UDPGlcNAcenolpyruvoyltransferase (MurA), uma enzima envolvida na biossíntese de peptidoglicano (KAHAN et al., 1974). Ambas envolvem o PEP em seu processo catalítico e são alvos de moléculas com potencial herbicida (Glifosato) e antibiótico (Fosfomicina). Além disso a enzima 3-desoxi-D-manno-2-octulosonato-8-fosfato (KDO8P) sintase, catalisa a formação de KDO8P, um açúcar de 8 carbonos, a partir do açúcar de 5 carbonos, D-arabinose-5-fosfato (A5P), e PEP e a enzima DAHP sintase, assim como a EPSP sintase, é encontrada na via do chiquimato para a biossíntese de aminoácidos aromáticos essenciais. Diante disso, este trabalho objetiva utilizar métodos de modelagem molecular no sentido de analisar diferentes derivados do substrato PEP a fim de encontrar moléculas que tenham afinidade com as enzimas supracitadas e identificar moléculas com potencial interesse biológico.
Material e métodos
Inicialmente foi realizado um levantamento na literatura no intuito de selecionar alguns análogos ao Fosfoenolpiruvato (PEP) e enzimas que utilizam o PEP como substrato natural. Neste estudo, foram utilizadas as estruturas cristalinas das enzimas DAHP sintase, EPSP sintase, MurA transferase, KDO8P sintase, cujo os códigos depositados no banco de dados de proteínas PDB (do inglês, Protein Data Bank) são respectivamente: 5HUC, 2O0E, 3UPK, 1FXQ. A estrutura química de cada molécula análoga ao substrato PEP foi desenhada no programa MarvinSketch. Em seguida, as estruturas foram otimizadas com o método HF/6-311G* no programa Gaussian09. Com o auxílio do pacote de programas Molegro Virtual Docker foram realizadas simulações de acoplamento molecular (do inglês, molecular docking) para análise da formação de complexos enzima-ligante das estruturas cristalográficas das enzimas que utilizam PEP com análogos do PEP. Essas simulações foram realizadas avaliando-se a energia da função de pontuação (E_score) fornecida pelo algoritmo Moldock score, cujo valor é obtido a partir da equação E_score=E_inter+E_intra, em que E_inter representa a energia de interação intermolecular (proteína-ligante) e E_intra, a energia intramolecular (ligante). Por fim, uma cuidadosa análise in silico das propriedades como distância de interação ligante-receptor e energia de interação foram feitas com o intuito de descrever o comportamento dos análogos do substrato PEP no sítio ativo da enzima.
Resultado e discussão
Após análise de dados da literatura que apontavam derivados do substrato PEP
como potenciais inibidores das enzimas DAHPS, EPSPS, MurA e KDO8PS, foram
selecionados 26 análogos que implicaram em 104 simulações de docking
molecular a fim de selecionar qual seria o análogo com maior potencial de
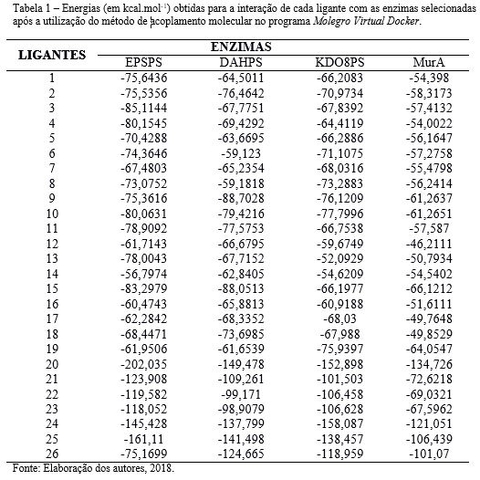
interação com estas enzimas. Neste sentido, a Tabela 1 apresenta os
resultados de energia obtidos com o algoritmo MolDock score e apresenta o
ligante 20 com o maior potencial de interação com a enzima EPSPS e se
destaca entre as melhores interações com as quatro enzimas estudadas.
Além disso, os ligantes 24 e 25 mostraram-se com um bom potencial de
interação com estas enzimas. Quanto a esta observação, pode-se destacar o
estudo feito por HARRISON; REICHAU; PARKER (2012) que analisa os ligantes
21, 22, 23, 24, 25 e 26 mostra o ligante 24 como o melhor inibidor da enzima
KDO8PS de Neisseria meningitidis(Ki 7,9 ± 1,6 μM). O ligante 20, com
constante de inibição contra KDO8PS de Escherichia coli de 0,37 μM, é
apontado por Du e colaboradores (1999) como o mais efetivo inibidor dessa
enzima já descrito na literatura.
As simulações realizadas com os análogos de PEP não só corroboram com os
trabalhos supracitados como também sugerem que estes inibidores podem ter
possibilidades de inibir outras enzimas que utilizam o PEP, como o caso da
enzima EPSPS que apresentou E_score= -202,035 kcal.〖mol〗^(-1). A Figura 1
aponta que esta afinidade com o sítio ativo da enzima EPSP sintase se dá
pelo grande número de interações de hidrogênio realizadas.
Conclusões
O estudo das principais interações entre os análogos do substrato PEP e as enzimas apontam o ligante 20 com potencial de interação não somente com a enzima KDO8PS, mas principalmente com a enzima EPSPS. Segundo estes resultados, o ligante 20 mostra-se como o melhor possível inibidor uma vez que possui uma energia melhor em relação aos outros. Desta forma, espera-se contribuir fornecendo dados que possam dar suporte ao planejamento de fármacos e de moléculas com interesse biotecnológico.
Agradecimentos
À Universidade Federal do Pará e ao Laboratório de Planejamento e Desenvolvimento de Fármacos pelo suporte à realização desta pesquisa.
Referências
ANDERSON, K.S. Detection of novel enzyme intermediates in PEP-utilizing enzymes. Archives of Biochemistry and Biophysics, Vol. 433, Ed. 1, p. 47-58, january, 2005.
DU, S.; FAIGER, H.; BELAKHOV, V.; BAASOV, T.Towards the Development of Novel Antibiotics: Synthesis and Evaluation of a Mechanism-Based Inhibitor of Kdo8P Synthase. Bioorganic & Medicinal Chemistry, Vol. 7, Ed. 12, p. 2671-2682, may, 1999.
HARRISON, A. N.; REICHAU, S.; PARKER, E. J. Synthesis and evaluation of tetrahedral intermediate mimic inhibitors of 3-deoxy-D-manno-octulosonate 8-phosphate synthase. Bioorganic and Medicinal Chemistry Letters, Vol. 22, Ed. 2, p. 907–911, 2012.
KAHAN, F.M.; KAHAN, J.S.; CASSIDY, P. J.; KROPP, H., The mechanism of action of fofomycin (phosphonomycin). The New York Academy of Sciences, Vol. 235, Ed. 1, p. 364-386, may, 1974.
STEINRUCKEN, H.C.; AMRHEIN, N., Herbicide glyphosate is a potent inhibitor of 5-enolpyruvyl-shikimic acid-3-phosphate synthase. Biochemical and Biophysical Research communications, Vol. 94, Ed. 4, p. 1207-1212, june, 1980.
WALSH, C. T.; DRUECKHAMMER, D. G.;VOGEL, K. W.; LAMBALOT, R. H.; GEHRING, A. M., Ability of Streptomyces spp. aryl carrier proteins and coenzyme A analogs to serve as substrates in vitro for E. coli holo-ACP synthase. Chemistry & Biology, Vol. 4, Ed. 1, p. 17-24, January, 1997.











