ESTUDO COMPARATIVO DE DOCKING MOLECULAR ENTRE O COMPLEXO METÁLICO ZINCO COM MICRORGANISMOS BACTERIANOS
ISBN 978-85-85905-25-5
Área
Bioquímica e Biotecnologia
Autores
Silva, W.F. (UFMA) ; Bastos, R.S. (UFMA-CIENATEC) ; Faria, M.B. (UFMA) ; Freitas, T.M.S. (UEMA) ; Ribeiro, F.V.S. (IFPA) ; Sousa, M.B. (UFMA) ; Barbosa, E.S. (UFPA) ; Silva, S.T.P. (IFPA) ; Rocha, J.A. (UFMA-CIENATEC) ; Lima, B.A.V. (UFMA-CIENATEC)
Resumo
Bactérias como E. coli S. aureus, S. epidermidis e E. Feacalis são microrganismo causadores de diversas enfermidades infecciosas. O processo de docking molecular interagiu o complexo metálico de zinco com ligante as bactérias com intuito de analisar as interações moleculares através do programa AUTODOCKING 1.5.6. Através do mesmo, foi possível analisar as interações hidrofóbicas, pontes de hidrogênio, energia de ativação e as constantes de inibição com ressalva nas enzimas 3KT5 e 3ZKF, sendo as que possuem menor energia de interação, -8,72 Kcal.mol-1 e -6,01 Kcal.mol-1, respectivamente, demonstrando resultados satisfatórios para as simulações.
Palavras chaves
Bactérias; Docagem Molecular; Complexo de zinco
Introdução
A partir do marco do computador digital Lenhard (2014), o mesmo oferecia uma capacidade funcional, que os químicos quânticos da época definiam como crucial pois viabilizariam muitos processos que não se podia fazer com os outros equipamentos antigos e obsoletos. O intuito do trabalho será o estudo da interação entre o complexo de zinco {Zn[CH3O(NO2)C6H3NNNC6H3(NO2)OCH3]2(C5H5N)2} (VILLIS, 2007), em enzimas bacterianas de Staphylococus aureus, Escherichia coli, Enterococcus Faecalis e Staphylococcus epidermidis disponíveis no banco de dados Protein Data Bank (PDB). O recurso terapêutico para infecção da S. aureus é extremamente árduo sendo um tratamento de aplicação de gotas de intervalos de 30 minutos por 48 horas. Além da mesma adquirir resistência a antibióticos com o tempo (KHAN, et al., 2016). A S. epidermidis era considera um comensal de pele inofensiva, ao logo dos estudos comprovou se que a mesma é responsável pela maioria da sepse neonatal tardia correlacionado com morbidades inflamatórias neonatais, a mais comum é a displasia bronco pulmonar (DONG et al. 2019). A E. coli possui vários clones altamente adaptados que adquiriram atributos de virulência específicos, o que confere uma maior capacidade de adaptação a novos nichos e permite que eles causem um amplo espectro de doenças (KAPER, NATARO, MOBLEY, 2004). Dentre a produção de alteração patológica para produzir toxinas, em alguns casos pode ser fatal em organismos debilitados que contenham a Enterococcus Faecalis (KAYAOGLU e ØRSTAVIK, 2004). A docagem molecular, consiste na predição da conformação bioativa de uma pequena molécula (ligante) no sítio de ligação de uma macromolécula (proteína-alvo), seguido da avaliação (pontuação) e classificação do modo de ligação proposto (GUIDO 2010).
Material e métodos
Obteve-se o complexo de zinco no banco de dados de moléculas no The Cambridge Crystallographic Data Centre (CCDC) e em seguida utilizou-se o programa Gaussian 9w para realização do cálculo de otimização estrutural molecular com método de Density Funcional Theory (DFT), usando o funcional hibrido BLYP e a base Gen/LANL2DZ para a molécula Zinco e 6-311-G para demais átomos (ABDEL-LATIF, 2018). Para a preparação do ligante e proteína foi utilizado o programa AUTODOCKTOOLS (SILVA et al, 2018). Para realização da docagem utilizaram os alvos disponíveis no banco de dados PDB (Protein Data Bank). Para a S. aureus serão estudas as moléculas e seus códigos, (1KA5) (MAURER et al., 2004), (2H29) (HAN et al., 2006). Para Escherichia coli (2GE5) (HILLER e PERONA, 2004) e (3ZFK) (CZENE et al, 2012). As enzimas utilizadas para Staphylococcus epidermides são (3DON) (HAN et al, 2009) e (3QT5) (BARTA et al 2011). Por último as enzimas de Enterococcus faecalis (2SQC) (WENDT, LENHART e SCHULZ, 1999) e (4GEE) (TARI et al, 2013). Para realização das simulações foi utilizada o software buscando os alvos nas moléculas centralizando o grid nas coordenadas, utilizando uma caixa cubica 60x60x60 e parâmetros metálico AD4, com algarismo de genérico Lamarckian GA (ROCHA, 2018; FERREIRA, 2015).
Resultado e discussão
Os resultados para as simulações do complexo metálico pelo ligante e a proteína
3KT5 como parâmetro possui energia de ligação igual a -8,72 Kcal.mol-1 e
constante de inibição de 0,4078x10-3 µM, havendo nesse complexo formação de
pontes de hidrogênio, são elas: ASN287, LYS189, LYS68, SER107, SER192. Este
também gerou resíduos hidrofóbicos ALA284, ARG193, GLY103, GLY285, LEU104,
LYS188, PRO286, SER106, SER139, THR100, TYR256 (Fig. 1A, 1B). Essas informações
podem ser observadas na Tabela 1. O complexo obtido pelo ligante em conjunto com
a proteína 3ZKF, obteve se como resultado a energia interação -6,01 Kcal.mol-1 e
uma constante de inibição 39,24µM. nessa interação houve uma ponte de hidrogênio
SER446 e as interações hidrofóbicas ocorreram nos aminoácidos ARG496, ARG538,
GLU542, HIS544, HIS545, HIS569, HIS573, ILE549, ILE570, LEU543, PHE492, PRO447
(Fig. 1C, 1D).
A enzima 2GE5 da E. coli demonstrou resultados insatisfatórios dos demais, sendo
especificações: não há constate de inibição, energia de ligação alta e tanto não
há ligações de hidrogênio como nenhuma interação hidrofóbicas.
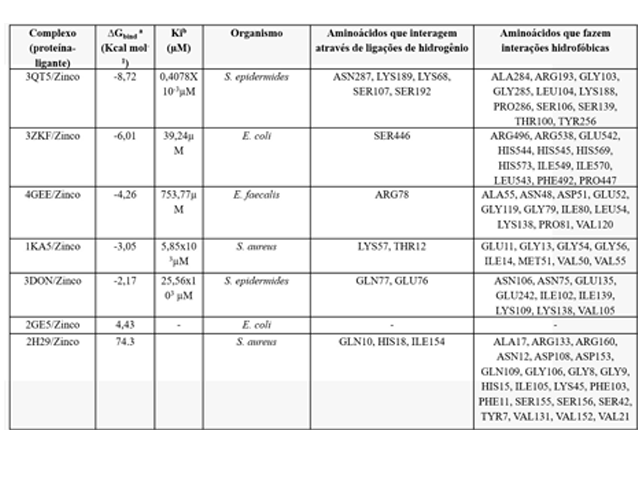
Parâmetros de Docagem Molecular
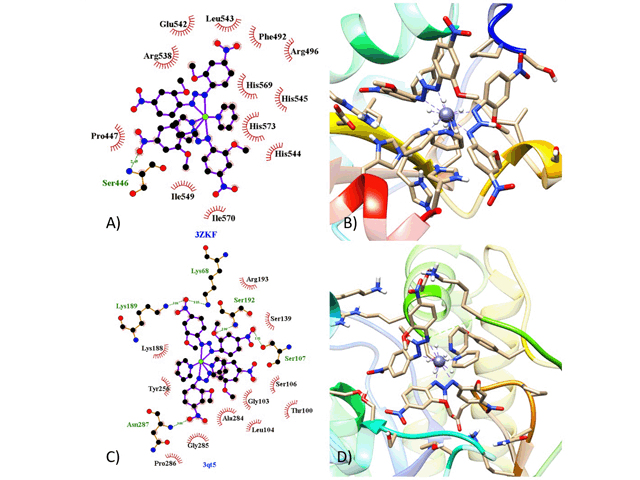
Docagem Molecular, A)Interação em 2D (3ZKF), B) Interação de Ribbons (3ZKF), C) Interação em 2D (3QT5), D) Interação de Ribbons (3QT5).
Conclusões
Percebe-se que a enzima 3KT5 da S. epidermides, no processo de docagem, demonstrou ser eficaz do complexo de zinco e ligantes. Comprovou um excelente rendimento nas interações hidrofóbicas e eficiência na baixa energia de interação atribuída com cinco interações de pontes de hidrogênio. Outra enzima com resultados consideráveis foi a 3ZKF, com a interação do complexo metálico com o ligante, do organismo E. coli. A mesma possui várias interações hidrofóbicas e uma ponte de hidrogênio além de ter a segunda menor energia de ligação e constante de inibição mais elevada que a primeira enzima 3KT5.
Agradecimentos
Agradeço a Deus, minha família, aos membros do grupo de pesquisa CIENATEC, aos técnicos da UFMA. Ao professor e Orientador Benedicto Augusto Vieira Lima. Ao estudante e auxiliador Ruan Bastos.
Referências
ABDEL-LATIF, Samir A.; MOHAMED, Adel A. Novel Zn (II) complexes of 1, 3-diphenyl-4-(arylazo) pyrazol-5-one derivatives: Synthesis, spectroscopic properties, DFT calculations and first order nonlinear optical properties. Journal of Molecular Structure, v. 1156, p. 712-725, 2018.
BARTA, Michael L. et al. Crystal structures of Staphylococcus epidermidis mevalonate diphosphate decarboxylase bound to inhibitory analogs reveal new insight into substrate binding and catalysis. Journal of Biological Chemistry, v. 286, n. 27, p. 23900-23910, 2011.
CZENE, Anikó et al. A new insight into the zinc-dependent DNA-cleavage by the colicin E7 nuclease: a crystallographic and computational study. Metallomics, v. 6, n. 11, p. 2090-2099, 2014.
DONG, Ying et al. An underestimated pathogen: Staphylococcus epidermidis induces pro-inflammatory responses in human alveolar epithelial cells. Cytokine, v. 123, p. 154761, 2019.
FERREIRA, Isabelle Mariane de Lima. Estudo por simulação molecular do sistema proteína quinase-inibidores NATAL, RN 2015. 2015. Trabalho de Conclusão de Curso. Universidade Federal do Rio Grande do Norte.
GUIDO, Rafael VC; ANDRICOPULO, Adriano D.; OLIVA, Glaucius. Planejamento de fármacos, biotecnologia e química medicinal: aplicações em doenças infecciosas. estudos avançados, v. 24, n. 70, p. 81-98, 2010.
HAN, Seungil et al. Crystal structure of nicotinic acid mononucleotide adenylyltransferase from Staphyloccocus aureus: structural basis for NaAD interaction in functional dimer. Journal of molecular biology, v. 360, n. 4, p. 814-825, 2006.
HAN, Cong et al. X‐ray crystallographic and enzymatic analyses of shikimate dehydrogenase from Staphylococcus epidermidis. The FEBS journal, v. 276, n. 4, p. 1125-1139, 2009.
HILLER, David A.; PERONA, John J. Positively charged C-terminal subdomains of EcoRV endonuclease: contributions to DNA binding, bending, and cleavage. Biochemistry, v. 45, n. 38, p. 11453-11463, 2006.
KAPER, James B.; NATARO, James P.; MOBLEY, Harry LT. Pathogenic escherichia coli. Nature reviews microbiology, v. 2, n. 2, p. 123, 2004.
KAYAOGLU, Güven; ØRSTAVIK, Dag. Virulence factors of Enterococcus faecalis: relationship to endodontic disease. Critical Reviews in Oral Biology & Medicine, v. 15, n. 5, p. 308-320, 2004.
LENHARD, Johannes. Studies in History and Philosophy of Science Disciplines, models, and computers: The path to computational quantum chemistry. Elsevier. Unversity of Bielefeld Germany. p. 89-96. 2014.
MAURER, Till et al. High-resolution structure of the histidine-containing phosphocarrier protein (HPr) from Staphylococcus aureus and characterization of its interaction with the bifunctional HPr kinase/phosphorylase. Journal of bacteriology, v. 186, n. 17, p. 5906-5918, 2004.
ROCHA, Jefferson A. et al. Computational quantum chemistry, molecular docking, and ADMET predictions of imidazole alkaloids of Pilocarpus microphyllus with schistosomicidal properties. PloS one, v. 13, n. 6, p. e0198476, 2018.
SILVA, Jacilene et al. ESTUDO COMPARATIVO DE DOCKING MOLECULAR ENTRE O INIBIDOR DE PROTEASE SAQUINAVIR E O CAROTENOIDE BIXINA COMO POTENCIAL INIBIDOR DO VÍRUS HIV TIPO I (1HXB). Revista Expressão Católica Saúde, v. 3, n. 1, p. 35-41, 2018.
TARI, Leslie W. et al. Pyrrolopyrimidine inhibitors of DNA gyrase B (GyrB) and topoisomerase IV (ParE). Part I: Structure guided discovery and optimization of dual targeting agents with potent, broad-spectrum enzymatic activity. Bioorganic & medicinal chemistry letters, v. 23, n. 5, p. 1529-1536, 2013.
VILLIS, Paulo Cesar Mendes et al. Avaliação da auto-associação molecular de complexos metálicos com ligantes triazenidos derivados do 1, 3-bis (fenil) triazeno. 2007. Tese de Doutorado. Universidade Federal de Santa Maria.
WENDT, K. U.; LENHART, A.; SCHULZ, G. E. The structure of the membrane protein squalene-hopene cyclase at 2.0 Å resolution. Journal of molecular biology, v. 286, n. 1, p. 175-187, 1999.









