Metabolômica aliada a RMN de ¹H no diagnóstico não invasivo do Câncer Colorretal
ISBN 978-85-85905-25-5
Área
Bioquímica e Biotecnologia
Autores
Ursulino, J.S. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL) ; Vieira, A.D. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL) ; Crispim, A.C. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL) ; Bento, E.S. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL) ; Aquino, T.M. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL) ; Neto, M.A.F.L. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL) ; Caetano, L.C. (UNIVERSIDADE FEDERAL DE ALAGOAS - UFAL)
Resumo
Melhores técnicas são necessárias para diagnosticar precocemente o Câncer de Colorretal (CCR) de modo não invasivo. A Ressonância Magnética Nuclear de Hidrogênio (RMN de ¹H) foi usada em um total de 16 amostras, sendo 5 diagnosticadas com CCR e 11 classificadas como Grupo Controle (GC), combinando com a Metabolômica fornece um parâmetro geral de potenciais biomarcadores, criando assim perfis metabólicos. O reconhecimento de padrões através da análise de componentes principais (PCA) e análise discriminante de mínimos quadrados parciais ortogonais (OPLS-DA) foi aplicado em dados processados por RMN de ¹H. OPLS-DA revelou a distinção entre os grupos, posteriormente validado por validação cruzada. ANOVA (p<0.05) mostrou que um sinal largo em 8.5ppm está presente apenas em pacientes com CCR.
Palavras chaves
CCR; RMN de ¹H; Metabolômica
Introdução
A metabolômica tem sido definida como a abrangente análise quantitativa de todos os metabólitos de um sistema biológico em estudo (FIEHN, O., 2001). Utilizando resultados de Ressonância Magnética Nuclear de Hidrogênio (RMN de ¹H), são criados dois ou mais grupos. Estes grupos geralmente incluem pelo menos um grupo de organismos que tem uma doença, ou foi exposto a um agente contaminante, droga ou toxina. Um grupo controle constituído por organismos que não apresente qualquer enfermidade ou tenha sido exposto a agente contaminante, droga ou toxina. Os resultados dessas análises revelam metabólitos cujas concentrações são estatisticamente diferentes entre os grupos de organismos em estudos, levando a identificação de biomarcadores, comumente chamados de discriminantes (GOLDANSAZ, S. A., et al., 2017; JOHNSON, C. H., IVANISEVIC, J., SIUZDAK, G., 2016). As ferramentas quimiométricas mais utilizadas na metabolômica incluem Análise de Componentes Principais (PCA), método não supervisionado empregado para verificar a separação dos grupos antes da classificação dos dados por meio das componentes principais (PCs). Para a classificação de dois ou mais grupos (saudável x doente), é uma tarefa comum com a ferramenta de Projeção Ortogonal para Análise Discriminante de Estruturas Latentes (OPLS-DA), classificada como método supervisionado. Outro recurso muito empregado é a Análise de Variância (ANOVA), com essa ferramenta, as intensidades dos metabólitos são comparadas entre indivíduos saudáveis e doentes, quando desregulados, a identificação do metabólito serve como biomarcador de uma determinada doença. Todo resultado deve ser validado, seja por validação cruzada ou permutação (ZACHARIAS, H. E., MICHAEL, A., GRONWALD, W., 2018). Células tumorais provenientes do CCR causam pertubações metabólicas característicos de uma disbiose, desequilíbrio da microbiota intestinal. Lin, et al., (2016) observaram em pacientes com CCR o aumento do consumo da glicose o que leva a diminuição na concentração de glicose fecal em paralelo um aumento no lactato e alanina, consistente com o aumento do metabolismo energético, e esta observação é apoiada por uma elevação no succinato e glutamato na ativação do ciclo de krebs. Recursos tradicionais como o uso da colonoscopia como uma ferramenta de triagem é limitado porque é um procedimento invasivo e desagradável que requer preparação intestinal e sedação. Em vez disso, o teste de sangue oculto nas fezes é comumente usado na clínica. Uma variação inaceitavelmente ampla ou a falta de sensibilidade e especificidade do teste tem dificultado sua aplicação clínica no diagnóstico de CCR, especialmente para lesões pré- cancerosas (LEVIN, B. et al., 2008). Marcadores tumorais, como o antígeno carcinoembrionário (CEA) e o antígeno carboidrato 19-9 (CA 19-9) também são comumente usados na clínica. No entanto, esses marcadores tumorais não podem ser usados isoladamente para rastrear ou diagnosticar o CRC porque sua sensibilidade é baixa (WATANABE, T. et al., 2012) A RMN ¹H para análise do metaboloma de tecidos e bifluídos aliada a quimiometria com a aplicação da PCA, OPLS-DA, ANOVA e Validação Cruzada, viabilizam biomarcadores para discriminação entre CCR e GC. A criação de um perfil metabólico pode ser usada para compreender as rotas do metabolismo em condições saudáveis, tal como, em circunstâncias que foram instigadas a um distúrbio metabólico, interrompendo o metabolismo em determinada etapa que não condiz com o produto final (NICHOLSON, J. K., LINDON J.C., 2008).
Material e métodos
As amostras de fezes foram todas obtidas no Hospital Universitário Professor Alberto Antunes – HUPAA por meio do Termo de Consentimento Livre e Esclarecido (T.C.L.E.), e foram submetidos ao questionário Protocolo de Pesquisa. Os pacientes foram recrutados no ambulatório de Gastroenterologia da seção médica do HUPAA, Maceió – Alagoas. Todos os pacientes com CCR convocados subsequentemente tiveram seu diagnóstico verificado por critérios clínicos e laboratoriais. Para a criação do GC, foi utilizado como critério de classificação a ausência de qualquer enfermidade clínica e laboratorial presente no TGI e qualquer distúrbio neurológico (BJERRUM, 2015). As águas fecais foram extraídas dentro de uma capela microbiológica em um almofariz no qual foi adicionado nitrogênio líquido e trituradas com o pistilo até a sua homogeneização total. Assim o material é transferido para um eppendorf no qual em seguida é submetido a centrifugação por 15 minutos a 14000 rotações por minuto (RPM). Assim, com auxílio de uma micropipeta 350μL (microlitro) das águas fecais são coletadas e finalmente adicionadas a uma misturada preparada em um tubo de RMN com 350μL de tampão fosfato 10% D¬¬2¬O com TSP a um pH=7,40. Todos os experimentos de RMN de ¹H foram realizados a 298,15K, usando um espectrómetro Bruker de 400 MHz do laboratório Núcleo de Análises e Pesquisa em Ressonância Magnética Nuclear (NAPRMN). Os espectros foram adquiridos com 64K pontos, 128scans com a largura espectral de 10,5ppm e supressão do sinal da água utilizando-se a sequência de pulsos noesypr1d, com tempo de relaxação de 2 segundos (s). Os resultados foram processados no programa TOPSPIN realizando manualmente as distorções de fase e de linha de base e referenciados ao sinal do TSP a 0,0ppm. O sinal da água 4.8ppm foi removido de todos os espectros para análise MVA, pois embora tenha sido realizado a supressão o sinal persistiu em aparecer. Inicialmente é realizado a análise PCA dos dados espectrais de RMN de ¹H para visualizar a estrutura geral do conjunto de dados e assim identificar qualquer anormalidade (com base nos princípios de Hotelling T²) e assim obter as PCs. Em seguida, os métodos multivariados supervisionados, OPLS-DA é aplicado facilitando a interpretação do modelo obtido, descriminando assim o perfil metabólico de cada grupo em questão. Um teste final de significância foi realizado com o uso do teste Análise de Variância, ANOVA (p<0,05). Os metabólitos responsáveis pela diferença entre os perfis metabólico de cada grupo, indicando pesos variáveis discriminatórios (ERIKSSON, et al., 2008).
Resultado e discussão
Com a mera inspeção visual dos resultados da espectroscopia de RMN de ¹H foi
possível a identificação dos metabólitos acetato, butirato, propionato,
alanina, glicina, etanol. Por meio da abordagem da metabolômica analisando
as águas fecais, foi possível a criação de dois grupos e realizar a
discriminação.
Para explorar ao máximo os resultados a PCA foi utilizada fornecendo
tendências e uma visão geral dos dados classificando as amostras de acordo
com grau de semelhança. Os valores das Componentes 1 e 2 são respectivamente
29.11% e 15.97%, sendo a componente 1 a responsável por classificar os
grupos.
O OPLS-DA foi aplicado aos nossos dados porque pode discriminar entre
grupos, com valores de componente 1 igual a 25.09% e componente 2 igual a
8.93% torna este modelo, embora prévio, com uma discriminação
estatisticamente significativa, sendo possível classificar as amostras de
CCR e GC.
A Análise de Variância (ANOVA p<0.05) serviu para reduzir o número
possivelmente grande de metabólitos para apenas aqueles que mostram a
resposta mais forte sob as condições investigadas, ou seja, examinar apenas
aqueles metabólitos que discriminam os grupos (ERIKSSON, et al., 2008). Os
ácidos graxos de cadeia curta (AGCC), como acetato (p=0.022255), propionato
(p=0.01649) e butirato (p=0.042202) são responsáveis pelo o equilíbrio
imunitário intestinal e servem como fonte de energia para as células
epiteliais do cólon (GHAZALPOUR, et al., 2016) em nossos resultados
revelaram uma covariância positiva para o GC além de estarem em maior
quantidade nas águas fecais.
Os sinais 8.5ppm e 6.0ppm ainda não foram identificados. O sinal 8.5ppm é
largo e aparece apenas no grupo CCR com valor de p=0.00012959. O 6.0ppm tem
p=0.003302. Ambos os sinais tem covariância positiva para CCR.
Os resultados foram validados por meio da Validação Cruzada, mostrando uma
precisão de 100.0% do resultado, no qual 100.0% das amostras do GC estão bem
classificadas assim como as amostras dos pacientes com CCR mostraram 100.0%
de classificação.
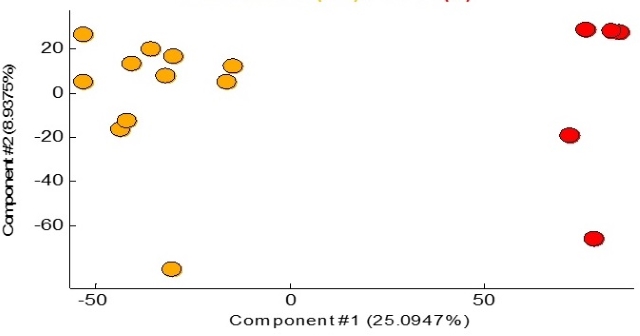
Cada ponto corresponde a um espectro obtido. Em amarelo temos 11 amostras do GC e em vermelho 5 de CCR. É possível observar dois grupos bem definidos.
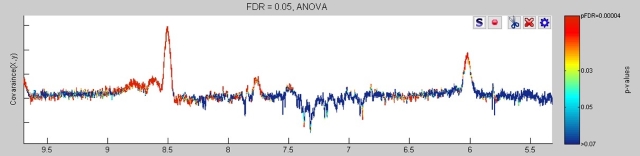
Os sinais 8.5ppm e 6.0ppm apresentam covariância positiva (cor vermelha) para o CCR e discriminam do GC.
Conclusões
A metabolômica baseada em espectroscopia de RMN ¹H de fezes humanas oferece uma oportunidade importante para investigar alterações metabólicas associadas à CCR que podem servir como biomarcadores. Nosso estudo foi desenvolvido para investigar diferentes padrões entre pacientes com CCR em comparação com controles saudáveis, assim como no estudo de Lin Y. et al., 2016 os AGCC caracterizam o GC. Embora seja um estudo preliminar demonstra um potencial método de classificação do Câncer Colorretal, isto porque a validação cruzada das amostras analisadas mostrou 100% de classificação.
Agradecimentos
Agradeço a UFAL e aos órgãos de fomento FAPEAL, CNPq e CAPES.
Referências
BJERRUM J. T., et al. Metabonomics of human fecal extracts characterize ulcerative colitis, Crohn’s disease and healthy individuals. Metabolomics. 11:122–133. 2015.
ERIKSSON, L., TRYGG, J., WOLD, S.. CV-ANOVA for significance testing of PLS and OPLS® models. Journal of Chemometrics. 22(11–12):594–600. 2008.
FIEHN, O., Combining genomics, metabolome analysis, and biochemical modelling to understand metabolic networks. Comp. Funct. Genom. 2 (3), 155e168. 2001.
GHAZALPOUR, et al., Expanding Role of Gut Microbiota in Lipid Metabolism. Curr Opin Lipidol . 2016 April ; 27(2): 141–147
GOLDANSAZ, S.A., et al., Livestock metabolomics and the livestock metabolome: a systematic review. PLoS One 12 (5), e0177675. 2017.
JOHNSON, C.H., IVANISEVIC, J., SIUZDAK, G., Metabolomics: beyond biomarkers and towards mechanisms. Nat. Rev. Mol. Cell Biol. 17 (7), 451e459. 2016.
Levin, B. et al. Screening and surveillance for the early detection of colorectal cancer and adenomatous polyps, 2008: a joint guideline from the American Cancer Society, the US Multi-Society Task Force on Colorectal Cancer, and the American College of Radiology. CA Cancer J Clin 58, 130–160, 2008
LIN, Y., et al. NMR-based fecal metabolomics fingerprinting as predictors of earlier diagnosis in patients with colorectal câncer. Oncotarget. 2016 May 17;7(20):29454-64.
NICHOLSON, J. K., LINDON J. C. Metabonomics. Nature, v. 455, p. 1054-1056, 2008.
Watanabe, T. et al. Japanese Society for Cancer of the Colon and Rectum (JSCCR) guidelines 2010 for the treatment of colorectal cancer. Int J Clin Oncol 17, 1–29, 2012.
ZACHARIAS, H. U., ALTENBUCHINGER, M., GRONWALD, W., Statistical Analysis of NMR Metabolic Fingerprints: Established Methods and Recent Advances. Metabolites, 8, 47, 2018.









