Análise de metabólitos em folhas de Dendê (Elaeis guineensis) com uso de imprinting MALDI Imaging MS
ISBN 978-85-85905-25-5
Área
Química Analítica
Autores
Rodrigues Neto, J.C. (UFG/EMBRAPA) ; Teixeira, T.S. (UFG/EMBRAPA) ; Souza Junior, M.T. (EMBRAPA) ; Abdelnur, P.V. (UFG/EMBRAPA)
Resumo
O imageamento químico por espectrometria de massas é uma técnica cada vez mais usada no estudo do perfil metabólico de superfícies vegetais, como as folhas. Porém, devido a espessura de algumas folhas como a de dendê (Elaeis guineensis) a ionização é desfavorável e por consequência a imagem química por MALDI Imaging MS também. Dessa forma, esse trabalho tem como objetivo apresentar um método de preparo de amostra de folha de dendê por imprinting em papel filtro, seguido de aplicação de matriz (DHB) por sublimação e análise metabolômica por MALDI Imaging MS. O perfil químico foi avaliado com a detecção em diferentes regiões da folha e identificação de metabólitos primário e secundários pertencentes a diferentes classes biológicas, como terpenos glicosilados, dissacarídeos e álcoois graxos.
Palavras chaves
metabolômica; espectrometria de massas; MALDI-IMS
Introdução
A Metabolômica é uma abordagem que vem sendo cada vez mais utilizada para relacionar características genotípicas e fenotípicas na busca de marcadores químicos de doenças (Abdelnur, 2010; Roessner et al, p. 11, 2001), influência no perfil metabólico por estímulo externo, interação planta- herbívoro, controle de qualidade e determinação da atividade de plantas medicinais (Wolfender et al, p. 1056, 2013). Devido à alta complexidade dos processos bioquímicos que ocorrem em sistemas biológicos, em alguns casos é necessária a elucidação dos compostos envolvidos, bem como sua localização espacial e distribuição no organismo. Nesses casos, a imagem química por espectrometria de massas é uma abordagem que se destaca (Agin et al., 2016). Dentre as técnicas de imagem química por espectrometria de massas, têm-se DESI-IMS (do inglês, Desorption Electrospray Ionization Imaging Mass Spectrometry), MALDI-IMS (do inglês, Matrix-Assisted Laser Desorption Ionization – Imaging Mass Spectrometry) e LDI-MSI (do inglês, Laser Desorption Ionization – Imaging Mass Spectrometry) Sendo que as duas últimas possuem a ampla utilização associada a possibilidade de uso de analisadores de alta resolução, como o TOF (do inglês, Time of Flight), e por permitir uma melhor resolução de imagem com uso de lasers cada vez mais sofisticados, que em módulos comerciais vão de 20 a 200µm de diâmetro. No entanto, diversos desafios ainda precisam ser superados nas análises por MALDI-MSI, desde a aplicação da matriz até a ionização dos compostos presentes na folha (Rodrigues-Neto, 2017). Em relação a aplicação da matriz, a técnica que tem apresentado melhor resultado pela alta reprodutibilidade e elevada uniformidade da deposição da matriz é a sublimação. Em relação a ionização de compostos, o desafio segue devido à dificuldade de acesso do laser do MALDI no interior de algumas folhas, como de dendê (Elaeis guineensis), que contêm espessas camadas de cera e parede vegetal em sua composição fisiológica. Para superar tais desafios, o imprinting foi utilizado, pela primeira vez, em folhas de Elaeis guineensis para análise por MALDI-IMS. A técnica se fundamenta na transferência dos compostos químicos da matriz original (folha) para uma superfície porosa, que pode ser teflon, EVA, papel filtro, entre outros, para que essa superfície seja analisada no lugar da folha, reduzindo eventuais problemas derivados da rígida estrutura da folha. O imprinting tem se mostrado como um método robusto e reprodutível quando aplicado em análises por DESI (do inglês, Desorption Electrospray Ionization) (Thunig et al, p. 3256, 2011), permitindo a detecção de sinais intensos e estáveis, uma vez que a presença da cutícula foliar e da parede celular é eliminada. Nesse experimento o papel filtro foi utilizado como superfície porosa e a matriz escolhida foi o ácido 2,5-dihidrobenzóico (DHB), que é indicada para análise de compostos de baixa massa molecular.
Material e métodos
Na realização do preparo de amostra por imprinting, uma prensa de aço inox foi construída, onde duas placas de aço são sobrepostas e prensadas entre si por 4 parafusos com rosca. O material vegetal utilizado foi a folha de dendê (Elaeis guineensis), coletada na Embrapa Agroenergia, que foi lavada com água corrente e levada para as análises no laboratório. Um pedaço da folha foi cortado, com cerca de 5 cm de comprimento, de forma a ter um material representativo de toda a folha, contendo as nervuras central e secundárias. Essa amostra foi então colocada na prensa de imprinting. A extração dos metabólitos do material vegetal fresco para a superfície porosa do papel filtro (Qualy, 205 μm de espessura, tamanho de poro médio de 14 μm e gramatura de 80 g/m²) foi realizada por pressão por 20 min através da prensa manual, com adaptações do método descrito por Thunig et al. (2011). Em seguida o papel filtro contendo os metabólitos do material vegetal foi retirado da prensa e fixado com fita dupla face condutora em uma placa de vidro para MALDI com ITO (do inglês, Indium Tin Oxide) Bruker Daltonics, Alemanha). Esta placa de vidro contendo a amostra foi então submetida a aplicação da matriz DHB (ácido 2,5-dihidroxidobenzoico, 50 mg/mL) por sublimação (Ace Glass, E.U.A) a 200 mBar, 200 ºC, por 15 min. Os experimentos de MALDI-IMS foram feitos no espectrômetro de massas modelo UltrafleXtreme (Bruker Daltonics, Alemanha) equipado com fonte de ionização MALDI e analisador de massas de alta resolução tipo TOF, com possibilidade de uso no modo linear ou refletor. Os dados de imagem foram obtidos no software flexImaging, versão 4.0 (Bruker Daltonics, Alemanha) e os espectros de massas obtidos das imagens foram avaliados no software flexAnalysis, versão 3.4 (Bruker Daltonics, Alemanha). As análises foram feitas no modo refletor negativo, na faixa de massas m/z 100-800, com frequência de 2000 Hz, raster width (espaço entre disparos) de 120 µm e laser de 120 µm de diâmetro, para um rastreamento mais eficiente de toda a superfície. Os dados de imagem foram processados aplicando normalização de RMS, filtro de massa para o íon m/z ± 0,5%, atribuindo escala de cores com variação de intensidade de 0 a 100%.
Resultado e discussão
Foi observada uma aplicação uniforme da matriz, característica da
reprodutibilidade do método de sublimação. A matriz DHB foi escolhida pela
conhecida eficiência na análise de metabólitos em MALDI (Goodwin, p. 4893,
2012) e capacidade de sublimação a partir de 140 ºC (Thomas et al, p. 2048,
2012).
Foram feitos 35.500 pontos na superfície de 225 mm2, em cerca de
9 horas de análise. E apesar da faixa de detecção da matriz se situar entre
m/z 150-400, foi possível distinguir os sinais de massas de interesse
dos sinais da matriz pela seleção de uma região fora da folha, onde a matriz
também foi aplicada.
Quatorze compostos foram observados nas imagens obtidas da folha de E.
guineensis. Na Figura 01 é mostrado o espectro de massas total com a média
das intensidades de cada íon detectado.
Dentre os íons detectados a localização química foi muito semelhante, e
destes, três compostos tiveram imagem química, fórmula molecular e classe
química atribuída. A atribuição dos compostos foi feita por comparação com o
banco de dados de metabolômica KEGG (Kanehisa et al. P. 27, 2000) avaliando
compostos com atividade biológica para indicação do composto mais provável,
sendo que a confirmação da identificação será feita em experimentos
posteriores de fragmentação. Também foi utilizado o banco de dados BMDB
(Bioenergy Metabolomics DataBase), criado pelo nosso grupo de pesquisa na
Embrapa Agroenergia que contém dados de metabólitos detectados por
espectrometria de massas (LC-ESI-MS/MS) em diversas espécies, incluindo o
Elaeis guineensis.
Na Figura 02 (A), o íon m/z 346, atribuído a um terpeno glicosilado
de fórmula molecular C16H28O8, teve sua
distribuição observada principalmente na nervura central e extremidade
esquerda da folha. Essa distribuição específica do composto sob a superfície
foliar pode estar relacionada a ação deste para defesa da planta. De forma
geral, terpenos são metabólitos secundários produzidos pela planta e
utilizados como mecanismo de defesa, por estresses bióticos e/ou abióticos.
As vias metabólicas do melavonato e do 1-desoxixilulose-5-fosfato (DXP) são
as mais afetadas pelo aumento ou diminuição da concentração dessa classe
metabólica (Kitaoka et al, 2015).
Já o íon m/z 424, Figura 02 (B), foi atribuído ao álcool graxo
nonacosan-10-ol (C29H60O), que teve sua distribuição
mais intensa e uniforme nas áreas acima e abaixo da nervura central. Álcoois
graxos de cadeias extensas geralmente são encontrados em plantas e são
obtidos a partir da transesterificação de triglicerídeos, que já foi
observado em espécies de palmeiras – Ordem das Arecales (Kreutzer, 1984). O
fato do dendê pertencer a ordem das Arecales corrobora para que o composto
tenha sido corretamente atribuído.
O íon m/z 502, como mostrado na Figura 02 (C), foi observado com
maior intensidade nas nervuras central e secundária inferior. Ele é
atribuído a um dissacarídeo de fórmula molecular
C24H41NO10. Os dissacarídeos são
metabólitos primários relacionados a diversos processos bioquímicos,
principalmente a produção de energia das plantas, pela fonte de carbono
proveniente dos carboidratos que compõe sua estrutura. A presença deste
dissacarídeo nas nervuras da folha sugere que esse composto não é produzido
na folha e sim levado até ela pela condução da seiva elaborada da planta
(Raven et al., 2014).
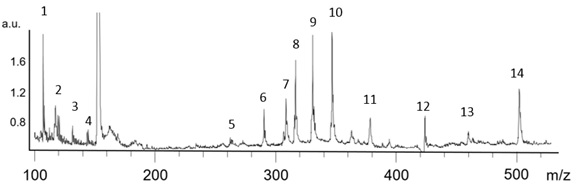
Espectro total médio dos íons detectados por MALDI-IMS em dendê. Apenas sinais referentes a amostra tiveram números atribuídos.
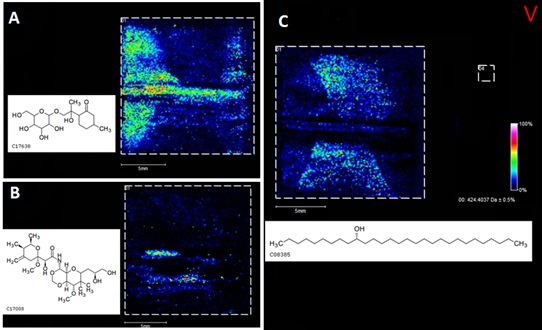
Imagens químicas de metabólitos em folha de dendê. A) m/z 346; B) m/z 424; C) m/z 502.
Conclusões
O método de imageamento utilizando o imprinting teve como principal vantagem o acesso a metabólitos internos. Quatorze íons foram detectados e tiveram sua localização química determinada, sendo que destes três deles tiveram fórmula molecular e classe química atribuída. Dessa forma, o imageamento químico se mostra útil para aplicações em diversas áreas,como na descoberta e detecção de biomarcadores de doenças, na interação planta-patógeno ou no estudo de perfis químicos , levando a um melhor entendimento dos processos biológicos envolvidos.
Agradecimentos
Referências
Abdelnur, P. V. A espectrometria de massas e as bio-moléculas: Relação estrutura/reatividade de peptídeos por reações íon/molécula e mobilidade de íons e busca de novos biomarcadores em clínica médica por imageamento químio-seletivo de tecidos. 2010. 188 f. Tese (Doutorado em Química Orgânica) – Instituto de Química, Universidade Estadual de Campinas, Campinas, SP.
Agin, A.; Heintz., D.; Ruhland, E.; Chao De La Barca, J.M.; Zumsteg, J.; Moal, V.; Gauchez, A.S.; Namer, I.J. Metabolomics – an overview. From basic principles to potential biomarkers (Part 1). Médecine Nucéaire. v.40, p.4-10, 2016
Goodwin, Richard JA. "Sample preparation for mass spectrometry imaging: small mistakes can lead to big consequences." Journal of proteomics, 75.16 (2012): 4893-4911.
Kanehisa, Minoru, and Susumu Goto. "KEGG: kyoto encyclopedia of genes and genomes." Nucleic acids research 28.1 (2000): 27-30.Kitaoka, Naoki, et al. "The application of synthetic biology to elucidation of plant mono-, sesqui-, and diterpenoid metabolism." Molecular plant 8.1 (2015): 6-16.
Kreutzer, Udo R. "Manufacture of fatty alcohols based on natural fats and oils." Journal of the American Oil Chemists' Society 61.2 (1984): 343-348.
Raven, P.H., Evert, R.F. & Eichhorn, S.E. 2014. Biologia Vegetal, 8a. ed. Coord. Trad. J.E.Kraus. Editora Guanabara Koogan, Rio de Janeiro.
Rodrigues-Neto, J.C. "Prospecção de marcadores químicos relacionados ao Amarelecimento Fatal em folhas de Elaeis guineensis utilizando abordagem metabolômica." 2017. Dissertação (Mestrado em Química) – Instituto de Química, Universidade Federal de Goiás, Goiânia, GO.
Roessner, Ute, et al. "Metabolic profiling allows comprehensive phenotyping of genetically or environmentally modified plant systems." The Plant Cell 13.1 (2001): 11-29.
Thomas, Aurélien, et al. "Sublimation of new matrix candidates for high spatial resolution imaging mass spectrometry of lipids: enhanced information in both positive and negative polarities after 1, 5-diaminonapthalene deposition." Analytical chemistry 84.4 (2012): 2048-2054.
Thunig, Janina, Steen H. Hansen, and Christian Janfelt. "Analysis of secondary plant metabolites by indirect desorption electrospray ionization imaging mass spectrometry." Analytical chemistry 83.9 (2011): 3256-3259.
Wolfender, Jean-Luc, et al. "Plant metabolomics: from holistic data to relevant biomarkers." Current Medicinal Chemistry 20.8 (2013): 1056-1090.









