DISTINÇÃO ENTRE SEMENTES DE ALGODÃO CONVENCIONAIS DE TRANSGÊNICAS UTILIZANDO ESPECTROSCOPIA NIR
ISBN 978-85-85905-25-5
Área
Química Analítica
Autores
Rocha, P.D. (UEPB) ; Farias, I.K.T. (UEPB) ; Silva, J.L.B. (UEPB) ; Simões, S.S. (UEPB) ; Medeiros, E.P. (EMBRAPA ALGODÃO)
Resumo
Técnicas de melhoramento genético são utilizadas para resolver problemas como resistência a pragas de algodoeiro.O uso destas técnicas, exige fiscalização para evitar contaminação de culturas convencionais.A maioria dos métodos convencionais utilizados para a detecção dos transgênicos são laboriosos,caros.Então o objetivo do trabalho é desenvolver uma metodologia rápida e não destrutiva, baseada na espectroscopia NIR,para a distinção entre sementes transgênicas e convencionais.Assim foram coletadas 20 amostras de sementes convencionais e transgênicas.Os espectros NIR das amostras foram registrados e técnicas de reconhecimento de padrões foram aplicadas.Foi observada uma tendência de separação os modelos de SIMCA e PLS-DA apresentaram classificaçãode 100%,a um nível de confiança de 95%.
Palavras chaves
Metodologias Alternativas; Algodoeiro; Quimiometria
Introdução
Uma problemática envolvendo os algodoeiros são os danos causados por insetos e pragas, deste modo o melhoramento do algodão é de suma importância e essa melhoria está sendo feita utilizando principalmente tecnologias de engenharia genética (Baohang, 2019), a partir das quais pode-se obter uma variedade da cultivar com maior teor de proteínas e óleos das sementes (Wilkins, 2000), com maior produtividade e resistentes a insetos (Bolek, 2005). As culturas transgênicas se consolidaram como uma tecnologia rápida na agricultura moderna (ISAAA,2018), no entanto para que um organismo transgênico seja liberado para comercialização o mesmo deve passar por rigorosos testes (BRASIL, 2005) por instituições públicas e privadas credenciadas junto aos órgãos reguladores. As metodologias mais comuns para detecção dos transgênicos são caracterizados pela presença de um ou mais segmentos de DNA exógenos, podendo ou não acomodar a expressão de novas proteínas, esses são os chamados métodos convencionais, que são caros, onerosos e destrutivos (PETIT et al., 2003). Outras formas de detecção e quantificação de OGMs vem sendo desenvolvidos, as novas metodologias têm maior sensibilidade, confiabilidade, custos mais baixos, com certa rapidez quando comparadas aos métodos convencionais mostrados anteriormente. Dentre esses novos métodos vem se destacando a cromatografia, a espectrometria de massa e a espectrometria no infravermelho próximo (OBEID et al., 2004). Os espectros NIR contém informação sobre a composição química da amostra, como também relacionam a informação física da mesma (METROHM, 2013). As análises de cultivares transgênicas utilizando o NIR é baseada na absorção espectral de ligações moleculares que estão relacionadas às mudanças fenotípicas (nível de expressão) causadas por mudanças genotípicas. Assim a espectroscopia NIR é uma ferramenta útil para a identificação de transgênicos, pois possui como caracteristicas ser um método que não necessita da preparação da amostra, o tempo e o custo de análise são reduzidos, pode ter a caracterização simultânea de múltiplos componentees influenciados pelo genótipo, viabilidade de se fazer a análise online, é uma técnica não destrutiva e não invasiva (HAI-FENG et al, 2012). O NIR apresenta algumas desvantagens, como ter espectros sobrepostos e complexos (MAGALHÃES, 2014), que podem ser resolvidas pela utilização de técnicas quimiométricas (PEREIRA, 2011). Diante do exposto o presente trabalho visa desenvolver uma metodologia simples e precisa capaz de distinguir entre os genótipos convencionais e transgênicos de algodão utilizando espectroscopia NIR e métodos de reconhecimento de padrões como PCA (Principal Component Analysis), SIMCA (Soft Independent Modeling Class Analogy) e PLS-DA (Análise Discriminante de Mínimos Quadrados Parciais).
Material e métodos
Os experimentos foram realizados no Laboratório Avançado de Tecnologia Química(LATECQ) localizada na Embrapa Algodão, em Campina Grande-PB.O tratamento estatístico de dados foi realizado nas dependências da Universidade Estadual da Paraíba–UEPB. As amostras das sementes de cultivares de algodões transgênicos e convencionais foram cedidos pela Embrapa Algodão, sendo uma espécie de algodão transgênica denominada (BRS 368 RF) e uma espécie de algodão convencional denominada (BRS Aroeira). Foram coletadas 10 amostras de cada uma destas totalizando uma quantidade de 20 amostras. Após a coleta das sementes na forma de plumas de algodão, foi realizada a limpeza da semente de forma a deixar a semente sem resíduo de fibra. A cultivar transgênica, BRS 368 RF, utilizada neste trabalho foi originada de um cruzamento biparental,realizado pela Embrapa Algodão, obtido a partir de um programa de melhoramento de retrocruzamento para incorporação do evento comercial MON 88913 que é conhecido como Roundup Ready® Flex(RF) para conferir resistencia ao herbicida glifosato em germoplasma de algodão. Esse cruzamento se deu com a cultivar convencional BRS 286, proveniente da Embrapa, que foi utilizada como pai recorrente e a cultivar de algodão de alto rendimento Sure Grow 125 utilizada como doadora de características, proveniente da Monsanto(BOWMAN et al, 2006;BARROSO,2017). A cultivar transgênica BRS 368 RF que tem como características ser de porte baixo, tem o ciclo de médio a precoce, com rendimento de fibra de 40% e potencial produtivo entre 4.200 e 4.500 kg/ha e é resistente ao herbicida glifosato. Por esse motivo, a mesma oferece maior flexibilidade no controle de plantas daninhas, permitindo a aplicação em qualquer fase do desenvolvimento da cultura sem necessidade de pulverizações com herbicidas não seletivos em jato dirigido (EMBRAPA,2017). A cultivar convencional, BRS Aroeira, produz em média 3841 kg/ha, dentre as cultivares plantadas no Brasil, esta é a que dispõe de maior teor de óleo na semente (25% a 27%) comparada com as outras que possuem em média 14% de óleo, possui fibras mais finas, com maior comprimento, representam uma alternativa para a produção de custo baixo e possui resistência múltipla a doenças como ramulose, viroses, além de tolerância a bacteriose, manchas de ramularia, entre outros (FREIRE, 2002). As medidas espectrais de reflectância difusa foram realizadas utilizando um espectrômetro VIS/NIR modelo XDS Analyser (Foss Analytical,Hogans,Sweden), abrangendo a região do visível e infravermelho próximo (400-2500 nm),afim de obter o máximo de informação relacionada a comprimentos de onda que tivessem relação com a ocorrência ou não da transgenia nas sementes. Os espectros foram registrados com resolução de 0,5 nm e 32 scans. Para o registro dos espectros foi utilizado o acessório de reflectância. Os modelos de reconhecimento de padrão (PCA,SIMCA e PLS-DA) foram construídos utilizado o pacote PLS tollbox(versão 3.5) para uso em MATLABTM.
Resultado e discussão
Os espectros NIR brutos das amostras convencional e transgênica são
mostrados na figura 1A. Pode-se observar que não é possível encontrar
visualmente características espectrais que possam diferenciar as amostras
transgênicas e convencionais a partir dos mesmos. Também podemos observar na
Figura 1a que os espectros brutos apresentam deslocamento de linha de base e
um espalhamento no sinal espectral devido à natureza da amostra. Deste modo,
alguns pré-processamentos espectrais foram aplicados a fim de remover estas
características físicas relacionadas aos aspectos físicos das amostras. Os
pré-processamentos testados foram baseline offset, SNV, suavização com
janela de 9 pontos e polinômio Savitzky Golay de segunda ordem mostradas
respectivamente nas figuras 1B, 1C e 1D.
A análise em componentes principais (PCA) foi aplicada a fim de realizar uma
análise exploratória dos dados brutos e pré-processados. Tanto a PCA dos
dados brutos quanto dos pré-processados apresentou uma tendência de
separação entre as sementes convencionais e transgênicas, como mostra a
figura 2. De modo geral, as amostras transgênicas se localizaram nos escores
negativos de PC1, enquanto as amostras convencionais se localizaram nos
escores positivos de PC1. Também pode-se observar que as amostras
convencionais apresentam uma menor variabilidade espectral, apresentando uma
menor distância intraclasse. Já as amostras transgênicas apresentam uma
maior variabilidade e consequentemente uma maior distância intraclasse. Os
dados tratados com SNV + baseline offset + suavização foram os que
apresentaram a melhor distinção entre as duas classes. E aqueles que
apresentaram a menor distinção entre as classes. Estes resultados indicam
que os dados em questão forneceram modelos de reconhecimento de padrões
supervisionados com uma boa capacidade preditiva.
Visando o desenvolvimento de uma metodologia alternativa aos métodos
convencionais que possa ser utilizada para distinguir sementes convencionais
e transgênicas, foram construídos modelos de reconhecimento de padrões
supervisionados utilizando os métodos SIMCA e PLS-DA. Levando em conta os
resultados da análise exploratória os modelos SIMCA e PLS-DA foram
construídos utilizando os dados tratados com SNV + Baseline offset +
suavização com janela de 9 pontos e polinômio Savitzky Golay com polinômio
de segunda ordem. Para a construção dos modelos 6 amostras de cada classe
foram utilizadas para a etapa de treinamento e 4 amostras de cada classe na
etapa de teste. Deste modo, o conjunto de treinamento foi composto de 12
amostras e o conjunto de teste foi composto por 8 amostras. Visando a
representatividade e reprodutibilidade da amostragem, as amostras dos
conjuntos de treinamento e teste foram selecionadas com o auxílio do
algoritmo Kennard Stone (KENNARD, 1969).
Para a construção do modelo SIMCA, foi calculada uma PCA para a classe
convencional e uma para a classe transgênica, separadamente, utilizando-se
as 6 amostras de treinamento para cada classe, utilizando 4 componentes
principais. Em seguida o modelo SIMCA foi testado para as amostras de 8
amostras de teste, 4 convencionais (amostras de 1 a 4) e 4 OGM (amostras de
5 a 8). A Tabela 1 mostra o resultado da etapa de teste do modelo SIMCA. Ela
mostra a classificação de cada amostra nas classes. Como pode ser observado
foi alcançado 100% de classificação correta, a um nível de confiança de 95%,
não sendo apresentados resultados falsos positivos ou falsos negativos.
Variáveis que tem poder de modelagem próximo a unidade são importantes para
o modelo, já aquelas com poder de modelagem inferior a 0.3 tem pouca
importância para o modelo. De acordo com o estudo relativo ao poder de
modelagem (PM) pode-se observar que a maioria das variáveis espectrais tem
PM > 0.8 indicando que são importantes para a construção do modelo. Em
relação ao poder discriminatório (PD), que representa o quanto cada variável
contribui para a separação das classes, pode-se observar que a variável
2.432 nm possui um alto valor de poder discriminatório. O comprimento de
onda 2.432 nm pode ser atribuído ao grupo funcional aril (C-H aromático C-H)
(WORKMAN, 2007). A região de 2.400 a 2.493 nm apresenta variáveis com alto
valor de PD, dentre as variáveis desta região se encontra o comprimento de
onda 2445 nm (seta vermelha) que pode ser atribuído ao grupo funcional
CONH2, especialmente devido a ligação hidrogênio C=O com N-H do peptídeo
denominada estrutura alfa-hélice (WORKMAN, 2007). A α-hélice está presente
na estrutura secundária dos níveis de organização das proteínas e se
assemelha a uma escada em espiral. Nesta estrutura o esqueleto de
polipeptídio está estreitamente enrolado ao longo do maior eixo da molécula
e os grupos R dos resíduos de aminoácido projetam-se para fora do esqueleto
helicoidal. A estabilização se dá pela presença das ligações de hidrogênio
entre os grupamentos NH e CO da cadeia principal. O grupamento CO de cada
aminoácido forma ponte de hidrogênio com o grupamento NH do aminoácido que
está situado a quatro unidades adiante na sequência linear, sendo que todos
os grupamentos NH e CO formam pontes de hidrogênio (WORKMAN, 2007).
O modelo PLS-DA foi construído utilizando as amostras de treinamento
selecionadas pelo algoritmo Kennard Stone. Na etapa de calibração foi obtida
uma especificidade e sensibilidade igual a 1, já na etapa de validação
interna (em vermelho) foi obtida uma especificidade de 0,97. O limiar de
separação entre as classes foi de 0,37. Na etapa de predição, foi obtida uma
especificidade de 1 e uma sensibilidade de 0,92, a separação das classes foi
alcançada a um limiar de 0,43. A validação do modelo PLS-DA, construído foi
realizada utilizando as amostras do conjunto de teste selecionadas pelo
algoritmo Kennard Stone. Pode-se observar a separação entre as duas classes,
tanto na etapa de treinamento quanto na etapa de teste.
Figura 1 - Espectros brutos (A) e pré-processados com (B) Baseline offset,
(C) SNV + baseline offset e (D) SNV + baseline offset + suavização com
janela de 9 pontos e polinômio Savitizky Golay de 2ª ordem.
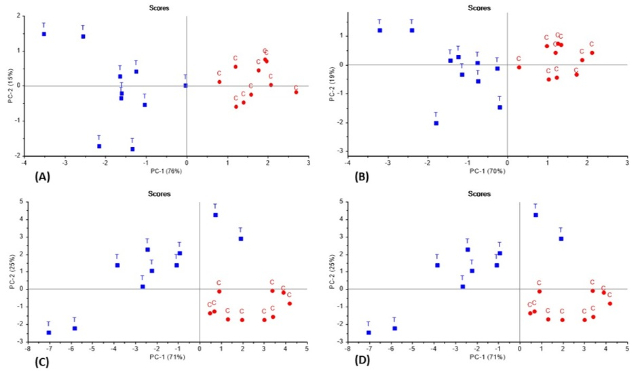
os T representam as amostras transgênicas (em azul) e C as amostras convencionais (em vermelho)
(A) Gráfico de influência e (B) elipse de confiança
Conclusões
A distinção entre cultivares convencionais e geneticamente modificadas é uma necessidade tanto para os centros de pesquisa, onde são desenvolvidos, quanto para os órgãos de fiscalização e os consumidores em geral. Deste modo, o desenvolvimento de metodologias alternativas aos métodos convencionais para identificação de eventos transgênicos que sejam rápidos, confiáveis e menos onerosos economicamente é uma realidade. A espectroscopia NIR mostrou-se uma boa alternativa aos métodos convencionais por ser uma técnica não destrutiva, rápida e que não necessita do consumo de reagentes químicos. Para o desenvolvimento da metodologia foram coletadas 20 amostras de sementes de algodão, sendo 10 convencionais e 10 OGM. O método de reconhecimento de padrões não supervisionados, a partir da técnica de Análise de Componentes Principais, realizou uma análise exploratória dos dados onde foi observada uma tendência de separação entre as duas classes de amostras. Os modelos de reconhecimento de padrão supervisionado, SIMCA e PLS-DA, foram desenvolvidos com os dados pré processados utilizando a correção de linha de base, associada ao SNV e a suavização utilizando janela de 9 pontos e polinômio Savitizky Golay de segunda ordem. Tanto as técnicas não supervisionadas (PCA) quanto as técnicas supervisionadas (SIMCA e PLS-DA) mostraram um bom desempenho, sendo capazes de distinguir entre as duas classes estudadas e alcançando uma taxa de classificação correta de 100% a um nível de confiança de 95%.
Agradecimentos
CAPES, CNPQ, EMBRAPA algodão, NUQAAPE
Referências
BAOHANG ZHANG (ed), Transgenic Cotton: Methods and Protocols, Methods in Molecular Biology, Springer Science + Business Media, part of Springer Nature, v.1902, 2019.
BARROSO, P.A.V; SUASSUNA, N.D; PEDROSA, M.B; MORELLO, C.L; FILHO, J.L.daS; LAMAS,F.M;BOGIANI, J.C; BRS 368RF: A glyphosate tolerant, midseason upland cotton cultivar for Northeast and North Brazilian cerrado. Crop Breeding and Applied Biotechnology, v. 17, p. 399-402, 2017
BOLEK, Y. K. M. EL-ZIK, A. E. PEPPER et al., “Mapping of verticillium wilt resistance genes in cotton,” Plant Science, vol.168, no. 6, pp. 1581–1590, 2005.
BOWMAN, D.T; GUTIERREZ, O.A; PERCY, R.G; CALHOUN, D.S; MAY, O.L. Pedigrees of upland and pima cotton cultivars released between 1970 and 2005. Mississippi Agricultural & Forestry Experiment Station, Starkville, 58p. (Bulletin 1155), 2006
BRASIL, Lei 11.105, 24 de março de 2005. Disponível em: http://www.planalto.gov.br/ccivil_03/_ato2004-2006/2005/lei/l11105.htm
EMBRAPA, Algodão resistente a herbicida será apresentada a produtores cearenses, 2017.
FREIRE, E.C; MORRELO,C de L; MATOS, J.P; SENHORELO, W.L,P. Desempenho comercial das cultivares BRS Aroeira e BRS Ipê no Estado de Goiás- Safra 2001/2002, 2002.
HAI-FENG, C; ZI-HONG, Y; LU-XU; XIAN-SHU, F; CUI-WEN, F; XIAO-PING, Y. Automatic and Rapid Discrimination of Cotton Genotypes by Near Infrared Spectroscopy and Chemometrics. Journal of Analytical Methods in Chemistry, China, 2012.
ISAAA: situação global dos cultivos transgênicos em 2017. Resumo executivo, 2018.
KENNARD, R. W.; STONE, L. A. Computer Aided Design of Experiments, Technometrics, v.11, n. 1, p. 137 – 148, Feb. 1969.
MAGALHÃES, D.F. Aplicação da espectroscopia de infravermelho próximo na monitorização de processos farmacêuticos. Lisboa, 2014.
METROHM. NIR Spectroscopy. A guide to near-infrared spectroscopic analysis of industrial manufacturing processes. Herisau, Suiça: Metrohm, 2013.
OBEID, P.J. et al. Rapid analysis of genetically modified organisms by in-house developed capillary electrophoresis chip and laser-induced fluorescence system. Electrophoresis, v.25, p.922-930, 2004.
PEREIRA, C. M. A Espectroscopia NIR no Controlo de Qualidade de Solventes Industriais. Dissertação para obtenção do Grau de Mestre em Engenharia Química. Lisboa:Universidade Técnica de Lisboa - Instituto Superior Técnico, 2011.
PETIT, L. et al. Screening of genetically modified organisms and specific detection of Bt176 maize in flours and starches by PCR-enzyme linked immunosorbent assay, European Food Research and Technology, v.217, p.83-89, 2003.
Wilkins, T. A. K. Rajasekaran, and D. M. Anderson, “Cotton biotechnology,” Critical Reviews in Plant Sciences, vol. 19, no. 6, pp. 511–550, 2000.
WORKMAN, J.J; WEYER, L. Practical Guide to Interpretive Near-Infrared Spectroscopy. CRC Press, 2007.









