Avaliação do perfil químico do fungo Colletotrichum acutatum na presença de moduladores epigenéticos
ISBN 978-85-85905-25-5
Área
Produtos Naturais
Autores
Faria Gomes, W. (UNIVERSIDADE FEDERAL DE SERGIPE) ; Ferreira Almeida, N. (UNIVERSIDADE FEDERAL DO TOCANTINS) ; dos Santos Niculau, E. (UNIVERSIDADE FEDERAL DO TOCANTINS) ; Fátima das Graças Fernandes da Silva, M. (UNIVERSIDADE FEDERAL DE SÃO CARLOS)
Resumo
Uma das formas de estimular a produção de metabólitos secundários por um microrganismo é cultivá-lo em diferentes condições das oferecidas em laboratório. Com a utilização de moduladores epigenéticos alguns genes podem ser silenciados, causando a ativação de outros. Assim, o objetivo desse trabalho é avaliar a produção de metabólitos secundários pelo fungo Colletotrichum acutatum na presença de moduladores epigenéticos. O fungo foi cultivado em meio BD na presença de alguns moduladores epigenéticos para avaliação do modulador mais viável para cultivo em larga escala. A avaliação foi feita por CCD. Após fracionamento por CC e análises espectroscópicas, foram isolados e identificados os metabólitos 2,5-dihidroximetilfurano, triptofol, indol-3-aldeído e indol.
Palavras chaves
Colletotrichum acutatum; Metabólitos secundários; Epigenética
Introdução
A epigenética consiste no estudo das interações das histonas com o DNA, produzindo fenótipos sem causar alterações na sequência primária do DNA (CHERBLANC et al, 2013). Essas interações podem ser introduzidas por metilação do nucleotídeo citosina ou por reações que ocorrem nas proteínas nucleossomais denominadas histonas (DOBOSY e SELKER, 2001). A metilação do DNA é um processo realizado pela enzima DNA metiltransferase, pelo qual catalisa a transferência de grupos metila desde S-adenosil metionina (SAM) à posição C-5 do nucleotídeo citosina. Já as histonas, que são proteínas nucleares associadas a cromatina, cumprem a função de servir de suporte para a compactação da molécula de DNA, diminuindo o seu tamanho e permitindo o maior controle dos genes por parte da célula (BIEL et al, 2005). Assim, algumas reações que ocorrem nas histonas como a acetilação e deacetilação alteram o empacotamento da molécula de DNA com a cromatina (CICHEWICZ, 2010). As modificações sobre as histonas acontecem sobre os resíduos N-terminal das proteínas. Há uma interação eletrostática entre as histonas (carregadas positivamente devido aos grupos amino existente na lisina e arginina) e o grupo fosfato do DNA (carregado negativamente) (CHERBLANC et al, 2013). A acetilação, realizada pelas enzimas histona acetiltransferases (HATs) neutraliza a carga positiva das histonas convertendo as aminas em amidas (reduzindo a capacidade de interação das histonas com o DNA). Esta redução da interação permite a expansão da cromatina e consequentemente a diminuição da transcrição genética. Por outro lado, a deacetilação de histonas é realizada pelas enzimas histonadeacetilases (HDACs) e são responsáveis por retirar esse grupo acetila. Essas enzimas HDAC tem um papel muito importante da remodelagem da cromatina (GRAESSLE et al, 2001). O descobrimento de moléculas que atuam como inibidores de HDACs e seus derivados sintéticos tem permitido aprofundar o conhecimento das funções dessas enzimas (CHERBLANC et al, 2013). No geral, a hipoacetilação de histonas está associada com silenciamento de genes, enquanto que hiperacetilação é mais comumente associada com ativação gênica (TRIBUS et al, 2010). A metilação de histonas também é um processo que comumente ocorre e seu efeito na cromatina depende da quantidade de metilação e de qual aminoácido sofreu a modificação, podendo resultar em ativação ou silenciamento gênico (LAWRENCE et al, 2004). O motivo pelo qual apenas alguns clusters de genes são afetados pelas enzimas HDACs e DNA-metiltransferases é algo que ainda não foi descoberto (LEE et al, 2009). A regulação da produção de metabólitos mediada por essas enzimas poderia ser um fenômeno disseminado em fungos filamentosos, que evoluiu como uma ferramenta para permitir a produção de metabólitos secundários sob algumas condições específicas. Consequentemente, a regulação epigenética do metabolismo secundário é uma eficiente forma dos fungos assegurarem que a produção energeticamente desfavorável de determinadas moléculas só ocorrerá quando for vantajosa (SHWAB et al, 2007). Assim, o tratamento de fungos com inibidores químicos das enzimas HDAC e DNA-metiltransferases pode promover o aumento da produção de metabólitos de interesse, além de auxiliar na identificação de novos produtos naturais que provavelmente não são detectados sob condições normais de cultivo. O uso de moduladores epigenéticos tem permitido a descoberta de novos metabolitos críticos em diversas espécies de fungos (WILLIAMS et al., 2008). Colletotrichum acutatum, causador da Podridão Floral dos Citros (PFC), é uma das espécies mais frequentemente descritas do gênero e causa doenças comumente conhecidas como antracnose em inúmeras plantas hospedeiras em todo o mundo. Assim, o principal objetivo desse trabalho é estimular a produção de novos metabólitos secundários através de moduladores epigenéticos, aumentando o conhecimento do perfil químico desse microrganismo.
Material e métodos
Foram testados alguns moduladores epigenéticos como 5-azacitidina, SAHA, butirato de sódio, ácido valproico, e incorporados ao cultivo de C. acutatum em BD. Os moduladores foram incorporados ao meio de cultivo nas concentrações (50 μM, 100 μM e 200 μM) e após 20 dias de cultivo sob agitação foram extraídos com AcOEt, e o extrato foi concentrado em um evaporador rotatório à pressão reduzida (MUKHERJEE et al, 2019). Os extratos obtidos foram analizados por CCD e CLAE-UV, onde foram escolhidos o modulador e a concentração mais viável para se trabalhar em uma escala ampliada. O extrato obtido foi fracionado por CC utilizando-se sílica gel como fase estacionária e Hex:AcOEt como eluentes. Após as devidas comparações por CCD foram reunidas as frações F1 (20% AcOEt), F2 (50% AcOEt), F3 (80% AcOEt) e F4 (100% AcOEt). As frações F2 e F3 foram purificadas por CC e após análises por RMN 1H e 13C e EM foram obtidas as substâncias.
Resultado e discussão
Dos moduladores epigenéticos utilizados, o butirato de sódio teve uma melhor
resposta com relação a todos os outros utilizados, sendo escolhido para
cultivo do fungo em larga escala. Os genes que codificam para o DMAT em
espécies do gênero Colletotrichum evidenciam a produção de alcaloides
indólicos derivados do triptofano. Em estudos químicos realizados
anteriormente com o fungo C. acutatum, não foi observada a produção destes
compostos. Com a utilização de um modulador epigenético que inibe a
deacetilação nas histonas, pôde-se observar a produção destes alcaloides. O
fato do modulador inibir a reação de deacetilação nas histonas provavelmente
faz com que ocorra uma hiperacetilação, transformando a histona em uma amida,
neutralizando a carga carregada positivamente. Com isso, não haverá uma
atração eletrostática por parte da histona com os grupos fosfato do DNA
carregados positivamente acarretando na desmodelagem na cromatina.
Assim, alguns genes ficam inativos já que não participam da divisão celular.
Acredita-se que o silenciamento de alguns genes fez com que houvesse uma
produção demasiada do furanoide 2,5-dihidroximetilfurano (1) além da produção
de alguns compostos indólicos triptofol (2), indol-3-aldeído (3) e indol (4)
(Figura 1). A diferença na produção destes compostos foi visualizada por CCD
em fase normal (sílica gel) com hexano e acetato de etila como eluientes (80%
AceOEt) (Figura 2).
Em alguns trabalhos reportados na literatura, o tratamento de diferentes
fungos com inibidores da enzima HDAC resultou na produção de vários
metabólitos que não haviam sido identificados nas espécies não tratadas (SHWAB
et al, 2008; FISCH et al, 2009). O fungo Chaetomium indicum, após tratamento
com ácido suberoidroxâmico, teve a produção de alguns policetídeos induzidos,
e acabou levando ao isolamento seis novos compostos, chaetofenol A-F (ASAI et
al, 2013). A produção desses compostos também foi detectada no controle sem
tratamento, mas foi estimulada em diferentes graus após o tratamento com o
inibidor de HDAC, sendo que apenas chaetofenol A, B, C e E parecem realmente
ter sido induzidos devido ao tratamento. Os autores ainda verificaram um maior
grau de acetilação em genes possivelmente envolvidos na biossíntese desses
compostos.
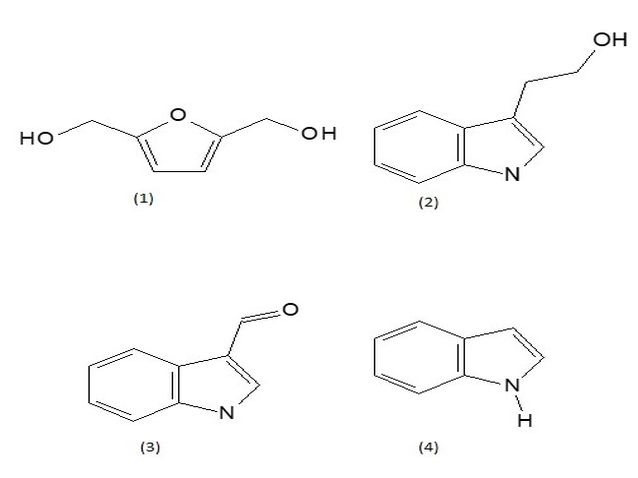
Estruturas químicas dos compostos produzidos por C. acutatum após modulação epigenética.
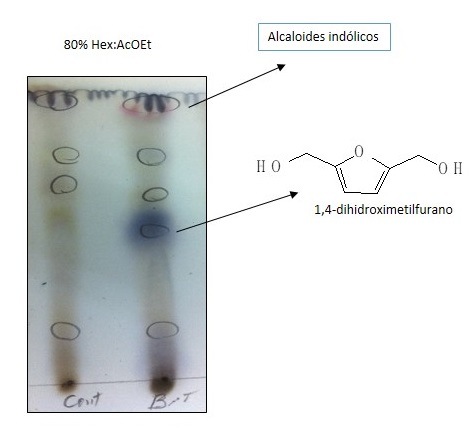
Cromatografia em camada delgada da fração contendo os compostos produzidos por C. acutatum após modulação epigenética.
Conclusões
Na tentativa de explorar o metabolismo de C. acutatum foram incorporados alguns moduladores epigenéticos no cultivo deste fungo podendo ser uma estratégia para este produzir metabolitos diferentes daqueles observados em condições normais de cultivo. Este capítulo mostra o primeiro relato de estudos epigenéticos com espécies do gênero Colletotrichum. Estes estudos mostraram que há uma modificação epigenética nas histonas quando se utiliza o modulador inibidor da deacetilase butirato de sódio, onde foi possível observar uma maior produção do furanoide 2,5-dihidroximetilfurano. Estes estudos também mostraram a capacidade de espécies deste gênero em produzir alcaloides indólicos produzidos por DMATS. Os compostos indólicos triptofol, indol-3-aldeído e indol foram isolados e identificados.
Agradecimentos
Ao CNPq-INCT: 573742/2008-1; FAPESP-INCT: 08/57859-5 e CAPES pelos auxílio Financeiro
Referências
ASAI, T.; YAMAMOTO, T.; SHIRATA, N.; TANIGUCHI, T.; MONDE, K.; FUJII, I.; GOMI, K.; OSHIMA, Y. Structurally diverse chaetophenol productions induced by chemically mediated epigenetic manipulation of fungal gene expression. Organic Letters, 15, 3346, 2013.
BIEL, M., WASCHOLOWSKI, V.; GIANNIS, A. Epigenetics - An epicenter of gene regulation: Histones and histone-modifying enzymes. Angewandte Chemie, 44, 3186, 2005.
CICHEWICZ, R. H. Epigenome manipulation as a pathway to new natural product scaffolds and their congeners. Natural Product Reports, 27, 11, 2010.
CHERBLANC, F. L., DAVIDSON, R. W., DI FRUSCIA, P., SRIMONGKOLPITHAK, N.; FUCHTER, M. J. Perspectives on natural product epigenetic modulators in chemical biology and medicine. Natural Product Reports, 30, 605, 2013.
DOBOSY, J. R; SELKER, E. U. Emerging connections between DNA methylation and histone acetylation. Cellular and Molecular Life Sciences, 58, 721, 2001.
FISCH, K. M.; GILLASPY, A. F.; GIPSON, M.; HENRIKSON, J. C.; HOOVER, A. R.; JACKSON, L.; NAJAR, F. Z.; WAGELE, H.; CICHEWICZ, R. H. Chemical induction of silent biosynthetic pathway transcription in Aspergillus niger. Journal of Industrial Microbiology and Biotechnology, 36, 1199, 2009.
GRAESSLE, S.; LOIDL, P.; BROSCH, G. Histone acetylation: plants and fungi as model systems for the investigation of histone deacetylases. Cellular and Molecular Life Sciences, 58, 704, 2001.
LAWRENCE, R. J.; EARLEY, K.; PONTES, O.; SILVA, M.; CHEN, Z. J.; NEVES, N.; VIEGAS, W.; PIKAARD, C. S. A concerted DNA methylation/histone methylation switch regulates rRNA gene dosage control and nucleolar dominance. Molecular Cell, 13, 599, 2004.
LEE, I.; OHA, J. H.; SHWAB, E. K.; DAGENAIS, T. R. T; ANDES, D.; KELLER, N. P. HdaA, a class 2 histone deacetylase of Aspergillus fumigatus, affects germination and secondary metabolite production. Fungal Genetics and Biology, 46, 782, 2009.
MUKHERJEE, K. M.; DUBOVSKIY, I.; GRIZANOVA, E.; LEHMANN, R.; VILCINSKAS, A. Epigenetic mechanisms mediate the experimental evolution of resistance against parasitic fungi in the greater wax moth Galleria mellonella. Scientific Reports, 9, 1-11, 2019.
SHWAB, E. K.; KELLER, N. P. Regulation of secondary metabolite production in filamentous ascomicetes. Mycological Research, 112, 225, 2008.
WILLIAMS, R. B., HENRIKSON, J. C., HOOVER, A. R., Lee, A. E.; CICHEWICZ, R. H. Epigenetic remodeling of the fungal secondary metabolome. Organic and Biomolecular Chemistry, 6, 1895, 2008.









