Autores
Sandoval-salazar, I.A. (CENTRO DE ENERGIA NUCLEAR NA AGRICULTURA, USP) ; Aratboni, H.A. (CENTRO DE ENERGIA NUCLEAR NA AGRICULTURA, USP) ; Rafei, N. (CENTRO DE ENERGIA NUCLEAR NA AGRICULTURA, USP) ; Winck, F.V. (CENTRO DE ENERGIA NUCLEAR NA AGRICULTURA, USP)
Resumo
Atualmente há um interesse em tecnologias de modulação da expressão genica em
microalgas com foco em aplicações biotecnológicas em cepas não-transgênicas de
interesse comercial. A integração da nanotecnologia com a biologia molecular e de
sistemas se apresenta como uma alternativa importante para o desenvolvimento de
novas formas de uso e entrega intracelular de oligonucleotídeos antisense (OAS) em
microalgas. No presente trabalho, nanopartículas de ouro foram utilizadas como
carreadoras de OAS para silenciar transitoriamente o gene NAGS da microalga modelo
C. reinhardtii. Nos diferentes tratamentos observou-se internalização do complexo
OAS-AuNPs, com indução química. A liberação dos OAS foi feita por processo
fototérmico resultando na diminuição da expressão genica do gene de interesse.
Palavras chaves
Nanopartículas; Genes; Silenciamento
Introdução
As nanopartículas de ouro (AuNPs) têm sido usadas como ferramenta molecular para
entrega intracelular não viral de biomoléculas, tais como os oligonucleotídeos
antisense (OAS) de DNA. Tais OAS podem formar um complexo com as nanopartículas
de ouro (OAS-AuNPs) e um vez liberado o antisense no interior celular podem
atuar na redução ou silenciamento da expressão gênica intracelular, modulando a
expressão de proteínas específicas em células vegetais (PON ET AL., 2010; ROSSI,
2006). A partir das informações da sequência do RNA mensageiro do gene alvo,
faz-se o desenho do oligonucleotídeo antisense que é sintetizado complementar a
essa sequência alvo. O OAS uma vez entregue e liberado na célula alvo pode
interagir com o transcrito gênico do gene alvo e inibir sua tradução ou induzir
sua degradação (LEBEDEVA & STEIN, 2001). No entanto, existem limitações nos
processos de entrega das moléculas de OAS à célula, relacionados a ligação à
membrana celular e internalização celular do complexo OAS-AuNPs causadas pelas
características inerentes das paredes celulares vegetais e sua complexa rede de
biopolímeros, liberação de OAS ao citosol, ligação a um mRNA particular e
clivagem dos duplexes OAS/mRNA para inibir ou reduzir a expressão da proteína de
interesse. Nesse sense, esforços são necessários para desenvolver diferentes
métodos que permitam a interiorização e transfecção mais eficiente mediada por
nanopartículas em células vegetais (EL-ANEED, 2004; KARINAGA, 2006; ZHANG ET
AL., 2021, ZUHORN, 2007). No presente trabalho, sintetizou-se nanopartículas de
ouro que foram funcionalizadas com oligonucleotídeos sense (contendo grupo Tiol)
e antisense (contendo fluoróforo Cy5) que atuam como um interruptor para o
controle remoto da interferência gênica em células de microalgas. Como gene
candidato ao silenciamento, selecionamos o gene que codifica a enzima N-
Acetilglutamato sintase (NAGS) envolvida com o metabolismo de aminoácidos,
crescimento e síntese lipídica na microalga Chlamydomonas reinhardtii. A
liberação do OAS foi induzida por termogênese com luz LED azul, e a alteração na
transcrição gênica verificada por PCR em tempo real.
Material e métodos
A microalga Chlamydomonas reinhardtii foi cultivada em condições mixotrópicas no
meio de cultura líquido. A síntese de nanopartículas de ouro (AuNPs) foi
realizada pelo método de redução de citrato (KIMLING ET AL., 2006), na qual
utilizou-se uma solução 0.5 mM de cloreto áurico (AuCl3) e uma solução 38.8 mM
de citrato de sódio. O processo de conjugação das AUNPs com os oligonucleotídeos
sense (OS) marcados com tiol e antisense marcados com Cy-5 para o gene do N-
Acetilglutamato sintase (NAGS) foram realizados primeiramente utilizando uma
solução 50 mM de DTT em tampão de fosfato de pH 8.3 e foi misturado com 11 uL de
OS para reduzir as ligações dissulfeto dos oligonucleotídeos e assim atingir a
forma sulfidrila ativa. seguidamente foi adicionado a solução de tampão de
fosfato de pH 6 para realizar a purificação das formas sulfidrila ativadas dos
oligonucleotídeos usando colunas e adicionou-se 40 uL do buffer fosfato salino.
Seguidamente foi misturada com 3 mL de AuNPs e deixou-se em incubação em
agitação constante em escuridade durante 3 horas, dessa forma os foram ligados a
traves do grupo tiol (-SH) na extremidade 3‘. Após a incubação foi adicionado 22
µL da solução com os OAS e foram incubados durante 2 minutos a 80 ºC e a 15
minutos a 65 ºC, finalmente foram incubadas em agitação constante em escuridade
durante 3 horas. A caracterização tanto das nanopartículas como da
funcionalização dos oligonucleotídeos foi realizada através de
espectrofotometria com varredura de absorbância entre 230 nm e 900 nm. Três
métodos de transfecção celular foram testados: Eletroporação a 500 V, 30 ms,
indução quimica utilizando solução de sorbitol 60 mM e um kit comercialmente
disponível (Max Efficiency, Thermo, EUA). finalmente, para verificar a eficácia
dos métodos de transfecção na introdução de OAS, foram analisadas por
microscopia confocal por fluorescência. A liberação dos OAS do complexo OAS-
AuNPs dentro da microalga foi realizado através da indução fototérmica
utilizando LEDs azul até atingir a temperatura de fusão das fitas sense e
antisense (45.5 ºC). As amostras foram incubadas durante 1h e 2h após a
transfecção, e logo submetidas a extração de RNA total usando o RNeasy® Plant
Mini Kit (Qiagen, EUA). As amostras de RNA total foram tratadas com turbo DNaseI
(RNase-free kit, Invitrogen™, USA) e 1ug de RNA total foi usado para a síntese
do cDNA usando kit SuperScript™ IV (Invitrogen™, USA). A quantificação da
expressão gênica foi feita por PCR em tempo real (RT-PCR) utilizando-se o SYBR
Green dye master mix (Thermo, EUA) e sistema CFX384 (Bio-Rad, USA). As reações
de RT-PCR foram realizadas em um volume total de 20 µl, com 10 µl SYBR Green PCR
Master Mix, 1 µl de primer Forward e Reverse (1 µM), 100 ng de cDNA como molde
(LIVAK & SCHMITTGEN, 2001). Os primers para os s gene Ubiquitina ligase (UBQ) e
para região intergênica 13 (IGR13) foram usados como controle interno (genes de
referência e de contaminação de cromossômica) para os ensaios de expressão.
Resultado e discussão
A Figura 1 mostra os resultados da síntese de nanopartículas de ouro e a
conjugação de oligonucleotídeos sense e antisense na superfície das
nanopartículas de ouro.
Pode-se observar que tanto as nanopartículas de ouro quanto os oligonucleotídeos
apresentaram os máximos picos de absorção entre 520-525 nm, indicando que o
preparo das nanopartículas de ouro e conjugação de oligonucleotídeos antisense
foram bem-sucedidos. No entanto, de acordo com as pesquisas realizadas por
Marcel et al., (2021) podem-se apresentar pequenas diferenças entre os espectros
de absorção das nanopartículas de ouro e a conjugação dos oligonucleotídeos
gerando diminuição do máximo pico e picos em comprimentos de onda mais longos,
evidenciando assim processos de agregação dos oligonucleotídeos nas
nanopartículas.
Observou-se com sucesso as células de microalgas após a transfecção com
nanopartículas de ouro e oligonucleotídeos antisense-Nanopartículas de ouro.
Os resultados indicam que os métodos testados permitiram a transfecção das
células, mostrando que as nanopartículas de ouro podem se difundir através da
parede e membrana celular por difusão passiva e podem também ser incorporadas
pela célula por eletroporação. No entanto, a eletroporação afeta e deteriora a
estrutura celular, podendo causar a morte celular, gerando uma diminuição da
concentração celular após o evento de transfecção. A distribuição das
nanopartículas de ouro nas células é um fator importante para identificar o
efeito biológico de acordo com os objetivos da sua aplicação. No entanto, o
tamanho do complexo OAS-AuNPs tem uma relação direta com a sua distribuição
intracelular, no caso do silenciamento gênico com os OAS, devem-se localizar no
citoplasma pelo qual de acordo com diferentes pesquisas AuNPS de 15-20 nm foram
encontradas no citoplasma e não conseguiram atingir uma penetração mais
profunda. Em nosso caso, o tamanho aproximado de 20nm das OAS-AuNPs obtidas nos
permite realizar a função de interesse de inibição citoplasmática de NAGs. As
AuNPs de 15-20 nm podem-se internalizar por difusão direta facilitando a sua
penetração nas células, no entanto, tamanhos superiores de 45 nm precisam ser
internalizadas por endocitose as quais depende de diferentes vias endocíticas
(GONG ET AL., 2015; HUANG ET AL., 2012; OH ET AL., 2011; WANG ET AL., 2010).
Foi realizado com sucesso a entrega dos oligonucleotídeos antisense mediante
processo fototérmico usando LED azul, onde a temperatura nas amostras foi
aumentando 5 ºC por minuto até atingir a temperatura de fusão do gene NAGs a
qual é 45.5 ºC.
Após atingir essa temperatura, as amostras foram incubadas em condições de
cultivo mencionadas e foi realizado a extração do RNA total cuja qualidade foi
analisada através da eletroforese em gel e utilizado finalmente para
a síntese do cDNA.
A Figura 2 apresenta as curvas de amplificação em relação ao número do ciclo de
PCR obtidos pelo PCR em tempo real, foram realizados testes em triplicata para
cada hora de incubação após a liberação do oligonucleotídeo antisense.
Observou-se mudanças nas curvas de amplificação e aumento do valor de ciclo de
quantificação (CQ) de 20,37 ± 0,07 para 22,87 ± 0,1 para as diferentes amostras,
obtidas após 1 hora e 2 horas da liberação do oligonucleotídeo antisense
indicando que houve redução da quantidade de transcritos para NAGS e possível
diminuição da expressão gênica do gene NAGS. Novos testes estão sendo feitos
para análise de tempos maiores de liberação de antisense e respostas de redução
do conteúdo de transcritos do gene alvo.
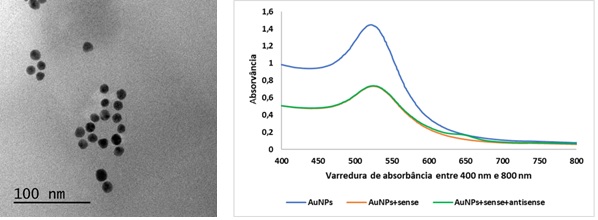
Síntese de AUNPs e conjugado de DNA oligonucleotídeo sense e antisense
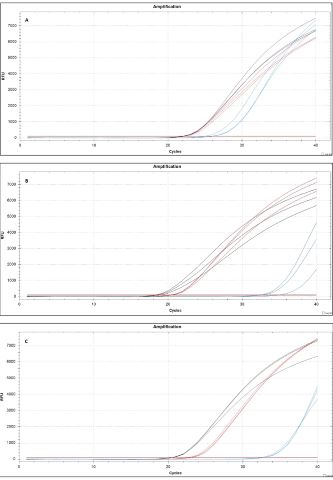
Curvas de amplificação em relação ao número do ciclo de PCR. (A) Controle sem OAS-AuNPs; (B) 1 h da liberação do OAS; (C) 2 h da liberação do OAS.
Conclusões
As nanopartículas de ouro funcionalizadas com oligonucleotídeos sense e antisense
podem ser usados para processos de redução de expressão de genes em microalgas
Chlamydomonas reinhardtii.
As nanopartículas são uma fonte promissora de transporte de moléculas de DNA que,
dependendo da natureza das AuNPs e de sua concentração, não geram problemas de
toxicidade para as microalgas. Para a internalização de OAS-AuNPs em microalgas
não são necessários processos físicos externos, pois por difusão direta conseguem
penetrar na membrana celular, tornando-se um mecanismo promissor para aplicações
biotecnológicas com controle transiente da expressão gênica de genes alvo, sem a
necessidade de produção de cepas geneticamente modificadas ou mutantes
constitutivos. A entrega dos oligonucleotídeos antisense a partir de processo
fototérmico permitiu obtenção de cepas com diminuição da abundância do transcrito
para o gene NAGS 2h após a entrega do oligonucleotídeo antisense.
Agradecimentos
Referências
EL-ANEED, Anas. An overview of current delivery systems in cancer gene therapy. Journal of Controlled Release, v. 94, n. 1, p. 1-14, 2004.
GONG, Ningqiang et al. Effects of the physicochemical properties of gold nanostructures on cellular internalization. Regenerative biomaterials, v. 2, n. 4, p. 273-280, 2015.
HUANG, Keyang et al. Size-dependent localization and penetration of ultrasmall gold nanoparticles in cancer cells, multicellular spheroids, and tumors in vivo. ACS nano, v. 6, n. 5, p. 4483-4493, 2012.
ISAKA, Yoshitaka et al. Oligonucleotidic therapeutics. Expert Opinion on Drug Discovery, v. 3, n. 9, p. 991-996, 2008.
KARINAGA, Ryouji et al. Galactose-PEG dual conjugation of β-(1→ 3)-D-glucan schizophyllan for antisense oligonucleotides delivery to enhance the cellular uptake. Biomaterials, v. 27, n. 8, p. 1626-1635, 2006.
KIMLING, Judith et al. Turkevich method for gold nanoparticle synthesis revisited. The Journal of Physical Chemistry B, v. 110, n. 32, p. 15700-15707, 2006.
LEBEDEVA, Irina; STEIN, C. A. Antisense oligonucleotides: promise and reality. Annual Review of Pharmacology and Toxicology, v. 41, p. 403, 2001.
LIVAK, Kenneth J.; SCHMITTGEN, Thomas D. Analysis of relative gene expression data using real-time quantitative PCR and the 2− ΔΔCT method. methods, v. 25, n. 4, p. 402-408, 2001.
OH, Eunkeu et al. Cellular uptake and fate of PEGylated gold nanoparticles is dependent on both cell-penetration peptides and particle size. ACS nano, v. 5, n. 8, p. 6434-6448, 2011.
POON, Lester et al. Photothermal release of single-stranded DNA from the surface of gold nanoparticles through controlled denaturating and Au− S bond breaking. ACS nano, v. 4, n. 11, p. 6395-6403, 2010.
ROSI, Nathaniel L. et al. Oligonucleotide-modified gold nanoparticles for intracellular gene regulation. Science, v. 312, n. 5776, p. 1027-1030, 2006.
WANG, Sheng-Hann et al. Size-dependent endocytosis of gold nanoparticles studied by three-dimensional mapping of plasmonic scattering images. Journal of nanobiotechnology, v. 8, n. 1, p. 1-13, 2010.
ZHANG, Huan et al. Gold-nanocluster-mediated delivery of siRNA to intact plant cells for efficient gene knockdown. Nano letters, v. 21, n. 13, p. 5859-5866, 2021.
ZUHORN, Inge S.; ENGBERTS, Jan BFN; HOEKSTRA, Dick. Gene delivery by cationic lipid vectors: overcoming cellular barriers. European Biophysics Journal, v. 36, n. 4, p. 349-362, 2007.
















