Autores
Tavares da Silva, K. (IFPA) ; Guedelha Pereira, M.I. (IFPA) ; Souza Alves, R. (IFPA) ; Medeiros da Rocha, E.C. (IFPA) ; Pereira Lopes, A. (UFPA) ; Pires Batista, A. (IFAP) ; Sena Pinheiro, A. (UFPA) ; da Rocha, J.A.P. (IFPA) ; Gemaque de Medeiros, I. (UFPA) ; Amador Trindade, D.H. (IFPA)
Resumo
O Zika vírus tornou-se um problema de saúde pública no Brasil. A contaminação
por esse patógeno ocorre através do mosquito Aedes aegypti acarretando em
complicações neurológicas. A constituição genômica do Zika é de uma cadeia de
RNA positiva. Neste contexto, a proteína não estrutural NS5 é um alvo promissor
para a desativação do mecanismo de replicação viral que desempenha o papel de
metiltransferase e RNA-polimerase. Assim, foram realizados cálculos de
ancoramento molecular, utilizando o software Molegro Virtual Docker 5.5, para um
conjunto de compostos oriundos do banco de moléculas ZINC, objetivando
prospectar potenciais inibidores da enzima. Os resultados obtidos demonstraram
que a metodologia empregada é eficaz e proporcionou a seleção de 2 compostos
promissores em inibir a enzima.
Palavras chaves
Ancoramento molecular; Molegro Virtual Docker; NS5
Introdução
O Zika vírus é um arbovírus, pertencente ao reino Riboviria, família
Flaviviridae (do latim Flavus, amarelo) e gênero Flavivirus (KUNO et al., 1998;
LANCIOTTI et al., 2007). Tal como outros membros do gênero Flavivírus, o Zika
vírus (ZIKV) possui uma estrutura envelopada e contém um genoma de RNA de cadeia
simples de sentido positivo (ssRNA) (LINDENBACH et al., 2013). O genoma do ZIKV
possui regiões não traduzidas de circularização do ácido nucleico em suas
extremidades 5’ e 3’, as quais têm sido relacionadas com fundamental importância
para a replicação viral (ALVAREZ et al., 2005).
A doença causada pelo ZIKV ocorre predominantemente em regiões tropicais do
planeta, atingindo grande parte da América Latina. A transmissão do vírus se dá
através do artrópode hematófago Aedes aegypti que necessita de sangue e água
para sua sobrevivência e reprodução, respectivamente (ICTV, 2016). O ZIKV foi
isolado pela primeira vez em 1947 em Uganda, país do continente africano e no
Brasil, a epidemia foi identificada em 2015, na região nordeste após o uso de
métodos moleculares que mostrou a rápida capacidade de dispersão do vírus,
semelhante ao Chikungunya que possui o mesmo agente transmissor (NUNES; PIMENTA,
2016; MOCELIN, et al., 2020).
Diante do agravamento dos sintomas em gestantes no ano de 2015 no Brasil, houve
um consenso científico de que o Zika vírus está associado a distúrbios
neurológicos raros como microcefalia, condição em que recém-nascidos possuem a
circunferência da cabeça menor do que 33 cm e a síndrome de Guillain-Barré
(SGB), esta última trata-se de uma doença autoimune rara que afeta o sistema
nervoso periférico (COUTARD, 2017). Em 2020, segundo o Ministério da Saúde do
Brasil (BRASIL, 2020), foram registrados aproximadamente 19.492 casos
confirmados de doença febril aguda associada ao vírus da Zika.
Segundo a OPAS - Organização Pan-Americana da Saúde (2018), os sintomas da
doença podem ser diagnosticados de acordo com o grau de virulência, como febre
baixa, erupções cutâneas, dor de cabeça e nas articulações, dor muscular, mal-
estar geral e conjuntivite não purulenta que aparecem entre 2 a 7 dias após a
contaminação pelo mosquito vetor.
Mesmo diante do exposto, apesar de existirem propostas para o desenvolvimento de
vacinas contra o vírus da Zika, ainda não há medicamento cientificamente
comprovado que ofereça segurança no combate ao vírus ou aos sintomas da doença
por ele causado. Assim, pesquisas que visam o tratamento da doença, a partir do
desenvolvimento de medicamentos específicos que possam atuar na desativação do
mecanismo de replicação viral, enfatizando principalmente as enzimas virais, são
de grande relevância. Neste aspecto, é oportuno destacar que um alvo promissor
no desenvolvimento de fármacos contra o Zika é a proteína não estrutural 5 (NS5)
(CANNALIRE, et al., 2017; WU et al., 2020; WAN, et al., 2020).
A NS5 é responsável pela etapa de síntese do RNA genômico, sendo constituída por
dois domínios: polimerase (RdRp) e metiltransferase (MTase) (RAMOS, 2020). Esta
proteína é a mais conservada dos flavivírus (vírus transmitidos por mosquitos ou
carrapatos), conhecida por sua funcionalidade de RNA-polimerase, RNA-dependente
(RpRd) na porção C-terminal e como uma metiltransferase (MTase) na porção N-
terminal responsável pela formação do CAP 5' no RNA viral, possuindo assim, um
papel vital para a replicação do vírus (ICTV, 2016; LIMA, 2017).
Desta forma, neste trabalho foi realizado um ensaio virtual com 2.100 compostos
com atividades contra metiltransferases, oriundos do banco de moléculas ZINC
(STERLING; IRWIN, 2015), para prospectar potenciais inibidores da enzima NS5
metiltransferase do vírus Zika
Material e métodos
Métodos computacionais
Os cálculos de ancoramento molecular foram realizados com as coordenadas
cristalográficas da enzima não estrutural 5 (NS5) metiltransferase do vírus da
Zika (de código PDB 5M5B) (LIM, 2006) recuperadas através do banco de dados de
proteínas PDB (Protein Data Bank).
Devido à estrutura da proteína utilizada ser homodimérica, somente uma cadeia
foi escolhida para a execução dos cálculos. Em seguida, foram removidos os íons
presentes no meio, além de todas as moléculas de água da cadeia escolhida
(PETTERSEN et al., 2004).
Utilizando o software MVD 5.5 foi realizada a redocagem do cofator S-Adenosil-
metionina (SAM) da proteína NS5, para validar a capacidade do programa em
reproduzir os modos de ligação entre o cofator, que ocupa uma cavidade de
ligação estratégica nesse alvo, e a enzima. Em seguida, foram realizados
cálculos de ancoramento molecular com 2.100 moléculas, também no MVD 5.5,
utilizando o conjunto de substâncias selecionadas do ZINC, um banco de dados
gratuito de compostos disponíveis comercialmente para triagem virtual em formato
tridimensional prontos para uso na enzima NS5 do vírus da Zika. Nesta etapa, aos
ligantes e à estrutura do receptor foram adicionados os hidrogênios e atribuídas
as cargas atômicas, usando um módulo de preparação automatizado padrão do
programa.
Para os resíduos de aminoácidos da proteína SER 62, GLY 92, ASP 137 e ASP 152,
localizados na região de alça do sítio ativo, e para o resíduo de VAL 138,
localizado na região de folha-beta da mesma cavidade do sítio ativo, foram
permitidos total flexibilidade durante os cálculos, uma vez que estes são
considerados importantes para a interação do substrato natural com a enzima
(COUTARD,2017). A cavidade de ligação foi computada em um espaço de 13 Å a
partir das coordenadas x = 8,61, y = 43,19 e z = 86,29, com uma resolução de
grade de 0,3 Å. Ademais, foram utilizadas 10 poses para cada molécula ligante.
Os demais parâmetros do programa foram mantidos com os valores padrões durante o
processo (THOMSEN e CHRISTENSEN, 2006; VIJAYAKUMAR e GOWDA, 2013; TORKTAZ et
al., 2013). Nesse contexto, foram selecionados os compostos de acordo com o
valor de energia MolDock e interações entre resíduos de aminoácidos.
Resultado e discussão
Na Figura 1 é apresentada a sobreposição entre as estruturas do cofator, S-
Adenosil-metionina (SAM), obtidas pela cristalografia e pela redocagem, no sítio
ativo da enzima.
Analisando a Figura 1, conclui-se que a metodologia computacional utilizada
gerou uma conformação molecular teórica de posicionamento próximo ao obtido em
nível experimental entre a estrutura ancorada e cristalográfica (LIM, 2006).
Geralmente, a reprodução de um modo de ligação experimental é considerada bem-
sucedida quando o desvio médio quadrático (“Root Mean Square Desviation”, RMSD)
calculado se encontra abaixo de 2,0 Å (RODRIGUES, 2012). No que se refere às
estruturas descritas na imagem, o valor de RMSD é de 1,69379 Å, ratificando a
boa qualidade da redocagem como sugerido por Rodrigues, 2012.
Através dos cálculos de ancoramento molecular, são apresentados os valores de
energia das estruturas moleculares para as 4 melhores conformações.
Os compostos de código ZINC000022448696_3 e ZINC000014879992 possuem valores de
energia -92,460 e -72,429, respectivamente. Estes apresentam propriedades
desfavoráveis para possíveis candidatos à fármacos, uma vez que violam a regra
dos 5 de Lipinski (LIPINSKI, 2004) em virtude de seus elevados valores de massa
molecular. Deste modo, somente os compostos de código ZINC000003604264 (Imid3) e
ZINC000016052277_1 (Doxiciclina) com valores de energia -75,152 e -74,466 são
biomoléculas promissoras, uma vez que apresentam interações com resíduos de
aminoácidos importantes do sítio ativo da enzima e não violam a regra dos 5 de
Lipinski (LIPINSKI, 2004). A regra de Lipinski descreve a biodisponibilidade de
um composto que atingir um alvo biológico a partir de 4 parâmetros múltiplos de
5: Log P maior ou igual a 5; massa Molecular menor ou igual a 500; aceptores de
ligação de Hidrogênio menor ou igual a 10 e doadores de ligação de hidrogênio
menor ou igual a 5.
É importante salientar que a substância IMID3 de fórmula molecular C13H11N3O4
e massa molécula igual a 259,265 é reportada na literatura como um potencial
imunomodulador para o tratamento de condições inflamatórias em humanos, como a
meningite tuberculosa (TBM). Além disso, a substância Doxiciclina de fórmula
molecular C22H24N2O8 e massa molecular igual a 444,44 é também amplamente
utilizada para o tratamento de algumas infecções associadas a bactérias como
cólera, tifo e sífilis e pode ser uma medida efetiva na profilaxia da infecção
por Leptospirose. (TSENOVA, 2002; MONTEIRO, 2003).A doxiciclina é um
antibacteriano bacteriostático utilizado principalmente em infecções
causadas por micro-organismos intracelulares, porém é eficaz também contra
bactérias Gram-negativas e Gram-positivas, clamídias, rickettsias,
micoplasmas e alguns protozoários. Estudos recentes têm demonstrado
outras propriedades desse fármaco além da antibacteriana, tais como
ação anti-inflamatória e de inibição da lipase, colagenase, apoptose e
angiogênese. Desta forma, a doxiciclina pode ser empregada como coadjuvante
em diversas circunstâncias clínicas, com destaque para as afecções
articulares.Mais estudos ainda são necessários acerca das propriedades
não antibacterianas da doxiciclina e sua segurança, porém as perspectivas são
promissoras (D’EL REY-DANTAS, 2018).
Observa-se na Figura 2 que os compostos selecionados apresentam interações
importantes com os resíduos de aminoácidos do sítio ativo da enzima. Os
compostos imid3 (A) e doxiciclina (B) realizam interações de hidrogênio com os
resíduos de aminoácidos, SER 62, GLY 92 e ASP 152, reportados na literatura e
descritos como importantes para promissores inibidores da enzima NS5
metiltransferase do vírus da Zika (COUTARD, 2017).
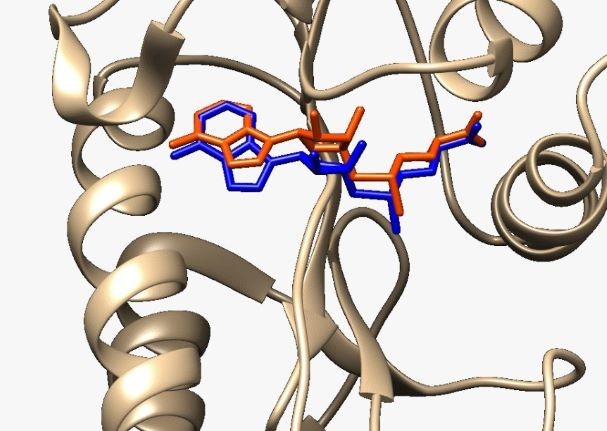
Sobreposição das estruturas obtidas pelo redoque e cristalografia no sítio ativo da NS5 metiltransferase através do cálculo de ancoramento molecular
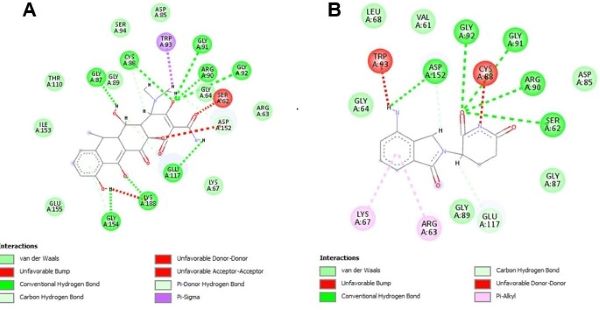
As interações entre os compostos imid3 (A) e doxiciclina (B) com os resíduos de aminoácidos da cavidade de ligação do SAM no sítio ativo da NS5.
Conclusões
O presente trabalho apresenta a realização de cálculos de ancoramento molecular,
tendo por finalidade inferir potenciais inibidores para a enzima NS5, que
desempenha um papel fundamental no mecanismo de replicação do vírus da Zika. A
partir das análises de interações intermoleculares e energias entre o complexo
ligante receptor preditas pelo programa Molegro Virtual Docker, obteve-se o total
de 2 estruturas consideradas satisfatórias para atuar contra a ação da enzima
viral.
Os compostos Imid3 e Doxiciclina mostram-se promissores a partir das interações
com os resíduos de aminoácidos, além da sua eficácia comprovada no tratamento de
infecções provocadas por outros agentes biológicos.
Quanto as propriedades de absorção, distribuição, metabolismo e excreção acerca
dos composto imid3 e doxiciclina ainda serão aprofundadas durante progressão da
pesquisa.
Nessa perspectiva, almeja-se realizar cálculos de Dinâmica Molecular (DM) para
avaliação e aprofundamento de futuros estudos no desenvolvimento de novos
fármacos.
Agradecimentos
O presente trabalho agradece ao Instituto Federal do Pará que contribuiu para a
construção dessa pesquisa com uma infraestrutura de qualidade e equipe de
multiprofissionais essenciais no processo de formação profissional.
Referências
ALVAREZ, Diego. E. et al. Long-range RNA-RNA interactions circularize the dengue virus. Journal of virology, v. 79, n. 11, p. 6631-6643, 2005. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1112138/pdf/2535-04.pdf. Acesso em: 10 out. 2021.
BRASIL. Secretaria de Vigilância em Saúde. Óbito por arboviroses no Brasil. Boletim Epidemiológico Brasília, 2020. Disponível em: https://www.gov.br/saude/pt-br/centrais-de-conteudo/publicacoes/boletins/boletins-epidemiologicos/edicoes/2020/boletim_epidemiologico_svs_48.pdf. Acesso em: 18 jan. 2022.
CANNALIRE, R. et al. Functionalized 2,1-benzothiazine 2,2-dioxides as New Inhibitors of Dengue NS5 RNA-dependent RNA Polymerase. Eur. J. Med. Chem, v. 143, p. 1667-1676, 2017. Disponível em: https://www.sciencedirect.com/science/article/pii/S0223523417308668?casa_token=rRipfJKuuJkAAAAA:KwmuBo7SrR-Auoumitg1jG28XloQNkxsFTzab4zpXAI8nFTtjme64VgWSyzbaIVB8sDcbgpQxQiD. Acesso em: 17 dez. 2021.
COMITÊ INTERNACIONAL DE TAXONOMIA DE VÍRUS. ICTV, 1966-2016. Disponível em: https://talk.ictvonline.org/ictv-reports/ictv_online_report/positive-sense-rna-viruses/w/flaviviridae/360/genus-flavivirus. Acesso em: 09 fev. 2022.
COUTARD, Bruno et al. The Zika virus methyltransferase: Structure and functions for drug design perspectives. Journal of Virology, v. 91, n. 5, 2017. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5309936/. Acesso em: 07 fev. 2022.
D’EL REY-DANTAS, Fernanda Timbó et al Doxiciclina: uma revisão sobre particularidades e utilização clínica na espécie equina. 2018.
KUNO, Goro et al. Phylogeny of the genus flavivirus. Journal of virology. v. 72, n. 1, pág. 73-83, 1998. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC109351/pdf/jv000073.pdf. Acesso em: 02 ago. 2021.
LANCIOTTI, Robert S. et al. Genetic and Serologic Properties of Zika Virus Associated with an Epidemic. Yap State, Micronesia, 2007. Emerging infectious diseases, v. 14. n. 8, p. 1232-2008. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2600394/. Acessado em: 19 ago. 2021.
LIMA, Adonis de Melo. Dinâmica molecular da proteína NS5 do vírus Zika. Projeto de investigação científica do Curso de Biomedicina – Centro Universitário Fibra, Belém, 2017. Disponível em: https://fibrapara.edu.br/resumos-expandidos-investigacao-cientifica/docs/edicao-3/2017/12-DINAMICA-MOLECULAR-DA-PROTEINAN-S5-DO-VIRUS-ZIKA.pdf. Acesso em: 14 ago. 2021.
LINDENBACH, B. D. et al. Chapter 25: Flaviviridae. Fields virology. 6th ed. Philadelphia, PA: Lippincott Williams & Wilkins, p. 712-46, 2013. Disponível em: https://www.worldcat.org/title/fields-virology/oclc/71812790. Acesso em: 03 set. 2021.
LIPINSKI C. A. Lead- and drug-like compounds: the rule-of-five revolution. Drug Discov. Today Technol., p. 337-342, 2004. Disponível em: https://www.sciencedirect.com/science/article/pii/S1740674904000551?casa_token=m5Sib3VShe4AAAAA:ost3FJIXt7wCDF5X3SAG92E4GwCmEYJ8jtpB1yjvEo5QQihO4beSGpXyCx7auXCKadqNEPxn0sG4. Acesso em: 03 jan. 2022.
MOCELIN, Helaine Jacinta Salvador et al. Abordagem profissional em tempos de pandemia: o que aprendemos com o Zika vírus? Interface - comunicação, saúde, educação. ed. Rosamaria Giatti Carneiro. Botucatu, 2020. Disponível em: https://www.scielosp.org/article/icse/2021.v25/e200427/pt/. Acesso em: 15 nov.2021.
MONTEIRO, G. R. G. Efetividade da doxiciclina na profilaxia contra letptospirose. 2003. Dissertação (Mestrado em Bioquímica; Biologia Molecular) - Universidade Federal do Rio Grande do Norte, Natal, 2003. Disponível em: https://repositorio.ufrn.br/handle/123456789/12622. Acessado em: 27 mar. 2022.
NUNES, João; PIMENTA, Denise Nacif. A epidemia de Zika na saúde global. Lua Nova: Revista de cultura e política, São Paulo, 2016. Disponível em: https://www.scielo.br/j/ln/a/gwvcht4Scg5wL3rnyLWcq7y/?format=html&lang=pt. Acesso em: 10 set. 2021.
PETTERSEN, E. F. et al. UCSF Chimera – A visualization system for exploratory research and analysis. J. Comp. Chem. p.1605-1612, 2004. Disponível em: https://www.cgl.ucsf.edu/home/tef/pubs/chimera.pdf. Acesso em: 02 fev. 2022.
RAMOS, Paulo Ricardo Pimenta da Silva. Integração de abordagens computacionais para identificar novos inibidores da proteína NS5 do vírus Zika. 2020, FF- Trabalho de Conclusão de Curso - Faculdade de Farmácia, Universidade Federal de Goiás, 2020. Disponível em: https://repositorio.bc.ufg.br/handle/ri/19413. Acesso em: 08 fev. 2022. (continua errada, atente para o modelo)
RODRIGUES, R. P. et al. Estratégias de Triagem Virtual no Planejamento de Fármacos. Revista Virtual de Química, v. 4, n. 6, p. 739-776, 2012. Disponível em: https://rvq-sub.sbq.org.br/index.php/rvq/article/view/295/290. Acesso em: 21 set. 2021.
STERLING, Teague; IRWIN, John J. ZINC 15- Ligand Discovery for everyone. Journal of Chemical Information and Modeling, v. 55, n. 11, p. 2324-2337, 2015. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4658288/pdf/ci5b00559.pdf. Acesso em: 22 jan. 2022.
THOMSEN, R. CHRISTENSEN, M. H. MolDock: A New Technique for HighAccuracy Molecular Docking. Journal of Medicinal Chemistry, v. 49, n. 11, p. 3315- 3321, 2006. Disponível em: https://pubmed.ncbi.nlm.nih.gov/16722650/. Acesso em: 19 jan. 2022.
TORKTAZ, I.; ZAHIRI, H. S.; NOHABI, K. A. In silico modeling of the type 2 IDI enzymes of Bacillus licheniformis, Pseudomonas stutzeri, Streptococcus pyogenes, and Staphylococcus aureus for virtual screening of potential inhibitors of this therapeutic target. J. Mol. Graph. Mod. p. 176-182, 2013. Disponivel em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8587310/ Acesso em: 9 fev. 2022.
TSENOVA, Liana et al. Use of imid3, a thalidomide analog, as na adjunct to therapy for experimental tuberculous meningitis. Antimicrobial agents and chemotherapy, v. 46, n. 6, p. 1887-1895, 2002. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC127267/pdf/1088.pdf. Acessado em: 27 mar. 2022.
VIJAYAKUMAR, K. R.; GOWDA, L. R. Rice (Oryza sativa) lípase: Molecular cloning, functional expression and substrate specificity. Protein Expression and Purification, p. 67-79, 2013. Disponível em: https://pubmed.ncbi.nlm.nih.gov/23202292/ Acesso em: 7 out. 2021.
WAN, Y. et al. [1,2,4] Triazolo [1,5-a] pyrimidine derivative (Mol-5) is a new NS5-RdRp inhibitor of DENV2 proliferation and DENV2- induced inflammation. Acta Pharmacol. Sin., v.41, p.706–718, 2020. Disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7471397/. Acesso em: 27 set. 2021.
WU, J. et al. A conformation-based intra-molecular initiation factor identified in the flavivirus RNA-dependent RNA polymerase. PLoS Pathog. v.16, p. 1-25, 2020. Disponível em: https://journals.plos.org/plospathogens/article?id=10.1371/journal.ppat.1008484. Acesso em: 14 fev. 2022.
















