Autores
dos Santos, I. (UNIVERSIDADE FEDERAL DO AMAPÁ) ; de Lima, L. (UNIVERSIDADE FEDERAL DO AMAPÁ) ; Sanches, V.H.S. (UNIVERSIDADE FEDERAL DO AMAPÁ) ; Ryan, R. (UNIVERSIDADE FEDERAL DO AMAPÁ) ; dos Santos, C. (UNIVERSIDADE FEDERAL DO AMAPÁ)
Resumo
O câncer de pele é uma doença comum e diversos alvos são estudados para a busca
de novas terapias. O aumento nos níveis do fator de iniciação eucariótica 4A1
(eIF4A1) estão relacionados ao mau prognóstico clínico em diversos tipos de
câncer.
Além disso, a Rocaglamida-A possui atividade citótoxica contra linhagens de
células de melanoma. O objetivo do trabalho foi avaliar, por meio de docking
molecular, a afinidade de ligação (∆G) de compostos promissores- provenientes de
triagem virtual hierárquica com base em Roc-A- no sítio ativo do receptor eIF4A1.
Palavras chaves
Rocaglamida-A; Docking Molecular; eIF4A1
Introdução
O câncer de Pele (CP) é uma doença comum, possui alto custo de tratamento e
impacta demasiadamente os sistemas públicos/ privados de saúde (GUY JR et al.,
2015). DE SOUZA et al. (2011) reportaram que 114 mil novos casos estimados no
Brasil, representariam elevado impacto financeiro ao sistema público e privado
de saúde de cerca de 63 milhões de reais. Sendo assim, a busca por terapias
anti-CP usando abordagens de bioinformática é uma estratégia promissora.Além
disso, as rocaglamidas são metabólitos secundários de plantas do gênero Aglaia
(família Meliaceae) (PAN et al., 2013). Estes compostos possuem
atividade anticâncer exercida, por meio de diversos mecanismos moleculares
(BASMADJIAN et al., 2013). Então, a Rocaglamida-A (Roc-A) é um composto de
referência devido à sua atividade citotóxica, observada em linhagens de células
de CP descrita por WU et al. (1997). Além disso, o aumento nos níveis do fator
de iniciação eucariótica 4A1 (eIF4A1) está relacionado ao mau prognóstico
clínico em diversos tipos de câncer, incluindo o CP (JOYCE et al., 2017). Então
objetivo do presente trabalho foi avaliar, por meio de docking molecular, a
afinidade de ligação (∆G) e interações dos compostos promissores- provenientes
de triagem virtual hierárquica com base em Roc-A, conforme descrito por SANTOS
et al.(2021) - no sítio ativo do receptor eIF4A1.
Material e métodos
Conforme descrito por SANTOS et al.(2021), as estruturas da Roc-A e 10 compostos
promissores, foram preparadas por meio do software Discovery Studio Visualizer
(BIOVIA et al., 2000). O receptor elF4A1-ATP (PDB ID 5ZC9, Homo sapiens,
inibidor: RCG, depositado por Iwasaki et al., 2019) foi submetido ao software
AutoDock 4.2/Vina 1.1.2 com interface gráfica no software PyRx versão 0.8.30
(https://pyrx.sourceforge.io). A metodologia foi validada pelo cálculo do root-
mean-square deviation (RMSD), realizado comparando a conformação do ligante
cristalográfico e a melhor conformação obtida via docking molecular. As
coordenadas do grid center para X, Y e Z foram as seguintes: 42.459, 5.194 e
44.166, respectivamente. Além disso, o tamanho do grid box foi ajustado em 32X,
38Y e 32Z. A medida do RMSD e análise de interações foram feitas usando o
Discovery Studio Visualizer na versão supracitada. O gráfico foi construído
usando o software GraphPadPrism 8.0 (GraphPad Software Inc., San Diego, CA).
Resultado e discussão
A partir da pose cristalográfica e da predita computacionalmente de RCG,
obtivemos um RMSD = 1.43Å, calculado via Discovery Studio Visualizer. Sendo
assim, de acordo com Hevener et al. (2009) o docking molecular é validado quando
o RMSD for menor que 2Å, i.e, a nossa análise foi validada. No sítio ativo do
receptor elF4A1-ATP obtivemos os seguintes resultados para afinidade de ligação
(∆G) em -kcal/mol: RCG = -5.8, Roc-A= -5.4, PC-7581023 = -5.0, PC-16811025 =
-5.4, PC-16810169 = -4.7, PC-16810171 = -4.8, PC-530932200 = -5.7, PC-53116405 =
-5.0, PC-9115580 = -5.4, PC-1852767 = -5.9, PC-135638768 = -4.9 e PC-16803784 =
-5.5. Além disso, a análise das interações do composto PC-1852767 demonstrou que
ele realiza ligações de hidrogênios convencionais com os resíduos de aminoácidos
(ASP197 e ASP198), enquanto RCG realiza interações do mesmo tipo com GLN195 e
G8.
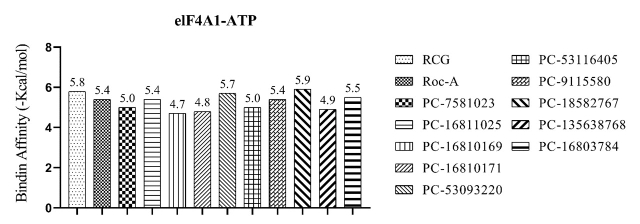
Resultados de afinidade de ligação do ligante complexado (RCG), molécula pivô (Roc-A) e compostos promissores com o receptor elF4A1-ATP (PDB ID 5ZC9).
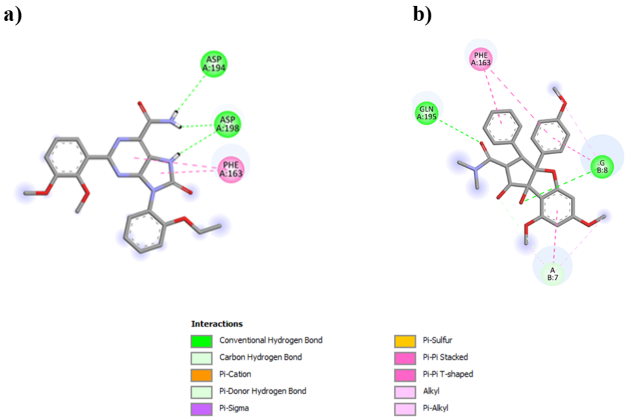
a) Interação do composto promissor PC-1852767 com resíduos de aminoácidos no sítio ativo do receptor elF4A1-ATP (PDB ID 5ZC9). b) Interação do inibido
Conclusões
Os resultados obtidos no presente estudo demonstraram que o composto promissor
PC-1852767 possui afinidade de ligação (∆G), no sítio de ligação do receptor
elF4A1-ATP, maior que o do inibidor complexado RCG. Além disso, a análise das
interações mostrou que o composto promissor realiza duas ligações de hidrogênios
convencionais, assim como o inibidor complexado, entretanto os resíduos de
aminoácidos são diferentes. Os resultados obtidos tornam o composto promissor PC-
1852767 candidato a teste biológicos para investigação robusta de sua atividade
anti-CP.
Agradecimentos
Universidade Federal do Amapá (EDITAL Nº 01/2021 - PROPESPG/UNIFAP), Laboratório
de Química e Modelagem Molecular-UNIFAP, Bionorte - Rede de Biodiversidade e
Biotecnologia da Amazônia Legal.
Referências
BIOVIA, D. S. ET AL. Dassault systèmes BIOVIA, discovery studio visualizer, v. 17.2, San Diego: Dassault Systèmes, 2016. J. Chem. Phys, [S. l.], v. 10, p. 21–9991, 2000.
GUY JR, GERY P.; MACHLIN, STEVEN R.; EKWUEME, DONATUS U.; YABROFF, K. ROBIN. Prevalence and costs of skin cancer treatment in the US, 2002− 2006 and 2007− 2011. American journal of preventive medicine, [S. l.], v. 48, n. 2, p. 183–187, 2015.
HEVENER, KIRK E.; ZHAO, WEI; BALL, DAVID M.; BABAOGLU, KERIM; QI, JIANJUN; WHITE, STEPHEN W.; LEE, RICHARD E. Validation of molecular docking programs for virtual screening against dihydropteroate synthase. Journal of chemical information and modeling, [S. l.], v. 49, n. 2, p. 444–460, 2009.
IWASAKI, SHINTARO ET AL. The Translation Inhibitor Rocaglamide Targets a Bimolecular Cavity between eIF4A and Polypurine RNA. Molecular Cell, [S. l.], v. 73, n. 4, p. 738- 748.e9, 2019. DOI: 10.1016/j.molcel.2018.11.026. Disponível em: https://doi.org/10.1016/j.molcel.2018.11.026.
WU, TIAN-SHUNG; LIOU, MEEI-JEN; KUOH, CHANG-SHENG; TENG, CHE-MING; NAGAO, TSUNEATSU; LEE, KUO-HSIUNG. Cytotoxic and antiplatelet aggregation principles from Aglaia elliptifolia. Journal of Natural Products, [S. l.], v. 60, n. 6, p. 606–608, 1997.
















