Autores
Ramos Menezes Santos Soeiro, Y. (UNIVERSIDADE FEDERAL DO MARANHÃO) ; de Jesus Gomes Varela Junior, J. (UNIVERSIDADE FEDERAL DO MARANHÃO) ; Monteiro dos Santos, A. (UNIVERSIDADE FEDERAL DO OESTE DO PARÁ)
Resumo
Ao todo o projeto foi divido em 2 etapas, sendo que no momento serão apresentados
os resultados referentes aos primeiros 100 nanosegundos de dinâmica molecular MM.
Objetiva-se analisar a histona 3 lisina 9 (H3K9)e suas mutações, os valores de
energia livre referente a cada um desses mutantes durante a reação, verificar o
comportamento dessas histonas referentes ao processo de metiltransferase (MT) e no
geral o comportamento reacional dos sistemas. Dos 5 sistemas estudados entre o
sistema nativo (H3K9) e seus mutantes (H3K9M, H3K9A, H3K9I e H3K9Nle) utilizou-se
de analises de RMSD e energia livre para apurar qual apresenta melhor
estabilidade. Os resultados apontam para o H3K9M e o H3K9I como os mais estáveis
inibidores da G9a e potenciais ocasionadores de problemas como o câncer.
Palavras chaves
Histona ; Metiltransferase; Dinâmica Molecular
Introdução
A metilação é um dos processos epigenéticos mais investigados (COSTA;
PACHECO,2013), entretanto apesar de ser um mecanismo natural esse processo chama
atenção por levar a problemas de carcinomas caso sofra modificações
desproporcionais ao longo da reação (SILVA et al,2020).Em razão dessa relação
de desproporcionalidade é importante mencionar a hipometilação e a
hipermetilação que podem acabar por exercer uma inconstância da regulação do
código genético influenciando o crescimento celular desordenado e
consequentemente o surgimento de algum tipo de câncer (SILVA et al,2020).
Contudo esses mecanismos estão ligados a uma proteína responsável pela
organização do DNA: As histonas. A H3K9, histona 3 com a lisina na posição 9,
tem suas responsabilidades desempenhadas e determinadas pelo tipo de mecanismo
relacionado a histona, se for o caso da ativação dos genes há a acetilação e em
relação ao silenciamento dos genes está mais associada a metilação (KARMODIYA et
al,2012). De certo modo os mecanismos relacionados a H3K9 estão correlacionados
a enzima G9a do qual é o agente contribuinte pela catalise da metiltransferase
nos resíduos de lisina 9 e lisina 27 juntamente ao cofactor SAM (TACHIBANA et
al,2008). Tendo em vista que
a metilação é um processo natural do nosso organismo e ela apresenta suas
singularidades, se faz necessário compreender o processo de interação entre a
histona humana H3 e suas variantes em relação as enzimas metiltransferases.
Material e métodos
As simulações de Dinâmica Molecular (MD) foram realizadas no software NAMD29.
Foram 5 sistemas analisados, provenientes de estrutura cristalográfica (PDB
code:51hn) (BERMAN et al.,2000) de complexos da enzima G9a-H3 sendo eles a H3
nativa H3K9 e mutações
da H3K9 (H3K9A, H3K9M, H3K9I, H3K9Nle) todos em complexos com o coligante SAM.
Os campos de força de todos os átomos CHARMM32m e CHARMM General Force Field
(CGenFF) serviram como fonte de parametrização. As cargas de proteína foram
calculadas em Gaussian 09 usando o método Hartree-Fock com base 6-31G*. A
solvatação se deu com um modelo explícito TIP3P em uma caixa de água octaédrica
truncada. Um valor de corte de 10,0 Å foi usado como distância entre a parede
celular e os átomos solvatados do sistema. Moléculas de água, hidrogênios, íons
e toda a
estrutura da proteína foram minimizados por cinco etapas de minimização usando a
descida mais acentuada e gradiente conjugado com relaxamento progressivo das
restrições. Em seguida, todo o sistema foi aquecido através de 14 etapas de
aquecimento. A 1ª etapa de aquecimento foi realizada a volume constante durante
5 ps (0 a 5K), depois da 2ª etapa até a 7ª etapa, utilizamos 50 ps para elevar a
temperatura gradualmente de 5 a 300K. Por fim, a partir da 7ª etapa de
aquecimento, a temperatura atinge 300 K, sendo mantida na 14ª. Realizamos 2 ns
de simulação MD para equilibrar a densidade do sistema a pressão constante (1
bar) mantendo a temperatura em 300K. Todas as ligações de hidrogênio foram
mantidas em suas distâncias de equilíbrio pré-definidas durante a minimização
com o algoritmo Shake para estabelecer o equilíbrio do sistema.
Resultado e discussão
A Figura 1A e Figura 1B são as estruturas dos sistemas mais estáveis, H3K9M e
H3K9I, interagindo
com o cofactor SAM respectivamente. Foi utilizado o RMSD por permitir com que
seja possível observar o comportamento de todo sistema durante a trajetória de
dinâmica molecular(CARUGO, Oliviero,2003). Na Figura 1C e 1D é possível ver o
RMSD dos dois mutantes que mais chamaram atenção, no qual comparando ambos os
gráficos podemos ver a diferença entre os dois mutantes. O H3K9M (Figura1C)
demonstrou pouca variação no comportamento do RMSD durante toda a dinâmica
molecular apresentando grande estabilidade durante maior parte do tempo de
simulação. Já o H3K9I(Figura 1D) apresentou pouca variação somente no início e
no fim da dinâmica molecular por volta de 0 a 20 ns e 80 e 100 ns
respectivamente onde tal fato se remete a busca do sistema em encontrar o melhor
ajuste conformacional durante o tempo de DM. Com a intenção de comparar os dados
de estabilidade do RMSD, foram realizados cálculos de energia(ΔGcp). Por meio
dos valores de energia é possível notar que os sistemas mais estáveis comentados
inicialmente continuam sendo os mais estáveis baseados nos valores de energia
obtidos. Foi observado que os resíduos H3K9A e H3K9Nle apresentaram os maiores
índices energéticos alcançados com valores de -77,66kcal.mol-1 e -49,25
kcal.mol-1 respectivamente. É notável que os dois outros sistemas continuaram a
se destacar em relação aos valores energéticos onde o H3K9I segue sendo um dos
mais estáveis com valor apresentado de -116,61 kcal.mol-1. O H3K9M segue a mesma
linha analisada até aqui onde apresenta um dos menores índices de energia,
-80,68 kcal.mol-1.
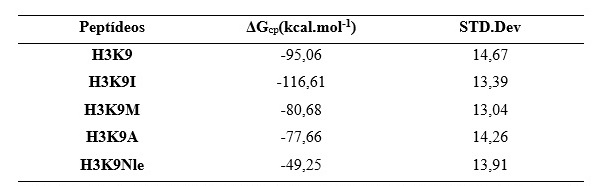
Valores de energia calculados computacionalmente (ΔGcp) e de STD. Deviation.
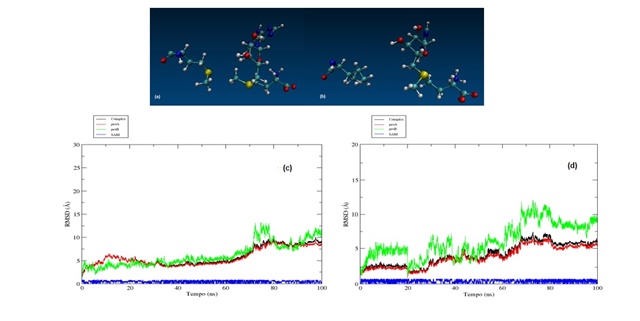
Figura 1A e Figura 1C são referentes ao H3K9M Figura 1B e Figura 1D são referentes ao H3K9I
Conclusões
Os mutantes H3K9I e H3K9M foram o que obtiveram maior destaque durante todo o
trabalho. Verificando todos os pontos analisados observamos que o H3K9M teve a
melhor performance no geral, sendo assim a lisina 9 com a mutação da metionina é
vista como o melhor inibidor da G9a e posteriormente ocasionar problemas como o
câncer. Não só por isso, mas o H3K9I também tem grande destaque no que tange a
inibição da atividade biológica da G9a sendo também um inibidor nessa reação. Em
suma, considerando os 100 nanosegundos iniciais desse trabalho, os resultados
obtidos são bastante animadores.
Agradecimentos
Os autores agradecem ao Programa de Pós Graduação em Química da Universidade
Federal do Maranhão (PPGQUIM-UFMA) pelo suporte e a FAPEMA pelo apoio financeiro
durante a realização do projeto.
Referências
BERMAN, H. M; WESTBROOK, J; FENG, Z; GILLILAND, G; BHAT, T. M; WEISSIG, H; SHINDYALOV, N. N; BOURNE, P. E. The Protein Data Bank. Nucleic acids research, v. 28, n. 1, p. 235–242, 2000.
CARUGO, Oliviero. How root-mean-square distance (rmsd) values depend on the resolution of protein structures that are compared. Journal of applied crystallography, v. 36, n. 1, p. 125-128, 2003.
COSTA, E.B., PACHECO, C. Epigenética: Regulação da Expressão Gênica em Nível Transcricional e Suas Implicações. Semina: Ciências Biológicas e da Saúde, Londrina, v. 34, n. 2, p. 125-136, jul./dez., 2013.
KARMODIYA, Krishanpal et al. H3K9 and H3K14 acetylation co-occur at many gene regulatory elements, while H3K14ac marks a subset of inactive inducible promoters in mouse embryonic stem cells. BMC genomics, v. 13, n. 1, p. 1-18, 2012.
SILVA, G. A; CASTRO, N. S; FIGUEIREDO, R. O. Mecanismos epigenéticos e a ação da expressão da proteína BRCA na carcinogênese mamária. Brazilian Journal of Development, v. 6, n. 10, p. 82596-82613, 2020.
TACHIBANA, M; MATSUMARA, Y; FUKUDA, M; KIMURA, H; SHINKAI, Y. G9a/GLP complexes independently mediate H3K9 and DNA methylation to silence transcription. The EMBO Journal. v. 27, p. 2681–2690. 2008.
















