Autores
Ferreira, L.C.F. (UNIVERSIDADE FEDERAL DO ESPÍRITO SANTO - UFES) ; Silva, J.A.B. (UNIVERSIDADE FEDERAL DO ESPÍRITO SANTO - UFES) ; Morais, P.A.B. (UNIVERSIDADE FEDERAL DO ESPÍRITO SANTO - UFES)
Resumo
A Lanosterol 14alfa-desmetilase é membro da família CYP51. A inibição dessa enzima
implica na perda de ergosterol, no caso dos fungos. Infecções causadas por fungos
apresentam grande incidência, aumentando a busca por novos fármacos. O Timol é um
produto natural que apresenta inúmeras atividades biológicas, inclusive,
antifúngica. Tendo em vista a necessidade por novos inibidores da enzima em
questão e as atividades biológicas apresentadas pelo Timol, o objetivo deste
trabalho é a predição, através de docking molecular, de possíveis modos de
interação entre os derivados do Timol e o alvo biológico. Após análises, observou-
se que os compostos 1a, 1b e 1c interagiram com a proteína através de ligação de
hidrogênio com Tyr140.
Palavras chaves
Docking Molecular; Lanosterol 14alfa-desmeti; Timol
Introdução
A Lanosterol 14alfa-desmetilase, membro da família CYP51, é uma enzima
importante na via biossintética humana e de fungos, participando da biossíntese
do colesterol e ergosterol. A inibição dessa enzima implica em uma depleção de
ergosterol das membranas celulares, prejudicando a fluidez da bicamada lipídica
e retardando seu crescimento (HAFIDI, et al. 2021). A taxa de incidência de
infecções fúngicas tem crescido nas últimas décadas. Entretanto, o cenário para
o tratamento destas infecções está se tornando cada vez mais complexo, tendo em
vista a resistência antifúngica. Frente a este problema, torna-se necessário a
busca por novas moléculas ativas (JUNG, et al. 2021; REVIE, et al. 2018).
Compostos químicos derivados de produtos naturais são uma excelente alternativa
na descoberta de novos agentes antifúngicos devido a seus fortes poderes de
inibição (MARTINEZ-ROSSI et al. 2018; HENDRICKSON et al. 2019). O Timol é um
monoterpeno e é o componente principal do óleo essencial do tomilho (Thymus
vulgaris). O produto natural em questão apresenta uma gama de atividades
biológicas, sendo estas anticarcinogênicas, anti-inflamatórias, atividade
antibacteriana e antifungica. O mecanismo de ação do timol contra patógenos
fúngicos deve—se à sua capacidade de adulteração na membrana, através da redução
da biossíntese de ergosterol (ELOY, et al 2021). Devido a gama de atividades
biológicas apresentadas pelo timol e a necessidade
por novos agentes antifúngicos, o objetivo deste trabalho é a predição, através
do estudo de docking molecular, de possíveis modos de interação entre os
derivados do timol com a enzima Lanosterol 14alfa-desmetilase.
Material e métodos
Os estudos de docking foram realizados utilizando a estrutura cristalográfica da
Lanosterol 14alfa-desmetilase G73E mutante (PDB: 5ESG; resolução 1.98 Å),
previamente retirada do Protein Data Bank
(http://www.rcsb.org/pdb/home/home.do), e as estruturas dos ligantes previamente
otimizadas. Os cálculos de docagem foram efetuados no programa software Gold
Suite 5.1 (Genetic Optimization for Ligand Docking 5.1) (CCDC Software Limited)
em computador de alta performance (Workstation). Os seguintes passos foram
seguidos: 1. Isolamento e análise da estrutura cristalográfica da Lanosterol
14alfa-desmetilase G73E mutante do Protein Data Bank (PDB); 2. Preparação dos
ligantes, por meio de otimização da geometria molecular
realizada no programa ChemBio3D Ultra 12.0; 3. Validação do método de docking,
por meio do redocking do ligante co-cristalizado e avaliação do RMSD (Root-Mean-
Square Deviation) da pose obtida e do ligante no cristal a fim de medir as
diferenças entre as soluções; 4. Realização do docking molecular dos ligantes
planejados no programa GOLD V5.1 (CCDC Inc.). Os estudos de docking foram
realizados a fim de obter 100 poses (soluções com diferentes conformações) para
cada ligante de modo a ter uma amostragem estatística das poses mais bem
ranqueadas; 5. Análise dos resultados. A análise dos resultados foi, em um
primeiro momento, realizada pela avaliação e seleção da melhor pose (maior valor
de score). Após a seleção foi realizada a análise visual da pose selecionada
para cada ligante, verificação da complementariedade química com o sítio ativo
da enzima e medidas das principais distâncias de interação. Os ligantes foram
sintetizados e publicados por nosso grupo (ELOY, et al. 2021).
Resultado e discussão
Os modos de ligação dos compostos 1a (1-(2-bromofenil)-4-((2-isopropil-5-
metilfenoxi)metil)-1H-1,2,3-triazol), 1b (4-((2-isopropil-5-
metilfenoxi)metil)-1-(2-metoxifenil)-1H-1,2,3-triazol) e 1c (4-((2-isopropil-5-
metilfenoxi)metil)-1-(3-nitrobenzil)-1H-1,2,3-triazol) mostraram que os
derivados do timol complementam o sítio de ligação de ATP. Foi
utilizada a função ChemScore dentro de uma esfera de raio de 10 Å, centrada no
ligante da cadeia A, desconsiderando moléculas de água e usando os parâmetros
padrão disponíveis, que incluíam uma população de 100 confórmeros, 100.000
operações, 95 mutações e 95 cruzamentos. A estrutura de energia mais baixa
apresentou um RMSD de 1,4001 Å (Figura 1), com base no ligante na cadeia A do
complexo itraconazol/Lanosterol 14alfa-desmetilase. O ligante cristalográfico
realiza ligação de hidrogênio com resíduo de Glu73 (2,9 Å), além de ligação com
o complexo de ferro (Figura 1). Os compostos 1a, 1b e 1c interagiram com a
proteína através de ligação de hidrogênio com Tyr140 (Figura 2). As ligações de
hidrogênio com resíduo de Tyr140 ( 2,0 Å e 2,8 Å para 1a; 1,9 Å e 2,2 Å para 1b
e 1,8 Å para 1c) são importantes para a interação com a enzima, assim como
mostrado nos complexos itraconazol/Lanosterol 14alfa-desmetilase (PDB 4ZDY) e
fluconazol/Lanosterol 14alfa-desmetilase (PDB 4ZE3) (SAGATOVA, et al. 2018). As
interações feitas pelos compostos podem explicar valores de concentração
inibitória mínima (CIM) e concentração fungicida mínima (CFM) encontradas em
testes de determinação de CIM e CFM para Trichophyton rubrum CCT 5506. Os
valores de CIM e CFM em µg/mL foram, respectivamente, 32 µg/mL e 128 µg/mL para
o composto 1a; 8 µg/mL e 16 µg/mL para o composto 1b; 4 µg/mL e 8 µg/mL para o
composto 1c e 0,03 µg/mL e 0,03 µg/mL para o itraconazol.
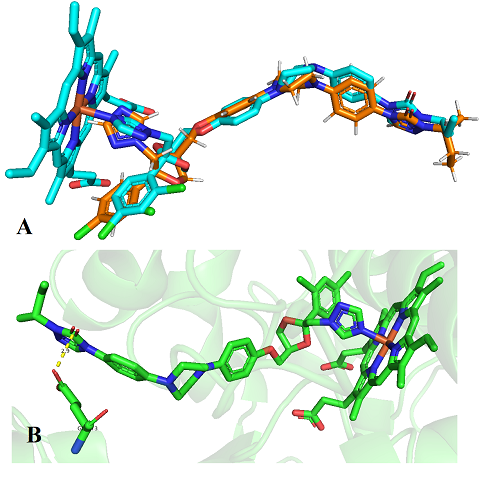
(A) Resultado do redocking. Em azul o ligante Itraconazol e em laranja o ligante do redocking. (B) Interação do ligante da proteína com Glu73.
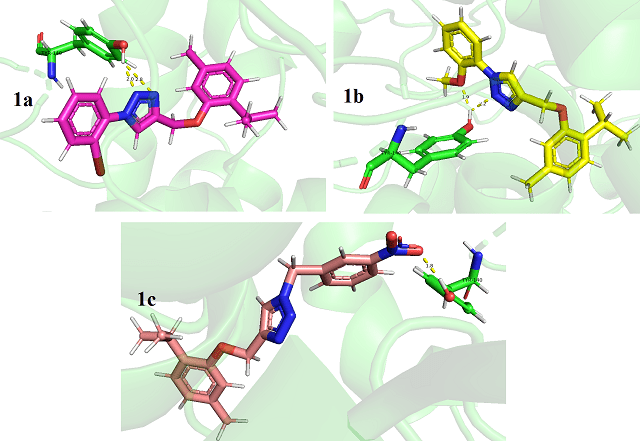
Interações do tipo ligação de hidrogênio dos compostos 1a, 1b e 1c com resíduos de Tyr140.
Conclusões
O ligante do PDB 5ESG realizou ligação de hidrogênio com resíduo de Glu73, além de
uma ligação com o complexo de ferro. Os compostos 1a, 1b e 1c interagiram com a
proteína através de ligação de hidrogênio com Tyr140, que são importantes para a
interação com a enzima, como mostrado nos PDB: 4ZDY e PDB: 4ZE3. Interações
observadas demonstram que os derivados do timol apresentam complementaridade no
sítio de ligação de ATP, justificando os ótimos valores encontrados para os testes
de CIM e CFM. Os resultados comprovam que os compostos 1a-c são promissores.
Agradecimentos
Agradecemos a Universidade Federal do Espírito Santo (UFES), ao Programa de Pós-
Graduação em Agroquímica (PPGAQ) e a Coordenação de Aperfeiçoamento de Pessoal
Nível Superior (CAPES).
Referências
ELOY, M. A. et al.; Thymol as na Interesting Building Block for Promising Fungicides against Fusarium solani. Agric. Food Chem. 2021, 69, 6958−6967.
HAFIDI, Z., EL ACHOURI, M., O. SOUSA, F. F., & PÉREZ, L. Antifungal activity of amino-alcohols based cationic surfactants and in silico, homology modeling, docking and molecular dynamics studies against lanosterol 14-α-demethylase enzyme. Journal of Biomolecular Structure and Dynamics. p. 1–17. 2021.
HENDRICKSON, J. A.; HU, C.; AITKEN, S. L.; BEYDA, N. Antifungal Resistance: a Concerning Trend for the Present and Future. Curr. Infect. Dis. Rep. 2019, 21.
JUNG, K. W.; CHUNG, M. S.; BAI, H. W.; CHUNG, B. Y.; LEE, S. Investigation of Antifungal Mechanisms of Thymol in the Human Fungal Pathogen, Cryptococcus neoformans. Molecules, 2021, 26, 1.
MARTINEZ-ROSSI, N. M.; BITENCOURT, T. A.; PERES, N. T. A.; LANG, E. A. S.; GOMES, E. V.; QUARESEMIN, N. R.; MARTINS, M. P.; LOPES, L.; ROSSI, A. Front. Microbiol. 2018, 9, 1.
REVIE, N. M.; IYER, K. R.; ROBBINS, N.; COWEN, L. E. Antifungal drug resistance: evolution, mechanisms and impact. Curr. Opin. Microbiol. 2018.
SAGATOVA AA, KENIYA MV, TYNDALL JDA, MONK BC. Impact of Homologous Resistance Mutations from Pathogenic Yeast on Saccharomyces cerevisiae Lanosterol 14α-Demethylase. Antimicrob Agents Chemother. 2018 Feb 23;62(3):e02242-17. doi: 10.1128/AAC.02242-17. PMID: 29263059; PMCID: PMC5826108.
















