Autores
Corredor, J. (UNAB) ; Jaña, G. (UNAB) ; Jimenez, V. (UNAB)
Resumo
Se evaluó el efecto de los estados de protonación de los residuos His126, Asp663 y
Asp709, cercanos al sitio activo de la enzima DPP4, en el reconocimiento del
inhibidor VIL. El estudio se realizó mediante cálculos de dinámica molecular MD,
cálculos MM/GBSA y análisis Hbond para determinar la formación de enlaces de
hidrogeno. Se estableció que el sistema con His126 protonada en épsilon y los
residuos Asp protonados favorece el reconocimiento de VIL. En esa configuración
VIL se posiciona tal como en la estructura cristalina y se generan enlaces de
hidrogeno con los residuos considerados de reconocimiento, coincidiendo con lo
planteado en el mecanismo de reacción.
Palavras chaves
DPP4; Vildagliptina; Dinamica molecular
Introdução
La diabetes mellitus (DM) es una enfermedad metabólica crónica considerada un
problema de salud pública debido a su alta prevalencia. Esta enfermedad se
manifiesta por una elevada concentración de glucosa en la sangre durante un
tiempo prolongado, debido a la deficiencia relativa o absoluta de la hormona
insulina, ocasionando complicaciones multisistémicas en el organismo (BILOUS et
al 2021). Se estima que 463 millones de personas entre 20-79 años padecen DM en
el mundo, proyectándose una cifra de 700 millones para el año 2045 (BILOUS et al
2021; ZHENG et al., 2018). Dependiendo de los factores que generan la
enfermedad, existen diferentes tipos de diabetes. Las formas más comunes y con
mayor impacto en la salud publica son la diabetes tipo 1 (DM1) y diabetes tipo 2
(DM2), siendo ésta última la de mayor prevalencia con aproximadamente el 90% de
casos (BILOUS et al 2021).
En el caso de DM2 se han desarrollado múltiples tratamientos enfocados en
diferentes dianas farmacológicas que participan en la liberación de la insulina
y en el metabolismo de carbohidratos (DIABETES CARE, 2021). Entre estos se
encuentran los inhibidores de la enzima dipeptidil peptidasa-4 (DPP4, EC
3.4.14.5), principal reguladora de la disponibilidad de péptido 1 tipo glucagón
(en inglés Glucagon-Like Peptide-1, GLP-1) y péptido insulinotrópico dependiente
de la glucosa (en inglés Glucose-dependent Insulinotropic Peptide, GIP) en el
organismo (DRUCKER et al, 2006; KIM et al, 2008). Esta enzima exopeptidasa
reconoce los aminoácidos alanina en la penúltima posición del sustrato e
hidroliza el enlace peptídico correspondiente suprimiendo su actividad
insulinotropica (KIM et al, 2008; SEINO et al., 2010). Por esta razón, la
inhibición de DPP4 implica un aumento en la disponibilidad de GLP-1 y GIP,
promoviendo la secreción de insulina en el páncreas y reduciendo la
hiperglicemia en personas afectadas por DM2. En la actualidad existe un número
importante de inhibidores de DPP4 que se administra en tratamientos para esta
enfermedad con un mínimo de efectos secundarios, lo que le confiere primacía
frente a los tratamientos dirigidos a otras dianas farmacológicas (DIABETES
CARE, 2021; KUMAR et al., 2021). Además, estudios recientes relacionan la DPP4
con la infección por SARS-CoV-2 (COVID-19) debido a su papel como segundo
receptor del virus y su rol en procesos inflamatorios en el pulmón,
incrementando el interés en el desarrollo de fármacos inhibitorios (DRUCKER,
2021; SOLERTE et al., 2020).
La enzima DPP4 es una proteína transmembrana que principalmente se presenta
formando estructuras diméricas ancladas en la superficie celular o disuelta en
el plasma. La estructura está compuesta los dominios α/β hidrolasa y β-
helicoidal con la cavidad catalítica localizada en medio de los dominios
(AERTGEERTS et al, 2004; KLEMANN et al., 2016; LAMBEIR et al., 2003; RÖHRBORN et
al., 2015). La enzima presenta una triada catalítica del tipo nucleófilo-acido-
base, compuesta por los residuos Ser630, Asp708 e His740 (LAMBEIR et al., 2003).
En la reacción enzimática, el sustrato se enlaza covalentemente a través del
ataque nucleofílico de la Ser630 sobre el sustrato, formando un intermediario
tetraédrico que conduce a la ruptura del enlace peptídico (MATTEUCCI &
GIAMPIETRO, 2009). Los inhibidores covalentes siguen el mismo mecanismo debido a
que su estructura es similar a la del sustrato. Algunos inhibidores presentan un
anillo de pirrolidina (análoga a la prolina) con un grupo nitrilo en posición 2,
el cual reacciona con la Ser630 formando el complejo covalente imidato de enzima
(KUMAR et al., 2021).
Vildagliptina es un inhibidor covalente de la enzima DPP4, potente y selectivo,
sintetizado en 2003 (VILLHAUER et al., 2003). Dadas sus propiedades
farmacocinéticas, biodisponibilidad y bajo potencial de interacción con otros
medicamentos y alimentos, este fármaco es considerado eficaz, seguro y bien
tolerado (HE, 2012). Actualmente se utiliza para el tratamiento de diabetes tipo
2, solo y en combinación con otros fármacos antidiabetes. En una investigación
reciente se ha propuesto un mecanismo de inhibición de VIL y su análogo
Saxagliptina, incrementando el interés en la comprensión de los detalles
fisicoquímicos implicados en el reconocimiento de los fármacos y la inhibición
de esta enzima (WANG et al., 2019). También se ha demostrado que la velocidad de
unión de los inhibidores catiónicos como VIL es influenciada por fuerzas
culombicas atractivas, las cuales además definen el perfil de reacción y las
barreras energéticas asociadas (PAN et al., 2013; SCHNAPP et al., 2016). Por
esta razón, en esta investigación hemos determinado el estado de protonación más
probable para los residuos ácidos y básicos que se encuentran en la cavidad
catalítica de la enzima DPP4 a través de Dinámica Molecular.
Material e métodos
Construcción del sistema
La estructura cristalográfica del complejo DPP4/VIL (6B1E, resol. 1,77 Å) está
disponible en el Protein Data Bank (PDB) (BERGER et al., 2018). Los estados de
protonación de los residuos ácidos y básicos se determinaron a pH fisiológico de
7,4 utilizando el servidor H++ (ANANDAKRISHNAN et al., 2012). La tríada
catalítica de estado de reactivo se fijó como SER-HID-ASP y los diferentes
estados de la proteína se obtuvieron cambiando la protonación de los residuos
His126, Asp663 y Asp709. El reactivo VIL se reconstruyó a partir de la
estructura cristalina 6B1E y se parametrizó usando el programa Antechamber con
cargas obtenidas al nivel AM1-BCC. Mediante el programa LEAP se obtuvieron los
complejos proteína-inhibidor, neutralizados con iones Na+ o Cl-, y solvatados en
una caja octaédrica periódica con moléculas de agua OPC. Los complejos se
mantuvieron sumergidos a 10 Å desde el borde de la caja.
Dinámica molecular clásica
Los campos de fuerza ff19SB5225 y GAFF se implementaron para modelar las
estructuras de la proteína y de VIL. Las simulaciones de MD se realizaron con el
programa pmemd.CUDA incluido en AMBER20 (CASE, et al 2021). Se implementó el
método PME (Particle Mesh Ewald) para evaluar interacciones no covalentes hasta
un máximo de 10 Å. Todos los enlaces que incluyen hidrógeno se restringieron
utilizando el algoritmo SHAKE. El procedimiento para realizar las simulaciones
MD convencionales consistió en a) 1500 pasos de minimización descendente
seguidas de 3500 ciclos de minimización por gradiente conjugado, para la caja de
agua con el complejo restringido; b) 1.500 ciclos de minimización descendente,
seguidos de 6.500 ciclos de minimización del gradiente conjugado para todo el
sistema; c) 500 ps de calentamiento NVT de 0 a 300K; d) 500 ps de equilibrado
del sistema NVT; e) equilibrado NPT sin restricciones durante 20 ns a 300 K y f)
150 ns de dinámica de producción NPT sin restricciones a 300 K y 1 bar de
presión, con tiempos de integración de 2 fs. Este protocolo ha sido previamente
validado para el estudio de otros sistemas proteína-ligando (JIMÉNEZ et al.,
2021).
Los análisis de trayectorias se realizaron utilizando el software CPPTRAJ y VMD
(HUMPHREY, et al 1996). La herramienta Hbond se utilizó para determinar los
enlaces de hidrógeno formados. Las energías de enlace se calcularon utilizando
el método MM/GBSA.
Resultado e discussão
La secuencia de la enzima DPP4 humana se alineó con la estructura Uniprot P27487
con lo que se verificó que la estructura 6B1E no posee discontinuidades,
solamente presenta una mutación en el primer residuo (extremo N-terminal,
residuo 39) y por tanto la estructura es representativa de la enzima DPP4.
Teniendo en cuenta que se trata de una proteína homodimérica con actividad
catalítica independiente, se seleccionó uno de los monómeros y se determinó el
estado de protonación más probable a pH 7.4. En la cavidad catalítica de la
enzima se encuentran los residuos catalíticos Ser630, His740, Asp708 y Tyr547.
De igual forma se localizan los residuos ácidos y básicos: His126, Asp663,
Asp709, Asp739, His712 e His704 como se muestra en la Figura 1a. Los valores de
pKa calculados a pH fisiológico para los residuos His126, Asp663, Asp709 son
particularmente relevantes porque podrían no ser descritos correctamente por las
aproximaciones de H++. Con el fin de determinar el estado de protonación más
probable para los residuos mencionados, se prepararon los complejos con las
distintas combinaciones posibles para determinar la configuración que favorece
el reconocimiento de VIL en el sitio activo (Figura 1b).
Las dinámicas moleculares clásicas permitieron seleccionar los sistemas con el
mejor comportamiento en las cuales el inhibidor se mantiene durante más tiempo
en interacción con el sitio activo. Solamente seis de las configuraciones
estudiadas conservaron el ligando dentro de la cavidad catalítica y presentan
los menores valores de RMSD promedio (Figura 2a). Los valores RMSD calculados
han sido referenciados respecto a las posiciones atómicas de la estructura
cristalina por lo cual su valor es una medida relativa de la permanencia del
inhibidor en el sitio activo. Como se puede observar en la Figura 2b, de estos
seis sistemas, los sistemas 2, 3 y 8 presentan menores valores de RMSD con la
menor variabilidad.
En la reacción de inhibición covalente, el ataque nucleofílico de Ser630 sobre
el carbono del grupo nitrilo de VIL es el paso determinante de la reacción, por
lo tanto, la distancia que existe entre el residuo y el inhibidor define la
probabilidad de llevar a cabo la reacción. En la Figura 2c se muestran los
histogramas de frecuencia para la distancia Ser630-VIL medida entre los átomos
de Oxígeno y Carbono respectivamente. Para el sistema 8, la mayor frecuencia se
presenta para la distancia de 3-3.5 Å, con una diferencia contundente frente a
los otros sistemas. El análisis de los datos por prueba t-student a dos colas,
permite confirmar que el sistema seleccionado es estadísticamente diferente a
los demás, con un nivel de confianza del 95%.
Los cálculos de energía libre de unión a través de la aproximación MM/GBSA
(Figura 2d), muestran que el sistema 8 presenta el menor valor de energía y por
tanto es el sistema en el que el ligando genera interacciones más efectivas. La
tendencia de valores de energía respecto a la configuración del sistema permite
identificar que la protonación del residuo Asp663 contribuye significativamente
en el reconocimiento del inhibidor, dado que cinco de los seis sistemas implican
esa configuración. De igual forma, por la descomposición de los valores de
energía se pudo determinar que el incremento en la energía libre del sistema 8
es de naturaleza electrostática y no de dispersión de Van der Wals.
Por otra parte, la estructura cristalina del complejo DPP4/VIL, presenta
distancias entre el grupo amino de VIL y los residuos Glu205 y Glu206 del orden
de magnitud de los enlaces de hidrogeno. Esto también se observa entre el grupo
carbonilo y el residuo Asn710, sugiriendo que son interacciones relevantes en el
reconocimiento del inhibidor. El análisis Hbond de las trayectorias de los
sistemas 2, 3, 7 y 8 permitió establecer que únicamente en el sistema 8 se
presentan todas las interacciones mencionadas (Figura 2e). Esto indica que en
dicho complejo el inhibidor presenta el mejor posicionamiento en la cavidad
catalítica, muy similar a la estructura de los productos generados en la
reacción.
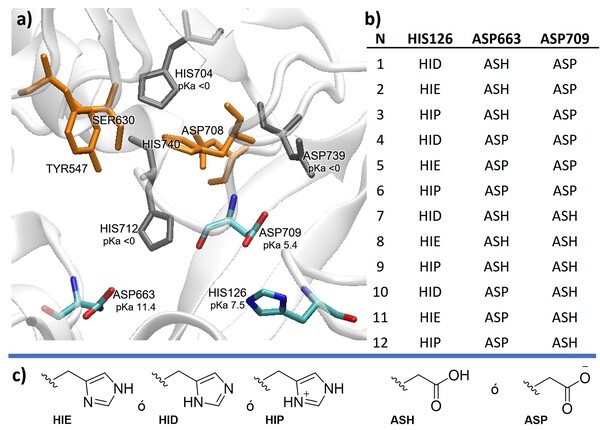
a) Residuos catalíticos (naranja) y residuos acidos y básicos (colores) b) Estados de protonación explorados. c) Estados de protonación de His y Asp.
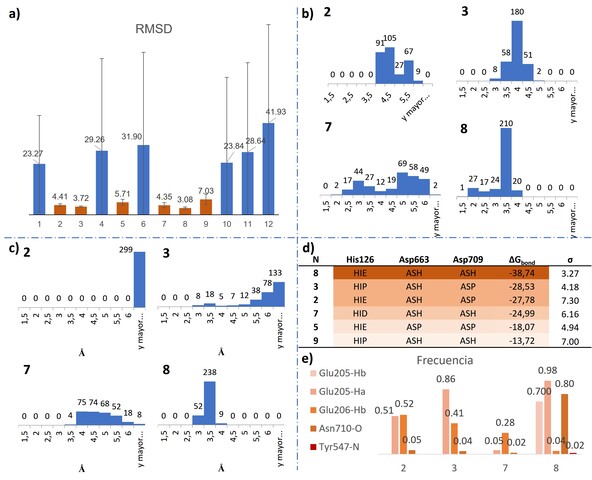
a) RMSD de los sistemas evaluados. b) Histogramas de frecuencia RMSD. c) Histogramas distancia Ser- VIL. d) Energía libre de unión MMGBSA. e) Frecuencia de formación de enlaces de hidrogeno.
Conclusões
Se estableció que el sistema 8, con His126 protonada en épsilon y los residuos
Asp663 y Asp709 protonados, favorece el reconocimiento de VIL. En esa
configuración VIL se posiciona tal como en la estructura cristalina y se generan
enlaces de hidrogeno con los residuos considerados de reconocimiento. La energía
libre de unión para ese sistema es considerablemente mayor, lo que sugiere una
mejor interacción del ligando con el sitio catalítico, coincidiendo con lo
planteado en el mecanismo de reacción.
Agradecimentos
A la Republica de Chile. Al programa de Doctorado en Fisicoquimica Molecular de la
Universidad Andres Bello de Chile. A la Agencia Nacional de investigación y
desarrollo ANID.
Referências
AERTGEERTS, K.; YE, SHENG; TENNANT, MIKE; KRAUS, MICHELLE; ROGERS, JOE; SANG, BI-CHING; SKENE, ROBERT; WEBB, DAVID; PRASAD, S. (2004). Crystal structure of human dipeptidyl peptidase IV in complex with a decapeptide reveals details on substrate specificity and tetrahedral intermediate formation. Protein Science, 13(2), 412–421.
ANANDAKRISHNAN, R., AGUILAR, B., & ONUFRIEV, A. V. (2012). H++ 3.0: automating pK prediction and the preparation of biomolecular structures for atomistic molecular modeling and simulations. Nucleic Acids Research, 40(W1), W537–W541.
BERGER, J. P., SINHAROY, R., POCAI, A., KELLY, T. M., SCAPIN, G., GAO, Y.-D., PRYOR, K. A. D., WU, J. K., EIERMANN, G. J., XU, S. S., ZHANG, X., TATOSIAN, D. A., WEBER, A. E., THORNBERRY, N. A., & CARR, R. D. (2018). A comparative study of the binding properties, dipeptidyl peptidase-4 (DPP-4) inhibitory activity and glucose-lowering efficacy of the DPP-4 inhibitors alogliptin, linagliptin, saxagliptin, sitagliptin and vildagliptin in mice. Endocrinology, Diabetes & Metabolism, 1(1), e00002. https://doi.org/10.1002/edm2.2
BILOUS, RUDY; DONNELLY, R., & IDRISS, I. (2021). Handbook of Diabetes (5th editio). John Wiley y Sons.
D.A. CASE, H.M. AKTULGA, K. BELFON, I.Y. BEN-SHALOM, S.R. BROZELL, D.S. CERUTTI, T.E. CHEATHAM, III, G.A. CISNEROS, V.W.D. CRUZEIRO, T.A. DARDEN, R.E. DUKE, G. GIAMBASU, M.K. GILSON, H. GOHLKE, A.W. GOETZ, R. HARRIS, S. IZADI, S.A. IZMAILOV, C. JIN, K. KA, AND P. A. K. (2021). Amber20 (Amber 2021). University of California.
DIABETES CARE. (2021). Pharmacologic approaches to glycemic treatment: Standards of medical care in diabetes-2021. Diabetes Care, 44(Suppl. 1), S111–S124.
DRUCKER, D. J. (2021). Coronavirus infections and type 2 diabetes-Shared pathways with therapeutic implications. Endocrine Reviews, 41(3), 457–469.
DRUCKER, D. J., & NAUCK, M. A. (2006). The incretin system: glucagon-like peptide-1 receptor agonists and dipeptidyl peptidase-4 inhibitors in type 2 diabetes. Lancet, 368, 1696–1705.
HE, Y. L. (2012). Clinical pharmacokinetics and pharmacodynamics of vildagliptin. Clinical Pharmacokinetics, 51(3), 147–162.
HUMPHREY, W., DALKE, A. AND SCHULTEN, K. (1996). VMD - Visual Molecular Dynamics. J. Molec. Graphics, 14, 33–38.
JIMÉNEZ, V. A., NAVARRETE, K. R., DUQUE-NOREÑA, M., MARRUGO, K. P., CONTRERAS, M. A., CAMPOS, C. H., & ALDERETE, J. B. (2021). Rational Design of Novel Glycomimetic Peptides for E-Selectin Targeting. Journal of Chemical Information and Modeling, 61(5), 2463–2474.
KIM, W., & EGAN, J. M. (2008). The role of incretins in glucose homeostasis and diabetes treatment. Pharmacological Reviews, 60(4), 470–512.
KLEMANN, C., WAGNER, L., STEPHAN, M., & VON HÖRSTEN, S. (2016). Cut to the chase: a review of CD26/dipeptidyl peptidase-4’s (DPP4) entanglement in the immune system. Clinical and Experimental Immunology, 185(1), 1–21.
KUMAR, S., MITTAL, A., & MITTAL, A. (2021). A review upon medicinal perspective and designing rationale of DPP-4 inhibitors. Bioorg. Med. Chem., 46(July).
LAMBEIR, A. M., DURINX, C., SCHARPÉ, S., & DE MEESTER, I. (2003). Dipeptidyl-peptidase IV from bench to bedside: An update on structural properties, functions, and clinical aspects of the enzyme DPP IV. Critical Reviews in Clinical Laboratory Sciences, 40(3), 209–294.
MATTEUCCI, E., & GIAMPIETRO, O. (2009). Dipeptidyl Peptidase-4 (CD26): Knowing the Function before Inhibiting the Enzyme. Current Medicinal Chemistry, 16(23), 2943–2951.
PAN, A. C., BORHANI, D. W., DROR, R. O., & SHAW, D. E. (2013). Molecular determinants of drug-receptor binding kinetics. Drug Discovery Today, 18(13–14), 667–673. https://doi.org/10.1016/j.drudis.2013.02.007
RÖHRBORN, D., WRONKOWITZ, N., & ECKEL, J. (2015). DPP4 in Diabetes. Frontiers in Immunology, 6(July), 386.
SCHNAPP, G., KLEIN, T., HOEVELS, Y., BAKKER, R. A., & NAR, H. (2016 sch). Comparative analysis of binding kinetics and thermodynamics of dipeptidyl peptidase-4 inhibitors and their relationship to structure. Journal of Medicinal Chemistry, 59(16), 7466–7477.
SEINO, Y., FUKUSHIMA, M., & YABE, D. (2010). GIP and GLP-1, the two incretin hormones: Similarities and differences. Journal of Diabetes Investigation, 1(1–2), 8–23.
SOLERTE, S. B., DI SABATINO, A., GALLI, M., & FIORINA, P. (2020). Dipeptidyl peptidase-4 (DPP4) inhibition in COVID-19. Acta Diabetologica, 57(7), 779–783.
VILLHAUER, E. B., BRINKMAN, J. A., NADERI, G. B., BURKEY, B. F., DUNNING, B. E., PRASAD, K., MANGOLD, B. L., RUSSELL, M. E., & HUGHES, T. E. (2003). 1-[[(3-Hydroxy-1-adamantyl)amino]acetyl]-2-cyano-(S)-pyrrolidine: A potent, selective, and orally bioavailable dipeptidyl peptidase IV inhibitor with antihyperglycemic properties. Journal of Medicinal Chemistry, 46(13), 2774–2789.
WANG, Y. H., ZHANG, F., DIAO, H., & WU, R. (2019). Covalent Inhibition Mechanism of Antidiabetic Drugs - Vildagliptin vs Saxagliptin. ACS Catalysis, 9(3), 2292–2302.
ZHENG, Y., LEY, S. H., & HU, F. B. (2018). Global aetiology and epidemiology of type 2 diabetes mellitus and its complications. In Nature Reviews Endocrinology (Vol. 14, Issue 2, pp. 88–98).
















