Autores
da Silva, A.F. (UNIVERSIDADE FEDERAL DE ALAGOAS) ; França, S.B. (UNIVERSIDADE FEDERAL DE ALAGOAS) ; Guimarães, A.S. (UNIVERSIDADE FEDERAL DE ALAGOAS) ; Correia, P.R.S. (UNIVERSIDADE FEDERAL DE ALAGOAS) ; Carvalho, C.M. (UNIVERSIDADE FEDERAL DE ALAGOAS) ; Lima, D.J.P. (UNIVERSIDADE FEDERAL DE ALAGOAS)
Resumo
Atualmente, o controle populacional do mosquito Ae. aegypti vem ocorrendo
mediante aplicação de inseticidas, entretanto, esses não vêm apresentando
eficácia devido à resistência generalizada entre as populações desse vetor.
Diante disso, o presente trabalho objetivou o planejamento racional de p-cloro -
cinamamidas. Para tanto, foi utilizado o método target fishing na busca
de múltiplos alvos do vetor. Os resultados in silico foram validados e
demostraram que essa classe de moléculas apresentam uma afinidade pronunciada
frente às proteínas acetilcolinesterase (Q6A2E2), hidrolase epóxida (Q8MMJ6) e
proteína de ligação ao hormônio juvenil (5V13), e pela análise de semelhança de
inseticida foi evidenciado que essa quimioteca pode ser um bom candidato a
agente larvicida.
Palavras chaves
Aedes aegypti; Cinamamidas; Estudos In Silico
Introdução
O Ae. aegypti é o vetor responsável pela transmissão de arboviroses, tais
como a Zika, Chikungunya, Dengue e Febre Amarela, afetando mais de cerca de meio
bilhão de pessoas mundialmente a cada ano (NATAL, 2002; BALWAN; SINGH; KOUR,
2022). Essas arboviroses têm causado grande impacto na saúde pública,
principalmente no que tange aos riscos de epidemias, considerando que não
existem vacinas eficazes para os arbovírus circulantes transmitido pelo Ae.
aegypti. Dessa forma, as tentativas de reduzir a disseminação dessas doenças
têm se concentrado no controle do manejo do vetor, com destaque ao meio químico,
que faz uso de inseticidas como o temefós (ZARA et al., 2016; JUAREZ
et al.,2021). Contudo, seu uso tem sido descontinuado devido a sua
toxicidade e carcinogenicidade, além das larvas apresentarem resistência
(MOREIRA, 2012; MÉLO, 2009). Diante desse cenário, faz-se necessário a busca por
moléculas mais ativas e seletivas frente a esse vetor (BARRETO, 2005). Nesse
aspecto, destacam-se os derivados do ácido cinâmico (DACs), os quais possuem uma
série de atividades farmacológicas, dentre elas: atividade antimicrobiana,
antitumoral, antioxidante, inseticida, larvicida entre outras. (PEPERIDOU et
al., 2017; SHARMA, 2011).
Nesse sentido, estudos desenvolvidos por França et al.(2021), reportam os
DAC’s como agentes larvicidas promissores, com destaque ao p-
clorocinamato de etila (Figura 1A) que apresentou uma CL 50 de 8,3
ppm. No mesmo estudo, França et al. (2021) demonstraram por estudos
in sílico e de relação-estrutura atividade, que o átomo de cloro na
posição para do anel aromático do grupo cinamoíl, exerce uma efetiva ação
larvicida. Por outro lado, as cinamamidas fazem parte de uma promissora classe
de compostos, descritas na literatura por possuírem uma vasta gama de atividades
biológicas, incluindo: antifúngica (DAI et al., 2015); herbicida (PENG
et al., 2021); antioxidantes, antibactericidas, anti-inflamatórias
(KUCHANA; RENUKA, 2018), entre outras. Nesse sentido, o planejamento in
sílico de derivados p-clorocinâmicos funcionalizado com o grupo
amida, formando assim a classe de compostos supracitada, pode indicar
informações cruciais para o desenvolvimento de novos compostos mais eficientes
para aplicação larvicida.
O estudo in sílico tem se difundido por toda a comunidade científica, em
que pesquisas acerca de novos agentes larvicida têm empregado essa estratégia
para acelerar a identificação e otimização de novos compostos (SILVA et al.
, 2019; KIOSHIMA et al., 2019). Alguns dos benefícios dos métodos
in silico estão relacionados à identificação computacional de potenciais
alvos biológicos, avaliação de propriedades farmacocinéticas, otimização do
tempo e custos em comparação as abordagens experimentais convencionais (SEGALL;
BARBER, 2014; BROGI et al., 2020).
Nesse sentido, o presente trabalho consiste em um planejamento racional de
cinamamidas como possíveis agentes larvicidas frente às larvas do Ae.
Aegypti. A estratégia computacional de múltiplos alvos biológicos será
utilizada para design e seleção de potenciais larvicidas.
Material e métodos
Inicialmente foi realizada uma análise voltada para a identificação de alvos
moleculares potenciais baseados na estrutura das moléculas por meio do servidor
da web “Similarity ensemble approach (SEA)” (https://sea.bkslab.org/), o qual se
baseia no princípio de similaridade química (KEISER et al., 2007;
SALVATORE et al., 2021). Posteriormente, foi realizada a homologia
molecular, validação dos alvos e posterior análise de docking molecular
visando analisar e discutir os potenciais alvos biológicos. Em seguida,
realizou-se a pesquisa das sequências de aminoácidos das enzimas hidrolase
epóxida (Q8MMJ6), proteína de ligação ao hormônio juvenil do mosquito (5V13),
acetilcolinesterase (Q6A2E2), glutationa S-transferase (5FT3), arilalquilamina
N-Acetiltransferase 7 (4FD7) 3-hidroxiquinurenina transaminase (6MFB) e
quitinase (O17411) por meio do banco de dados Uniprot
(https://www.uniprot.org/). Para a homologia molecular, essa foi realizada com a
utilização de modelos adequados presentes no Protein Data Bank (PDB). As
sequências FASTA (sequência de aminoácidos) foram submetidas ao modelo de
construção do SWISS-MODEL (https://swissmodel.expasy.org/). A validação das
estruturas tridimensionais das homologias foi realizada a partir da ferramenta
PDBsum por meio do gráfico de Ramachandran (FRANÇA et al., 2021). Em
seguida, foi realizado o docking molecular, em que, inicialmente as moléculas
projetadas foram desenhadas no Software Discovery Studio, otimizadas utilizando
o programa Avogadro e salvas no formato. mol2. Os estudos de docking molecular
foram realizados no programa GOLD 3.0 em computador PC Windows 11. As proteínas
foram previamente tratadas com a adição dos hidrogênios polares e a remoção de
todas as moléculas de água (MATEEV et al., 2022). Em seguida, foi
realizada a seleção da região catalítica num raio de busca de 6 Å. E o processo
de validação (Resolução e redocking molecular) para as proteínas co-
cristalizadas foi executado. Logo após, três funções de pontuação (ChemPLP,
GOLDScore e ChemScore) foram efetuadas e sua seleção foi feita a partir do
processo de redocking e do cálculo do Root Mean Square Deviation (RMSD),
respectivamente (BELL; ZHANG et al., 2019). A análise do sítio de ligação
e das ligações químicas formadas entre proteínas e ligantes foi realizada usando
o software Biovia Discovery Studio 2019 e o pymol versão 2.3.1 foi utilizado
para criar as ilustrações. Por fim, as propriedades físico-químicas das
moléculas planejadas foram determinadas no website Insecticide Physicochemical-
Properties Analysis Database (InsectiPAD).
Resultado e discussão
Estudos larvicidas têm demonstrado que a presença de grupos eletroretiradores,
com destaque aos átomos de halogênios, têm potencializado essa atividade
biológica devido ao aumento da polaridade e da lipofilicidade molecular
(HERNANDES et al., 2010). As larvas dos vetores com destaque ao Ae.
Aegypti possuem barreiras estruturais hidrofóbicas, com isso as propriedades
intensificadas pelos substituintes eletroretiradores exercem efeitos de suma
relevância para aumentar a atividade inseticida (FUJIWARA et al., 2017).
Diante disso, os derivados cinamamidas avaliados neste trabalho (Figura
1) foram prospectados com diferentes grupos eletroretiradores em diferentes
posições a fim de avaliar os efeitos desses na atividade em questão.
A partir dos resultados in silico Target Fishing, o pool dos
possíveis alvos moleculares da quimioteca de cinamamidas demonstrou a existência
de 3 supostos alvos enzimáticos (Hidrolase époxida - Q8MMJ6; arilalquilamina N-
Acetiltransferase 7 - 4FD7; quitinase - O17411) (Tabela 1). Ademais, na
literatura é amplamente discutido outros potenciais alvos biológicos do Ae.
Aegypti no desenvolvimento racional de inseticidas, também considerados
neste trabalho, quais sejam: Acetilcolinesterase (Q6A2E2), Glutationa S-
transferase (5FT3), 3-hidroxiquinurenina transaminase (4FD7), e a proteína do
hormônio juvenil do mosquito (5V13) (ARAÚJO et al., 2021). Sendo assim,
essas 7 proteínas foram alvos desse estudo das quais a 4FD7, 5FT3, 4FD7 e 5V13
possuem suas estruturas cristalográficas descritas no PDB, sendo essas
utilizadas na avaliação in silico. A Q8MMJ6, a O17411 e a Q6A2E2, por sua
vez, não se encontram descritas nos bancos de dados, sendo utilizada suas
respectivas homologias.
Assim, as biomoléculas Q8MMJ6, O17411 e a Q6A2E2 do vetor Ae. Aegypti
foram geradas por meio da homologia molecular, sendo construídas por comparação
da similaridade de sequências de aminoácidos com as de proteínas homólogas de
estruturas empiricamente determinadas (Q8MMJ6, O17411 e Q6A2E2). A identidade
sequencial das enzimas moldes para a realização da homologia da Q8MMJ6 e O17411
foi acima de 40% (Tabela 1), sendo condizente com os critérios
estabelecidos na literatura (>25%) (BRAY et al., 2018). Para tanto a
Q6A2E2 teve um maior percentual de identidade sequencial (>90%, Tabela
1), tendo sua validação já descrita por Franca et al. (2021).
Em seguida, foi realizado o processo de validação da homologia para a Q8MMJ6 e a
O17411 pelo gráfico Ramachandraw, para assim verificar os ângulos torcionais da
cadeia principal (φ e Ψ), ângulos torcionais das cadeias laterais (Chi) e
impedimentos estéricos (Figura 1B). Desse modo, foi possível inferir que
ambas as estruturas apresentaram mais de 90% dos resíduos em regiões permitidas,
o que configura a acurácia dos modelos gerados para posterior etapa de
realização de docking molecular.
Para as proteínas co-cristalizadas os métodos de validações foram a resolução da
macromolécula (Tabela 1), condizente aos resultados de biomoléculas com
conformações adequadas para o estudo in sílico (< 3,0 Å) (CHEUNG et
al., 2018; LI et al., 2014), e o processo de Redocking
molecular, a fim de verificar a acurácia do programa GOLD em reproduzir a
conformação bioativa do ligante cristalográfico (ligante co-cristalizado).
Assim, pôde-se notar que o input gerado pelo software conseguiu obter
conformações semelhantes aos ligantes co-cristalizados, obtendo RMSDs abaixo de
1, estando dentro da margem preconizada pela literatura para essa validação
(RMSD < 2) (BELL & ZHANG, 2019).
A seleção da melhor função de pontuação para as macromoléculas 4FD7 (RMSD = 0,36
e Função de pontuação: Chemplp), 5FT3 (RMSD = 0,87, Função de pontuação:
Chemscore), 6 MFB (RMSD = 0,85, Função de pontuação: Chemplp) e 5V13 (RMSD =
0,48, Função de pontuação: Chemplp), ocorreu a partir do menor valor de RMSD
para cada função de pontuação analisada. Ao passo que para as homologias a
escolha das funções de pontuações ocorreu pelo maior FitScore (Energia de
correlação) do complexo ligante/receptor (Figura 2). Em seguida a triagem
virtual das p-cloro - cinamamidas –n-substituídas foi realizada
para as sete macromoléculas, conforme a Tabela 1.
Os resultados de FitScore das p-cloro - cinamamidas n-
substituídas complexadas às macromoléculas demonstraram uma maior eficiência ou
semelhança ao p-clorocinamato de etila (Composto de referência, Figura
1; (FitScore (O17411) = 20,36; (FitScore (Q8MMJ6) = 40,32;
FitScore (5V13) = 65,67; (FitScore (Q6A2E2) = 45,82 e
(FitScore (6MFB) = 44,47) em interagir com as macromoléculas. A análise
dos complexos cinnamamidas-macromoléculas mostrou uma faixa de energia de
correlação maior para as proteínas O17411 (FitScore = 24,14 – 27,45),
Q8MMJ6 (FitScore = 41,82 – 50,75), 5V13 (FitScore= 72,41 – 88,82),
Q6A2E2 (FitScore = 48,13 – 59,65); 4FD7(FitScore = 17,77) e 6MFB
(FitScore= 52,70 – 65,22), respectivamente. Ao passo que para a
macromolécula 5FT13 (FitScore= 27,04 – 32,89) essa classe de moléculas
não teve um efeito tão pronunciado como o p-clorocinamato de etila
(FitScore = 45,08). Com isso, pode-se inferior que essa série cinâmica
pode vir a se tornar uma boa candidata como larvicida frente ao vetor Ae.
Aegypti.
Além disso, foi possível observar que o ligante (E)-3-(4-clorofenil)-N-
(4-fluorofenetil) acrilamida (10) se destacou frente a três alvos
biológicos, a 5V13, 6MFB, e a Q6A2E2, com FitScores de energias 88,82,
65,22 e 59,65, respectivamente (Figura 2A-B), demonstrando-se superior a
substância padrão, o que pode estar associado à presença da função amida e do
substituinte fenila com o flúor na cadeia lateral do esqueleto cinâmico. Já para
as proteínas 5FT3, O17411, 4FD7 e Q8MMJ6 as moléculas 2, 4, 9 e 12
tiveram as maiores energias de correlação, nessa ordem (Figura 2A-2B).
No que se refere às interações entre as cinamamidas projetadas com resíduos de
aminoácidos dos alvos biológicos avaliados, pôde-se notar uma prevalência de
interações do tipo: Van der Waals, ligações de halogênio, interações Pi-Pi
Stack, ligações de hidrogênio entre outras. Além do mais, como demonstrado no
cluster na Figura 1C para a molécula 10, todos os compostos
tenderam a interagir principalmente com resíduos de aminoácidos da cavidade do
sítio catalítico enzimático de cada proteína.
Em consonância aos resultados de docking molecular, as propriedades físico-
químicas in sílico dos compostos avaliados (Figura 2C) estão
dentro das normas que preveem o desenvolvimento bio-racional de um inseticida
seletivo e ambientalmente seguro (HAO et al., 2011), em que para todos os
compostos foi possível obter MM < 435 Da, ClogP < 6, nHBA < 6, nHBD ≤ 2, nROT <
9 e nARB < 17. Dentre esses parâmetros, destacam-se principalmente a nROT e
CLogP, as quais relacionam à fotoestabilidade e à permeabilidade na cutícula
lipofílica do inseto, respectivamente, nas quais, as cinamamidas em questão
apresentaram valores iguais ou semelhantes a derivados do ácido cinâmico
contendo o grupo hidrazina (NARESH et al., 2022).
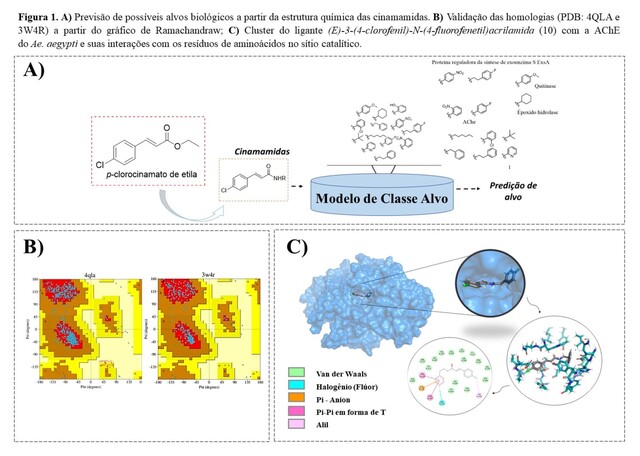
A) Previsão dos possíveis alvos biológicos a partir da estrutura química. B) Gráfico de Ramanchandraw; C) Interações do composto 10 com a AChE.
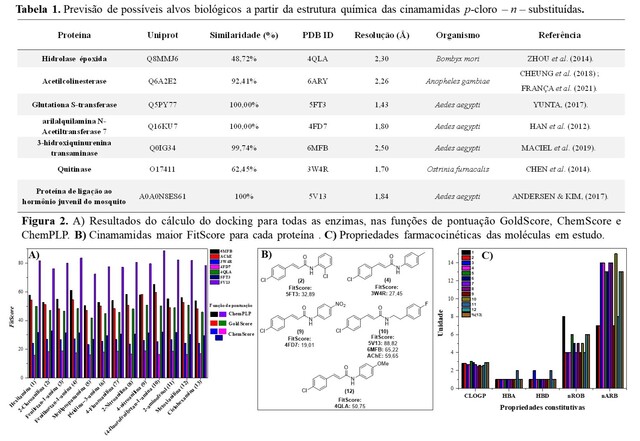
Tabela 1. Possíveis alvos; Figura 2. A) Docking para todas as enzimas; B) Maior FitScore para cada proteína; C) Propriedades farmacocinéticas.
Conclusões
Em suma, no trabalho foram projetadas 13 p-cloro - cinamamidas - n- substituídas,
selecionado 7 alvos biológicos presentes no Ae. aegypti e realizado estudos in
sílico para determinar a eficiência dessas moléculas como possíveis inseticidas.
Para a seleção dos alvos biológicos, utilizou-se a ferramenta Target Fishing e a
literatura, em que para realizar os estudos de docking molecular foram utilizadas
as 5FT3, 4FD7, 6MFB e 5V13 co-cristalizadas presentes no PDB, enquanto as
macromoléculas Q6A2E2, Q8MMJ6 e O17411 foram construídas via homologia estrutural
de alvos biológicos macromoleculares com similaridade entre 48,72% - 92,41% com o
Ae aegypti. Dos compostos avaliados, destaca-se a molécula 10, que apresentou uma
eficiência superior ao p-cloro-cinamato de etila frente a 3 dos 7 alvos biológicos
avaliados. Além disso, os compostos apresentaram propriedades constitutivas dentro
das normas que preveem a boa eficiência de um possível inseticida. Com isso, os
resultados em questão sugerem bons candidatos à inseticida, embora estudos
experimentais precisem ser realizados para comprovar a acuracidade dos estudos in
sílico.
Agradecimentos
Universidade Federal de Alagoas (UFAL); Instituto de Química e Biotecnologia,
LMC,CNPq, CAPES e FAPEAL.
Referências
ARAÚJO, M. O.; PÉREZ-CASTILLO, Y.; OLIVEIRA, L. H.; NUNES, F. C.; SOUSA, D. P. D.. Larvicidal Activity of Cinnamic Acid Derivatives: Investigating Alternative Products for Aedes aegypti L. Control. Molecules, v. 26, nº 1, 10.3390, 2021. Doi: https://doi.org/10.3390/molecules26010061
BALWAN, W. K.; SINGH, A.; KOUR, S.. Dengue-The Breakbone Fever: A Crisis within a Crisis. 2022. Doi: DOI: 10.36349/easms.2022.v05i04.006
BARRETO, C. F. Aedes aegypti - Resistência aos inseticidas químicos e as novas alternativas de controle. Revista Eletrônica Faculdade Montes Belos, Goiás, v.1, n.2, p.62-73, nov. 2005.
BELL, E. W.; ZHANG, Y. DockRMSD: An open-source tool for atom mapping and RMSD calculation of symmetric molecules through graph isomorphism. Journal of Cheminformatics, v. 11, n. 1, p. 1–9, 2019. . Doi: https://doi.org/10.1186/s13321-019-0362-7
BRAY, F.; FERLAY, J.; SOERJOMATARAM, I.; SIEGEL, R. L.; TORRE, L. A.; JEMAL, A.. Global cancer statistics: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: a cancer journal for clinicians, 68(6), 394-424. 2018.
BROGI, S.; RAMALHO, T. C.; KUCA, K.; MEDINA-FRANCO, J. L.; VALKO, M.. In silico methods for drug design and discovery. Frontiers in chemistry, 8, 612. 2020.
CHEN, L.; LIU, T.; ZHOU, Y.; CHEN, Q.; SHEN, X.; YANG, Q.. Structural characteristics of an insect group I chitinase, an enzyme indispensable to moulting. Acta Crystallographica Section D: Biological Crystallography, 70(4), 932-942. 2014.
CHEUNG, J. et al. Structure of the G119S mutant acetylcholinesterase of the malaria vector Anopheles gambiae reveals basis of insecticide resistance. Structure, v. 26, n. 1, p. 130-136. e2, 2018. Doi: https://doi.org/10.1016/j.str.2017.11.021
DAI, L.; ZANG, C.; TIAN, S.; LIU, W.; TAN, S.; CAI, Z.; JIANG, Y.. Design, synthesis, and evaluation of caffeic acid amides as synergists to sensitize fluconazole-resistant Candida albicans to fluconazole. Bioorganic & Medicinal Chemistry Letters, 25(1), 34-37. 2015.
DI SALVATORE, V.; MALEKI, A.; RUSSO, G.; SGROI, G.; PALUMBO, G. A. P.; PAPPALARDO, F. A multi-step and multi-scale bioinformatic approach to investigate potential source of cross-reactive immunity against SARS-CoV-2 UK variant. In: 2021 IEEE International Conference on Bioinformatics and Biomedicine (BIBM). IEEE, 2021. p. 3303-3307.
FRANÇA, S. B.; DE LIMA, L. C. B.; DA SILVA CUNHA, C. R.; ANUNCIAÇÃO, D. S.; DA SILVA-JÚNIOR, E. F.; BARROS, M. E. D. S. B.; DA PAZ LIMA, D. J.. Larvicidal activity and in silico studies of cinnamic acid derivatives against Aedes aegypti (Diptera: Culicidae). Bioorganic & Medicinal Chemistry, 44, 2021.
FUJIWARA, G. M.; ANNIES, V.; DE OLIVEIRA, C. F.; LARA, R. A.; GABRIEL, M. M.; BETIM, F. C.; ZANIN, S. M.. Evaluation of larvicidal activity and ecotoxicity of linalool, methyl cinnamate and methyl cinnamate/linalool in combination against Aedes aegypti. Ecotoxicology and Environmental Safety, v. 139, p. 238-244, 2017. Doi: https://doi.org/10.1016/j.ecoenv.2017.01.046.
HAN, Q.; FANG, J.; LI, J.. 3-Hydroxykynurenine transaminase identity with alanine glyoxylate transaminase: a probable detoxification protein in Aedes aegypti. Journal of Biological Chemistry, v. 277, n. 18, p. 15781-15787, 2002. Doi: https://doi.org/10.1074/jbc.M201202200
HAN, Q.; ROBINSON, H.; DING, H.; CHRISTENSEN, B. M.; LI, J.. Evolution of insect arylalkylamine N-acetyltransferases: structural evidence from the yellow fever mosquito, Aedes aegypti. Proceedings of the National Academy of Sciences, v. 109, n. 29, p. 11669-11674, 2012.
HAO, G.; DONG, Q.; YANG, G.. A comparative study on the constitutive properties of marketed pesticides. Molecular informatics, v. 30, n. 6‐7, p. 614-622, 2011.
HERNANDES, M. Z.; CAVALCANTI, S. M. T.; MOREIRA, D. R. M.; DE AZEVEDO JUNIOR, W. F.; LEITE, A. C. L. Halogen Atoms in the Modern Medicinal Chemistry: Hints for the Drug Design. Current Drug Targets, v. 11, n° 3, p. 303 – 314, 2010. Doi: https://doi.org/10.2174/138945010790711996.
JUAREZ, J. G.; GARCIA-LUNA, S. M.; MEDEIROS, M. C.; DICKINSON, K. L.; BORUCKI, M. K.; FRANK, M.; HAMER, G. L.. The eco-bio-social factors that modulate Aedes aegypti abundance in South Texas Border Communities. Insects, 12(2), 183. 2021.
KEISER, M. J.; ROTH, B. L.; ARMBRUSTER, B. N.; ERNSBERGER, P.; IRWIN, J. J.; SHOICHET, B. K.. Relating protein pharmacology by ligand chemistry. Nature biotechnology, 25(2), 197-206. 2007.
KIM, I. H.; PHAM, V.; JABLONKA, W.; GOODMAN, W. G.; RIBEIRO, J. M.; ANDERSEN, J. F.. A mosquito hemolymph odorant-binding protein family member specifically binds juvenile hormone. Journal of Biological Chemistry, v. 292, n. 37, p. 15329-15339, 2017.
KIOSHIMA, E. S.; SHINOBU-MESQUITA, C. S.; ABADIO, A. K. R.; FELIPE, M. S. S.; SVIDZINSKI, T. I. E.; MAIGRET, B.. Selection of potential anti-adhesion drugs by in silico approaches targeted to ALS3 from Candida albicans. Biotechnology letters, 41(12), 1391-1401. 2019.
KUCHANA, M.; KUMMARI, R.. Synthesis and evaluation of novel α-cyano-N-(2-hydroxyphenyl) cinnamamides for antioxidant, antibacterial and anti-inflammatory activities: in silico prediction of drug likeness properties. Int J Pharm Res, v. 10, n. 3, p. 300-10, 2018.
LI, J.; LI, G.. Transamination of 3-hydroxykynurenine to produce xanthurenic acid: a major branch pathway of tryptophan metabolism in the mosquito, Aedes aegypti, during larval development. Insect biochemistry and molecular biology, v. 27, n. 10, p. 859-867, 1997.
MACIEL, L. G.; OLIVEIRA, A. A.; ROMÃO, T. P.; LEAL, L. L.; GUIDO, R. V.; SILVA-FILHA, M. H. N.; SOARES, T. A.. Evidence for the role of 3-hydroxykynurenine transaminase from Aedes aegypti as a molecular target for selective inhibitors. European Journal of Medicinal Chemistry, 2019
MATEEV, E.; VALKOVA, I.; ANGELOV, B.; GEORGIEVA, M.; ZLATKOV, A. Validation Through Re-Docking, Cross-Docking And Ligand Enrichment In Various Well-Resoluted Mao-B Receptors. Int J Pharm Sci Res, v. 13, p. 1000-8, 2022.
MOREIRA, M. F.; MANSUR, J. F.. Resistência e inseticidas: estratégias, desafios e perspectivas no controle de insetos. Instituto Nacional de Ciência e Tecnologia em Entomologia Molecular, cap. 15, p. 1-23, 2012.
NARESH, P.; KUMAR, R. R.; VISHWAS, H. N.; RAJAGOPAL, G.; PRABHA, T.; JUBIE, S.. Larvicidal and histopathological efficacy of cinnamic acid analogues: a novel strategy to reduce the dengue vector competence. RSC advances, v. 12, n. 16, p. 9793-9814, 2022.
NATAL, D.. Bioecologia do Aedes aegypti. Biológico, v. 64, n. 2, p. 205-207, 2002.
PENG, W.; FU, X.; XIONG, Z.; XIANG, M.; YANG, Y.; WU, H.; LI, B.. Chemical components from the stems and leaves of Clausena lansium (Lour.) Skeels and their potential herbicidal effects. Pest Management Science, v. 77, n. 3, p. 1355-1360, 2021.
PEPERIDOU, A.; PONTIKI, E.; HADJIPAVLOU-LITINA, D.; VOULGARI, E.; AVGOUSTAKIS, K. Multifunctional Cinnamic Acid Derivatives. Molecules, v. 22, n° 1247, p. 1-17, 2017.
SEGALL, M. D.; BARBER, C.. Addressing toxicity risk when designing and selecting compounds in early drug discovery. Drug discovery today, v. 19, n. 5, p. 688-693, 2014.
SHARMA, P. Cinnamic acid derivatives: a new chapter of various pharmacological activities. Journal of Chemical and Pharmaceutical Research, v.3, n°2, p. 403-423, 2011.
VEBER, D. F.; JOHNSON, S. R.; CHENG, H. Y.; SMITH, B. R.; WARD, K. W.; KOPPLE, K. D.. Molecular properties that influence the oral bioavailability of drug candidates. Journal of medicinal chemistry, v. 45, n. 12, p. 2615-2623, 2002.
YANG, K. et al. Synthesis, mitochondrial localization of fluorescent derivatives of cinnamamide as anticancer agents. European Journal of Medicinal Chemistry, v. 170, p. 45-54, 2019.
YUNTA, C., ISMAIL, H.M., PAINE, M.J.I.. A simple test for the determination of Ddt on sprayed surfaces. Doi: 10.2210/pdb5FT3/pdb
ZARA, A. L. D. S. A.; SANTOS, S. M. D.; FERNANDES-OLIVEIRA, E. S.; CARVALHO, R. G.; COELHO, G. E.. Estratégias de controle do Aedes aegypti: uma revisão. Epidemiologia e Serviços de Saúde, 25, 391-404. 2016.
ZHOU, K.; JIA, N.; HU, C.; JIANG, Y. L.; YANG, J. P.; CHEN, Y.; ZHOU, C. Z.. Crystal structure of juvenile hormone epoxide hydrolase from the silkworm B ombyx mori. Proteins: Structure, Function, and Bioinformatics, v. 82, n. 11, p. 3224-3229, 2014.
















